+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human ACKR3 in complex with CXCL12, a small molecule partial agonist CCX662, and an extracellular Fab | ||||||||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | Atypical Chemokine Receptor / MEMBRANE PROTEIN / SIGNALING PROTEIN / SIGNALING PROTEIN-IMMUNE SYSTEM complex | ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報oculomotor nerve development / positive regulation of mesenchymal stem cell migration / telencephalon cell migration / chemokine (C-X-C motif) ligand 12 signaling pathway / negative regulation of leukocyte tethering or rolling / response to ultrasound / regulation of actin polymerization or depolymerization / chemokine receptor binding / C-X-C chemokine binding / CXCL12-activated CXCR4 signaling pathway ...oculomotor nerve development / positive regulation of mesenchymal stem cell migration / telencephalon cell migration / chemokine (C-X-C motif) ligand 12 signaling pathway / negative regulation of leukocyte tethering or rolling / response to ultrasound / regulation of actin polymerization or depolymerization / chemokine receptor binding / C-X-C chemokine binding / CXCL12-activated CXCR4 signaling pathway / CXCR chemokine receptor binding / C-X-C chemokine receptor activity / positive regulation of axon extension involved in axon guidance / positive regulation of vasculature development / positive regulation of dopamine secretion / Signaling by ROBO receptors / induction of positive chemotaxis / integrin activation / negative regulation of dendritic cell apoptotic process / C-C chemokine receptor activity / negative regulation of intrinsic apoptotic signaling pathway in response to DNA damage / cellular response to chemokine / chemokine-mediated signaling pathway / C-C chemokine binding / positive regulation of monocyte chemotaxis / chemokine activity / blood circulation / Chemokine receptors bind chemokines / scavenger receptor activity / positive regulation of calcium ion import / detection of temperature stimulus involved in sensory perception of pain / animal organ regeneration / detection of mechanical stimulus involved in sensory perception of pain / positive regulation of T cell migration / vasculogenesis / Nuclear signaling by ERBB4 / coreceptor activity / clathrin-coated pit / positive regulation of endothelial cell proliferation / positive regulation of neuron differentiation / positive regulation of cell adhesion / axon guidance / adult locomotory behavior / cell chemotaxis / growth factor activity / calcium-mediated signaling / defense response / response to peptide hormone / recycling endosome / receptor internalization / response to virus / integrin binding / neuron migration / intracellular calcium ion homeostasis / chemotaxis / : / positive regulation of cytosolic calcium ion concentration / angiogenesis / G alpha (i) signalling events / Estrogen-dependent gene expression / early endosome / response to hypoxia / positive regulation of ERK1 and ERK2 cascade / cell adhesion / endosome / immune response / positive regulation of cell migration / G protein-coupled receptor signaling pathway / signaling receptor binding / negative regulation of cell population proliferation / external side of plasma membrane / intracellular membrane-bounded organelle / cell surface / signal transduction / extracellular exosome / extracellular region / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | ||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Yen YC / Schafer CT / Gustavsson M / Handel TM / Tesmer JJG | ||||||||||||||||||||||||
| 資金援助 |  米国, 7件 米国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Structures of atypical chemokine receptor 3 reveal the basis for its promiscuity and signaling bias. 著者: Yu-Chen Yen / Christopher T Schafer / Martin Gustavsson / Stefanie A Eberle / Pawel K Dominik / Dawid Deneka / Penglie Zhang / Thomas J Schall / Anthony A Kossiakoff / John J G Tesmer / Tracy M Handel /    要旨: Both CXC chemokine receptor 4 (CXCR4) and atypical chemokine receptor 3 (ACKR3) are activated by the chemokine CXCL12 yet evoke distinct cellular responses. CXCR4 is a canonical G protein-coupled ...Both CXC chemokine receptor 4 (CXCR4) and atypical chemokine receptor 3 (ACKR3) are activated by the chemokine CXCL12 yet evoke distinct cellular responses. CXCR4 is a canonical G protein-coupled receptor (GPCR), whereas ACKR3 is intrinsically biased for arrestin. The molecular basis for this difference is not understood. Here, we describe cryo-EM structures of ACKR3 in complex with CXCL12, a more potent CXCL12 variant, and a small-molecule agonist. The bound chemokines adopt an unexpected pose relative to those established for CXCR4 and observed in other receptor-chemokine complexes. Along with functional studies, these structures provide insight into the ligand-binding promiscuity of ACKR3, why it fails to couple to G proteins, and its bias toward β-arrestin. The results lay the groundwork for understanding the physiological interplay of ACKR3 with other GPCRs. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25175.map.gz emd_25175.map.gz | 97.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25175-v30.xml emd-25175-v30.xml emd-25175.xml emd-25175.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25175_fsc.xml emd_25175_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25175.png emd_25175.png | 87.1 KB | ||
| Filedesc metadata |  emd-25175.cif.gz emd-25175.cif.gz | 6.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25175 http://ftp.pdbj.org/pub/emdb/structures/EMD-25175 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25175 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25175 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25175_validation.pdf.gz emd_25175_validation.pdf.gz | 495.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25175_full_validation.pdf.gz emd_25175_full_validation.pdf.gz | 494.8 KB | 表示 | |
| XML形式データ |  emd_25175_validation.xml.gz emd_25175_validation.xml.gz | 11.7 KB | 表示 | |
| CIF形式データ |  emd_25175_validation.cif.gz emd_25175_validation.cif.gz | 15.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25175 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25175 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25175 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25175 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7sk7MC  7sk3C  7sk4C  7sk5C  7sk6C  7sk8C  7sk9C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25175.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25175.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex structure of NB-CID25-ACKR3-CXCL12-CCX662
| 全体 | 名称: Complex structure of NB-CID25-ACKR3-CXCL12-CCX662 |
|---|---|
| 要素 |
|
-超分子 #1: Complex structure of NB-CID25-ACKR3-CXCL12-CCX662
| 超分子 | 名称: Complex structure of NB-CID25-ACKR3-CXCL12-CCX662 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|
-分子 #1: Atypical chemokine receptor 3
| 分子 | 名称: Atypical chemokine receptor 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 45.196406 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GAPDLHLFDY SEPGNFSDIS WPCNSSDCIV VDTVMCPNMP NKSVLLYTLS FIYIFIFVIG MIANSVVVWV NIQAKTTGYD THCYILNLA IADLWVVLTI PVWVVSLVQH NQWPMGELTC KVTHLIFSIN LFGSIFFLTC MSVDRYLSIT YFTNTPSSRK K MVRRVVCI ...文字列: GAPDLHLFDY SEPGNFSDIS WPCNSSDCIV VDTVMCPNMP NKSVLLYTLS FIYIFIFVIG MIANSVVVWV NIQAKTTGYD THCYILNLA IADLWVVLTI PVWVVSLVQH NQWPMGELTC KVTHLIFSIN LFGSIFFLTC MSVDRYLSIT YFTNTPSSRK K MVRRVVCI LVWLLAFCVS LPDTYYLKTV TSASNNETYC RSFYPEHSIK EWLIGMELVS VVLGFAVPFS IIAVFYFLLA RA ISASSDQ EKHSSRKIIF SYVVVFLVCW LPYHVAVLLD IFSILHYIPF TCRLEHALFT ALHVTQCLSL VHCCVNPVLY SFI NRNYRY ELMKAFIFKY SAKTGLTKLI DASRVSETEY SALEQSTKGR PLEVLFQGPH HHHHHHHHHD YKDDDDK UniProtKB: Atypical chemokine receptor 3 |
-分子 #2: Stromal cell-derived factor 1
| 分子 | 名称: Stromal cell-derived factor 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 7.97846 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KPVSLSYRCP CRFFESHVAR ANVKHLKILN TPNCALQIVA RLKNNNRQVC IDPKLKWIQE YLEKALNK UniProtKB: Stromal cell-derived factor 1 |
-分子 #3: CID25 Fab light chain
| 分子 | 名称: CID25 Fab light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.531129 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQYYYPLFTF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQYYYPLFTF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #4: CID25 Fab heavy chain
| 分子 | 名称: CID25 Fab heavy chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.461383 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NFSYSSIHWV RQAPGKGLEW VAYIYSSYGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARVYPWWYYK YYHGALDYWG QGTLVTVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD Y FPEPVTVS ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NFSYSSIHWV RQAPGKGLEW VAYIYSSYGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARVYPWWYYK YYHGALDYWG QGTLVTVSSA STKGPSVFPL APSSKSTSGG TAALGCLVKD Y FPEPVTVS WNSGALTSGV HTFPAVLQSS GLYSLSSVVT VPSSSLGTQT YICNVNHKPS NTKVDKKVEP KSCDKTHT |
-分子 #5: Anti-Fab nanobody
| 分子 | 名称: Anti-Fab nanobody / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.390644 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSQVQLQESG GGLVQPGGSL RLSCAASGRT ISRYAMSWFR QAPGKEREFV AVARRSGDGA FYADSVQGRF TVSRDDAKNT VYLQMNSLK PEDTAVYYCA IDSDTFYSGS YDYWGQGTQV TVSS |
-分子 #6: (1R)-4-[7-(3-carboxypropoxy)-6-methylquinolin-8-yl]-1-{[2-(4-hydr...
| 分子 | 名称: (1R)-4-[7-(3-carboxypropoxy)-6-methylquinolin-8-yl]-1-{[2-(4-hydroxypiperidin-1-yl)-1,3-thiazol-4-yl]methyl}-1,4-diazepan-1-ium タイプ: ligand / ID: 6 / コピー数: 1 / 式: GJ9 |
|---|---|
| 分子量 | 理論値: 540.697 Da |
| Chemical component information | 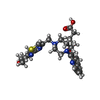 ChemComp-GJ9: |
-分子 #7: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 7 / コピー数: 1 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 53.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)