+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Lassa virus glycoprotein construct(Josiah GPCysR4) recovered from GPC-I53-50 nanoparticle by localized reconstruction | |||||||||
 マップデータ マップデータ | Lassa virus glycoprotein (GPCysR4) map reconstructed from GPCysR4-I53-50 nanoparticle by localized reconstruction - Main Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | glycoprotein / Lassa / Josiah / vaccine design / nanoparticle / protein design / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / receptor-mediated endocytosis of virus by host cell / host cell endoplasmic reticulum membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) | |||||||||
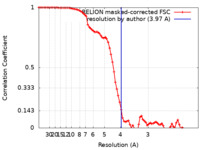
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.97 Å | |||||||||
 データ登録者 データ登録者 | Antanasijevic A / Brouwer PJM | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2022 ジャーナル: Cell Host Microbe / 年: 2022タイトル: Lassa virus glycoprotein nanoparticles elicit neutralizing antibody responses and protection. 著者: Philip J M Brouwer / Aleksandar Antanasijevic / Adam J Ronk / Helena Müller-Kräuter / Yasunori Watanabe / Mathieu Claireaux / Hailee R Perrett / Tom P L Bijl / Marloes Grobben / Jeffrey C ...著者: Philip J M Brouwer / Aleksandar Antanasijevic / Adam J Ronk / Helena Müller-Kräuter / Yasunori Watanabe / Mathieu Claireaux / Hailee R Perrett / Tom P L Bijl / Marloes Grobben / Jeffrey C Umotoy / Angela I Schriek / Judith A Burger / Khadija Tejjani / Nicole M Lloyd / Thijs H Steijaert / Marlies M van Haaren / Kwinten Sliepen / Steven W de Taeye / Marit J van Gils / Max Crispin / Thomas Strecker / Alexander Bukreyev / Andrew B Ward / Rogier W Sanders /     要旨: The Lassa virus is endemic in parts of West Africa, and it causes hemorrhagic fever with high mortality. The development of a recombinant protein vaccine has been hampered by the instability of ...The Lassa virus is endemic in parts of West Africa, and it causes hemorrhagic fever with high mortality. The development of a recombinant protein vaccine has been hampered by the instability of soluble Lassa virus glycoprotein complex (GPC) trimers, which disassemble into monomeric subunits after expression. Here, we use two-component protein nanoparticles consisting of trimeric and pentameric subunits to stabilize GPC in a trimeric conformation. These GPC nanoparticles present twenty prefusion GPC trimers on the surface of an icosahedral particle. Cryo-EM studies of GPC nanoparticles demonstrated a well-ordered structure and yielded a high-resolution structure of an unliganded GPC. These nanoparticles induced potent humoral immune responses in rabbits and protective immunity against the lethal Lassa virus challenge in guinea pigs. Additionally, we isolated a neutralizing antibody that mapped to the putative receptor-binding site, revealing a previously undefined site of vulnerability. Collectively, these findings offer potential approaches to vaccine and therapeutic design for the Lassa virus. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25107.map.gz emd_25107.map.gz | 37.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25107-v30.xml emd-25107-v30.xml emd-25107.xml emd-25107.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25107_fsc.xml emd_25107_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25107.png emd_25107.png | 109.7 KB | ||
| マスクデータ |  emd_25107_msk_1.map emd_25107_msk_1.map | 40.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-25107.cif.gz emd-25107.cif.gz | 7.2 KB | ||
| その他 |  emd_25107_half_map_1.map.gz emd_25107_half_map_1.map.gz emd_25107_half_map_2.map.gz emd_25107_half_map_2.map.gz | 30.9 MB 30.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25107 http://ftp.pdbj.org/pub/emdb/structures/EMD-25107 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25107 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25107 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25107_validation.pdf.gz emd_25107_validation.pdf.gz | 749.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25107_full_validation.pdf.gz emd_25107_full_validation.pdf.gz | 748.9 KB | 表示 | |
| XML形式データ |  emd_25107_validation.xml.gz emd_25107_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_25107_validation.cif.gz emd_25107_validation.cif.gz | 19.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25107 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25107 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25107 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25107 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25107.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25107.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Lassa virus glycoprotein (GPCysR4) map reconstructed from GPCysR4-I53-50 nanoparticle by localized reconstruction - Main Map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_25107_msk_1.map emd_25107_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Lassa virus glycoprotein (GPCysR4) map reconstructed from GPCysR4-I53-50...
| ファイル | emd_25107_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Lassa virus glycoprotein (GPCysR4) map reconstructed from GPCysR4-I53-50 nanoparticle by localized reconstruction - Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Lassa virus glycoprotein (GPCysR4) map reconstructed from GPCysR4-I53-50...
| ファイル | emd_25107_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Lassa virus glycoprotein (GPCysR4) map reconstructed from GPCysR4-I53-50 nanoparticle by localized reconstruction - Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Lassa virus glycoprotein construct(Josiah GPCysR4) recovered from...
| 全体 | 名称: Lassa virus glycoprotein construct(Josiah GPCysR4) recovered from GPC-I53-50 nanoparticle |
|---|---|
| 要素 |
|
-超分子 #1: Lassa virus glycoprotein construct(Josiah GPCysR4) recovered from...
| 超分子 | 名称: Lassa virus glycoprotein construct(Josiah GPCysR4) recovered from GPC-I53-50 nanoparticle タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Josiah GPCysR4 is a trimeric complex consisting of 3 copies of the head domain and 3 copies of the stem domain. Nanoparticle was assembled by combining equimolar amounts of GPC-I53-50A and I53-50B. |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
-分子 #1: Josiah GPCysR4 I53-50A
| 分子 | 名称: Josiah GPCysR4 I53-50A / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
| 分子量 | 理論値: 73.84557 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGQIVTFFQE VPHVIEEVMN IVLIALSVLA VLKGLYNFAT CGLVGLVTFL LLCGRSCTTS LYKGVYELQT LELNMETLNM TMPLSCTKN NSHHYIMVGN ETGLELTLTN TSIINHKFCN LSDAHKKNLY DHALMSIIST FHLSIPNFNQ YEAMSCDFNG G KISVQYNL ...文字列: MGQIVTFFQE VPHVIEEVMN IVLIALSVLA VLKGLYNFAT CGLVGLVTFL LLCGRSCTTS LYKGVYELQT LELNMETLNM TMPLSCTKN NSHHYIMVGN ETGLELTLTN TSIINHKFCN LSDAHKKNLY DHALMSIIST FHLSIPNFNQ YEAMSCDFNG G KISVQYNL SHSYAGDAAN HCGTVANGVL QTFMRMAWGG SYIALDSGCG NWDCIMTSYQ YLIIQNTTWE DHCQFSRPSP IG YLGLLSQ RTRDIYISRR RRGTFTWTLS DSEGKDTPGG YCLTRWMLIE AELKCFGNTA VAKCNEKHDE EFCDMLRLFD FNK QAIQRL KAPAQMSIQL INKAVNALIN DQLIMKNHLR DIMCIPYCNY SKYWYLNHTT TGRTSLPKCW LVSNGSYLNE THFS DDIEQ QADNMITEML QKEYMERQGG SGGSGGSGGS GGSEKAAKAE EAARKMEELF KKHKIVAVLR ANSVEEAIEK AVAVF AGGV HLIEITFTVP DADTVIKALS VLKEKGAIIG AGTVTSVEQC RKAVESGAEF IVSPHLDEEI SQFCKEKGVF YMPGVM TPT ELVKAMKLGH DILKLFPGEV VGPEFVKAMK GPFPNVKFVP TGGVDLDNVC EWFDAGVLAV GVGDALVEGD PDEVREK AK EFVEKIRGCT EGSLEWSHPQ FEK UniProtKB: Pre-glycoprotein polyprotein GP complex |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
詳細: TBS | |||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 7 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot time 4s, Wait time 10s, Blot force 0. | |||||||||
| 詳細 | Lassa virus glycoprotein construct(Josiah GPCysR4) recovered from GPC-I53-50 nanoparticle by localized reconstruction. Nanoparticle was assembled by combining equimolar amounts of GPC-I53-50A and I53-50B. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 平均露光時間: 10.5 sec. / 平均電子線量: 50.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)