+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24930 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of dolphin Prestin: Inhibited I (Chloride + Salicylate) | |||||||||
 マップデータ マップデータ | High Cl Salicylate | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Outer hair cells / electromotility / mechanotransduction / hearing / deafness / frequency sensation / echolocation / SLC26 / SLC26A5 / MOTOR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cochlear outer hair cell electromotile response / secondary active sulfate transmembrane transporter activity / oxalate transmembrane transporter activity / bicarbonate transmembrane transporter activity / chloride transmembrane transporter activity / sensory perception of sound / regulation of cell shape / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Bavi N / Clark MD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: The conformational cycle of prestin underlies outer-hair cell electromotility. 著者: Navid Bavi / Michael David Clark / Gustavo F Contreras / Rong Shen / Bharat G Reddy / Wieslawa Milewski / Eduardo Perozo /  要旨: The voltage-dependent motor protein prestin (also known as SLC26A5) is responsible for the electromotive behaviour of outer-hair cells and underlies the cochlear amplifier. Knockout or impairment of ...The voltage-dependent motor protein prestin (also known as SLC26A5) is responsible for the electromotive behaviour of outer-hair cells and underlies the cochlear amplifier. Knockout or impairment of prestin causes severe hearing loss. Despite the key role of prestin in hearing, the mechanism by which mammalian prestin senses voltage and transduces it into cellular-scale movements (electromotility) is poorly understood. Here we determined the structure of dolphin prestin in six distinct states using single-particle cryo-electron microscopy. Our structural and functional data suggest that prestin adopts a unique and complex set of states, tunable by the identity of bound anions (Cl or SO). Salicylate, a drug that can cause reversible hearing loss, competes for the anion-binding site of prestin, and inhibits its function by immobilizing prestin in a new conformation. Our data suggest that the bound anion together with its coordinating charged residues and helical dipole act as a dynamic voltage sensor. An analysis of all of the anion-dependent conformations reveals how structural rearrangements in the voltage sensor are coupled to conformational transitions at the protein-membrane interface, suggesting a previously undescribed mechanism of area expansion. Visualization of the electromotility cycle of prestin distinguishes the protein from the closely related SLC26 anion transporters, highlighting the basis for evolutionary specialization of the mammalian cochlear amplifier at a high resolution. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24930.map.gz emd_24930.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24930-v30.xml emd-24930-v30.xml emd-24930.xml emd-24930.xml | 12.2 KB 12.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24930_fsc.xml emd_24930_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24930.png emd_24930.png | 97.4 KB | ||
| Filedesc metadata |  emd-24930.cif.gz emd-24930.cif.gz | 5.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24930 http://ftp.pdbj.org/pub/emdb/structures/EMD-24930 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24930 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24930 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24930_validation.pdf.gz emd_24930_validation.pdf.gz | 610.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24930_full_validation.pdf.gz emd_24930_full_validation.pdf.gz | 609.7 KB | 表示 | |
| XML形式データ |  emd_24930_validation.xml.gz emd_24930_validation.xml.gz | 10.7 KB | 表示 | |
| CIF形式データ |  emd_24930_validation.cif.gz emd_24930_validation.cif.gz | 14.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24930 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24930 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24930 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24930 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24930.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24930.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | High Cl Salicylate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.063 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM Structure of dolphin Prestin in complex with Salicylate: ...
| 全体 | 名称: Cryo-EM Structure of dolphin Prestin in complex with Salicylate: Inhibited I, Chloride + Salicylate |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM Structure of dolphin Prestin in complex with Salicylate: ...
| 超分子 | 名称: Cryo-EM Structure of dolphin Prestin in complex with Salicylate: Inhibited I, Chloride + Salicylate タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Prestin
| 分子 | 名称: Prestin / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 80.97375 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDHVEETEIL AATQRYYVER PIFSHPVLQE RLHKKDKISE SIGDKLKQAF TCTPKKIRNI IYMFLPITKW LPAYRFKEYV LGDIVSGIS TGVLQLPQGL AFAMLAAVPP VFGLYSSFYP VIMYCFFGTS RHISIGPFAV ISLMIGGVAV RLVPDDIVIP G GVNATNST ...文字列: MDHVEETEIL AATQRYYVER PIFSHPVLQE RLHKKDKISE SIGDKLKQAF TCTPKKIRNI IYMFLPITKW LPAYRFKEYV LGDIVSGIS TGVLQLPQGL AFAMLAAVPP VFGLYSSFYP VIMYCFFGTS RHISIGPFAV ISLMIGGVAV RLVPDDIVIP G GVNATNST EARDALRVKV AMSVTLLTGI IQFCLGVCRF GFVAIYLTEP LVRGFTTAAA VHVFTSMLKY LFGVKTKRYS GI FSVVYST VAVLQNVKNL NVCSLGVGLM VFGLLLGGKE FNERFKEKLP APIPLEFFAV VMGTGISAGF SLHESYNVDV VGT LPLGLL PPANPDTSLF HLVYVDAIAI AIVGFSVTIS MAKTLANKHG YQVDGNQELI ALGLCNSTGS LFQTFAISCS LSRS LVQEG TGGKTQLAGC LASLMILLVI LATGFLFESL PQAVLSAIVI VNLKGMFMQF SDLPFFWRTS KIELTIWLTT FVSSL FLGL DYGLITAVII ALMTVIYRTQ SPSYIVLGQL PDTDVYIDID AYEEVKEVPG IKIFQINAPI YYANSDLYSS ALKRKT GVN PAFILGARRK AMKKYAKEVG NANMANATVV KVDAEVDAED GTKPEEEEDE IKYPPIVTKS TLPEELQRFM PPGDNVH TI ILDFTQVNFM DSVGVKTLAG IVKEYGDVGI YVYLAGCSAQ VVSDLTQNQF FENPALLDLL FHSIHDAVLG SQVREALA E QEATAAPPQE DSEPNATPEA UniProtKB: Prestin |
-分子 #2: 2-HYDROXYBENZOIC ACID
| 分子 | 名称: 2-HYDROXYBENZOIC ACID / タイプ: ligand / ID: 2 / コピー数: 2 / 式: SAL |
|---|---|
| 分子量 | 理論値: 138.121 Da |
| Chemical component information | 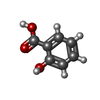 ChemComp-SAL: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 360 mM NaCl, 20 mM Tris-HCl, 3mM DTT, 1mM EDTA, 0.02 % GDN + 50 mM Sodium Salicylate | |||||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV 詳細: 4.5 s blot times, blot force 3, and double filter papers on each side of the vitrobot.. | |||||||||||||||||||||
| 詳細 | Monodisperse peak at around 14 ml using SEC (Superose 6 column) |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























