+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24519 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human p97-R155H mutant bound to ATPgS. | |||||||||
 マップデータ マップデータ | Cryo-EM structure of human p97-R155H bound to ATPgS. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | p97 / VCP / TERA / Inhibitor / CB-5083 / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / flavin adenine dinucleotide catabolic process / VCP-NSFL1C complex / endosome to lysosome transport via multivesicular body sorting pathway / endoplasmic reticulum stress-induced pre-emptive quality control / BAT3 complex binding / cellular response to arsenite ion / protein-DNA covalent cross-linking repair / Derlin-1 retrotranslocation complex / positive regulation of protein K63-linked deubiquitination ...: / flavin adenine dinucleotide catabolic process / VCP-NSFL1C complex / endosome to lysosome transport via multivesicular body sorting pathway / endoplasmic reticulum stress-induced pre-emptive quality control / BAT3 complex binding / cellular response to arsenite ion / protein-DNA covalent cross-linking repair / Derlin-1 retrotranslocation complex / positive regulation of protein K63-linked deubiquitination / cytoplasm protein quality control / positive regulation of oxidative phosphorylation / : / aggresome assembly / deubiquitinase activator activity / mitotic spindle disassembly / ubiquitin-modified protein reader activity / regulation of protein localization to chromatin / VCP-NPL4-UFD1 AAA ATPase complex / cellular response to misfolded protein / negative regulation of protein localization to chromatin / positive regulation of mitochondrial membrane potential / vesicle-fusing ATPase / K48-linked polyubiquitin modification-dependent protein binding / regulation of aerobic respiration / retrograde protein transport, ER to cytosol / stress granule disassembly / ATPase complex / regulation of synapse organization / ubiquitin-specific protease binding / positive regulation of ATP biosynthetic process / MHC class I protein binding / ubiquitin-like protein ligase binding / RHOH GTPase cycle / polyubiquitin modification-dependent protein binding / autophagosome maturation / negative regulation of hippo signaling / HSF1 activation / endoplasmic reticulum to Golgi vesicle-mediated transport / translesion synthesis / interstrand cross-link repair / ATP metabolic process / endoplasmic reticulum unfolded protein response / proteasomal protein catabolic process / Protein methylation / Attachment and Entry / ERAD pathway / lipid droplet / proteasome complex / viral genome replication / Josephin domain DUBs / negative regulation of smoothened signaling pathway / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / macroautophagy / establishment of protein localization / Hh mutants are degraded by ERAD / positive regulation of protein-containing complex assembly / Hedgehog ligand biogenesis / Defective CFTR causes cystic fibrosis / positive regulation of non-canonical NF-kappaB signal transduction / Translesion Synthesis by POLH / ADP binding / ABC-family proteins mediated transport / autophagy / cytoplasmic stress granule / Aggrephagy / positive regulation of protein catabolic process / azurophil granule lumen / Ovarian tumor domain proteases / KEAP1-NFE2L2 pathway / positive regulation of canonical Wnt signaling pathway / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / double-strand break repair / E3 ubiquitin ligases ubiquitinate target proteins / cellular response to heat / site of double-strand break / Neddylation / ubiquitin-dependent protein catabolic process / secretory granule lumen / protein phosphatase binding / regulation of apoptotic process / ficolin-1-rich granule lumen / proteasome-mediated ubiquitin-dependent protein catabolic process / Attachment and Entry / protein ubiquitination / ciliary basal body / protein domain specific binding / DNA repair / intracellular membrane-bounded organelle / DNA damage response / ubiquitin protein ligase binding / Neutrophil degranulation / lipid binding / endoplasmic reticulum membrane / perinuclear region of cytoplasm / glutamatergic synapse / endoplasmic reticulum / ATP hydrolysis activity / protein-containing complex / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
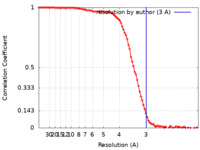
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Caffrey B / Zhu X / Berezuk A / Tuttle K / Chittori S / Subramaniam S | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2021 ジャーナル: J Biol Chem / 年: 2021タイトル: AAA+ ATPase p97/VCP mutants and inhibitor binding disrupt inter-domain coupling and subsequent allosteric activation. 著者: Brian Caffrey / Xing Zhu / Alison Berezuk / Katharine Tuttle / Sagar Chittori / Sriram Subramaniam /  要旨: The human AAA+ ATPase p97, also known as valosin-containing protein, a potential target for cancer therapeutics, plays a vital role in the clearing of misfolded proteins. p97 dysfunction is also ...The human AAA+ ATPase p97, also known as valosin-containing protein, a potential target for cancer therapeutics, plays a vital role in the clearing of misfolded proteins. p97 dysfunction is also known to play a crucial role in several neurodegenerative disorders, such as MultiSystem Proteinopathy 1 (MSP-1) and Familial Amyotrophic Lateral Sclerosis (ALS). However, the structural basis of its role in such diseases remains elusive. Here, we present cryo-EM structural analyses of four disease mutants p97, p97, p97, p97, as well as p97, implicated in resistance to the drug CB-5083, a potent p97 inhibitor. Our cryo-EM structures demonstrate that these mutations affect nucleotide-driven allosteric activation across the three principal p97 domains (N, D1, and D2) by predominantly interfering with either (1) the coupling between the D1 and N-terminal domains (p97 and p97), (2) the interprotomer interactions (p97), or (3) the coupling between D1 and D2 nucleotide domains (p97, p97). We also show that binding of the competitive inhibitor, CB-5083, to the D2 domain prevents conformational changes similar to those seen for mutations that affect coupling between the D1 and D2 domains. Our studies enable tracing of the path of allosteric activation across p97 and establish a common mechanistic link between active site inhibition and defects in allosteric activation by disease-causing mutations and have potential implications for the design of novel allosteric compounds that can modulate p97 function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24519.map.gz emd_24519.map.gz | 31.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24519-v30.xml emd-24519-v30.xml emd-24519.xml emd-24519.xml | 12.2 KB 12.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24519_fsc.xml emd_24519_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24519.png emd_24519.png | 61.7 KB | ||
| Filedesc metadata |  emd-24519.cif.gz emd-24519.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24519 http://ftp.pdbj.org/pub/emdb/structures/EMD-24519 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24519 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24519 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24519_validation.pdf.gz emd_24519_validation.pdf.gz | 517 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24519_full_validation.pdf.gz emd_24519_full_validation.pdf.gz | 516.5 KB | 表示 | |
| XML形式データ |  emd_24519_validation.xml.gz emd_24519_validation.xml.gz | 11 KB | 表示 | |
| CIF形式データ |  emd_24519_validation.cif.gz emd_24519_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24519 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24519 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24519 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24519 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7rl7MC  7rl6C  7rl9C  7rlaC  7rlbC  7rlcC  7rldC  7rlfC  7rlgC  7rlhC  7rliC  7rljC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24519.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24519.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of human p97-R155H bound to ATPgS. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0125 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Full-length Hexameric p97-R155H mutant.
| 全体 | 名称: Full-length Hexameric p97-R155H mutant. |
|---|---|
| 要素 |
|
-超分子 #1: Full-length Hexameric p97-R155H mutant.
| 超分子 | 名称: Full-length Hexameric p97-R155H mutant. / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 540 KDa |
-分子 #1: Transitional endoplasmic reticulum ATPase
| 分子 | 名称: Transitional endoplasmic reticulum ATPase / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: vesicle-fusing ATPase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 91.212602 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHGTSE NLYFQGASGA DSKGDDLSTA ILKQKNRPNR LIVDEAINED NSVVSLSQPK MDELQLFRGD TVLLKGKKRR EAVCIVLSD DTCSDEKIRM NRVVRNNLRV RLGDVISIQP CPDVKYGKRI HVLPIDDTVE GITGNLFEVY LKPYFLEAYR P IRKGDIFL ...文字列: HHHHHHGTSE NLYFQGASGA DSKGDDLSTA ILKQKNRPNR LIVDEAINED NSVVSLSQPK MDELQLFRGD TVLLKGKKRR EAVCIVLSD DTCSDEKIRM NRVVRNNLRV RLGDVISIQP CPDVKYGKRI HVLPIDDTVE GITGNLFEVY LKPYFLEAYR P IRKGDIFL VHGGMRAVEF KVVETDPSPY CIVAPDTVIH CEGEPIKRED EEESLNEVGY DDIGGCRKQL AQIKEMVELP LR HPALFKA IGVKPPRGIL LYGPPGTGKT LIARAVANET GAFFFLINGP EIMSKLAGES ESNLRKAFEE AEKNAPAIIF IDE LDAIAP KREKTHGEVE RRIVSQLLTL MDGLKQRAHV IVMAATNRPN SIDPALRRFG RFDREVDIGI PDATGRLEIL QIHT KNMKL ADDVDLEQVA NETHGHVGAD LAALCSEAAL QAIRKKMDLI DLEDETIDAE VMNSLAVTMD DFRWALSQSN PSALR ETVV EVPQVTWEDI GGLEDVKREL QELVQYPVEH PDKFLKFGMT PSKGVLFYGP PGCGKTLLAK AIANECQANF ISIKGP ELL TMWFGESEAN VREIFDKARQ AAPCVLFFDE LDSIAKARGG NIGDGGGAAD RVINQILTEM DGMSTKKNVF IIGATNR PD IIDPAILRPG RLDQLIYIPL PDEKSRVAIL KANLRKSPVA KDVDLEFLAK MTNGFSGADL TEICQRACKL AIRESIES E IRRERERQTN PSAMEVEEDD PVPEIRRDHF EEAMRFARRS VSDNDIRKYE MFAQTLQQSR GFGSFRFPSG NQGGAGPSQ GSGGGTGGSV YTEDNDDDLY G UniProtKB: Transitional endoplasmic reticulum ATPase |
-分子 #2: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 2 / コピー数: 12 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 12 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: Protein Storage Buffer with ATPgS. | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 5265 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)