+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23789 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Composite structure of EC+EC | |||||||||
 マップデータ マップデータ | Composite map of EC EC with sharpening by post processed ion Relion | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | pol II / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / : / : / : / TFIIF-class transcription factor complex binding / transcriptional start site selection at RNA polymerase II promoter / transcription factor TFIIF complex / RNA Polymerase I Transcription Initiation / Processing of Capped Intron-Containing Pre-mRNA ...: / : / : / : / : / TFIIF-class transcription factor complex binding / transcriptional start site selection at RNA polymerase II promoter / transcription factor TFIIF complex / RNA Polymerase I Transcription Initiation / Processing of Capped Intron-Containing Pre-mRNA / RNA Polymerase III Transcription Initiation From Type 2 Promoter / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / mRNA Capping / RNA polymerase II transcribes snRNA genes / TP53 Regulates Transcription of DNA Repair Genes / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / RNA polymerase II general transcription initiation factor activity / termination of RNA polymerase III transcription / RNA Polymerase II Pre-transcription Events / RNA-templated transcription / positive regulation of nuclear-transcribed mRNA poly(A) tail shortening / Formation of TC-NER Pre-Incision Complex / transcription initiation at RNA polymerase III promoter / RNA Polymerase I Promoter Escape / termination of RNA polymerase I transcription / nucleolar large rRNA transcription by RNA polymerase I / Gap-filling DNA repair synthesis and ligation in TC-NER / transcription initiation at RNA polymerase I promoter / Estrogen-dependent gene expression / maintenance of transcriptional fidelity during transcription elongation by RNA polymerase II / transcription by RNA polymerase III / positive regulation of translational initiation / Dual incision in TC-NER / protein phosphatase activator activity / RNA polymerase I complex / transcription elongation by RNA polymerase I / RNA polymerase III complex / RNA polymerase II, core complex / tRNA transcription by RNA polymerase III / RNA polymerase II preinitiation complex assembly / transcription by RNA polymerase I / translation initiation factor binding / transcription-coupled nucleotide-excision repair / transcription initiation at RNA polymerase II promoter / transcription elongation by RNA polymerase II / P-body / positive regulation of transcription elongation by RNA polymerase II / ribonucleoside binding / DNA-directed RNA polymerase / DNA-directed RNA polymerase activity / ribosome biogenesis / single-stranded DNA binding / nucleic acid binding / transcription by RNA polymerase II / single-stranded RNA binding / protein dimerization activity / nucleotide binding / regulation of DNA-templated transcription / nucleolus / DNA binding / zinc ion binding / nucleoplasm / metal ion binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.54 Å | |||||||||
 データ登録者 データ登録者 | Yang C / Murakami K | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2022 ジャーナル: Mol Cell / 年: 2022タイトル: Structural visualization of de novo transcription initiation by Saccharomyces cerevisiae RNA polymerase II. 著者: Chun Yang / Rina Fujiwara / Hee Jong Kim / Pratik Basnet / Yunye Zhu / Jose J Gorbea Colón / Stefan Steimle / Benjamin A Garcia / Craig D Kaplan / Kenji Murakami /  要旨: Previous structural studies of the initiation-elongation transition of RNA polymerase II (pol II) transcription have relied on the use of synthetic oligonucleotides, often artificially discontinuous ...Previous structural studies of the initiation-elongation transition of RNA polymerase II (pol II) transcription have relied on the use of synthetic oligonucleotides, often artificially discontinuous to capture pol II in the initiating state. Here, we report multiple structures of initiation complexes converted de novo from a 33-subunit yeast pre-initiation complex (PIC) through catalytic activities and subsequently stalled at different template positions. We determine that PICs in the initially transcribing complex (ITC) can synthesize a transcript of ∼26 nucleotides before transitioning to an elongation complex (EC) as determined by the loss of general transcription factors (GTFs). Unexpectedly, transition to an EC was greatly accelerated when an ITC encountered a downstream EC stalled at promoter proximal regions and resulted in a collided head-to-end dimeric EC complex. Our structural analysis reveals a dynamic state of TFIIH, the largest of GTFs, in PIC/ITC with distinct functional consequences at multiple steps on the pathway to elongation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23789.map.gz emd_23789.map.gz | 64.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23789-v30.xml emd-23789-v30.xml emd-23789.xml emd-23789.xml | 35.7 KB 35.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23789.png emd_23789.png | 45.2 KB | ||
| Filedesc metadata |  emd-23789.cif.gz emd-23789.cif.gz | 10.5 KB | ||
| その他 |  emd_23789_additional_1.map.gz emd_23789_additional_1.map.gz | 45.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23789 http://ftp.pdbj.org/pub/emdb/structures/EMD-23789 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23789 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23789 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23789_validation.pdf.gz emd_23789_validation.pdf.gz | 479.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23789_full_validation.pdf.gz emd_23789_full_validation.pdf.gz | 478.9 KB | 表示 | |
| XML形式データ |  emd_23789_validation.xml.gz emd_23789_validation.xml.gz | 6.6 KB | 表示 | |
| CIF形式データ |  emd_23789_validation.cif.gz emd_23789_validation.cif.gz | 7.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23789 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23789 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23789 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23789 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7meiMC  7mk9C  7mkaC  7ml0C  7ml1C  7ml2C  7ml3C  7ml4C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10865 (タイトル: Structural visualization of de novo initiation of RNA polymerase II transcription EMPIAR-10865 (タイトル: Structural visualization of de novo initiation of RNA polymerase II transcriptionData size: 14.3 TB Data #1: raw micrographs for PIC + ITC maps [micrographs - multiframe] Data #2: raw micrographs for EC+EC map [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23789.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23789.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map of EC EC with sharpening by post processed ion Relion | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
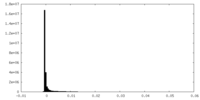
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Composite map of EC EC without sharpening
| ファイル | emd_23789_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map of EC EC without sharpening | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
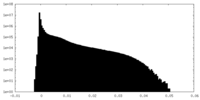
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Head-to-end Colliding RNAPII Elongation
+超分子 #1: Head-to-end Colliding RNAPII Elongation
+分子 #1: DNA (74-MER)
+分子 #2: DNA (74-MER)
+分子 #3: DNA-directed RNA polymerase subunit
+分子 #4: DNA-directed RNA polymerase subunit beta
+分子 #5: DNA-directed RNA polymerase II subunit RPB3
+分子 #6: DNA-directed RNA polymerase II subunit RPB4
+分子 #7: DNA-directed RNA polymerases I, II, and III subunit RPABC1
+分子 #8: DNA-directed RNA polymerases I, II, and III subunit RPABC2
+分子 #9: DNA-directed RNA polymerase II subunit RPB7
+分子 #10: DNA-directed RNA polymerases I, II, and III subunit RPABC3
+分子 #11: DNA-directed RNA polymerase II subunit RPB9
+分子 #12: DNA-directed RNA polymerases II subunit RPABC5
+分子 #13: DNA-directed RNA polymerase II subunit RPB11
+分子 #14: DNA-directed RNA polymerases II subunit RPABC4
+分子 #17: Transcription initiation factor IIF subunit alpha
+分子 #18: Transcription initiation factor IIF subunit beta
+分子 #15: RNA (5'-R(P*AP*AP*CP*UP*AP*GP*UP*UP*AP*AP*GP*AP*GP*GP*UP*U)-3')
+分子 #16: RNA (5'-R(P*UP*CP*GP*AP*GP*AP*GP*GP*AP*UP*CP*CP*CP*AP*A)-3')
+分子 #19: ZINC ION
+分子 #20: MAGNESIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 20 mM HEPES PH 7.6 50 mM KoAc 5 mM DTT 2 mM MgoAc |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 |
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM CPC |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 1.25 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.75 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 81000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)