+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23494 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM of the SLFN12-PDE3A complex: PDE3A body refinement | |||||||||
 マップデータ マップデータ | Body output from Relion Multibody refinement | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | complex / velcrin / molecular glue / DNMDP / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報3',5'-cGMP-inhibited cyclic-nucleotide phosphodiesterase activity / estrogen binding / positive regulation of oocyte development / regulation of ribonuclease activity / regulation of meiotic nuclear division / cellular response to cGMP / rRNA catabolic process / negative regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway / positive regulation of vascular permeability / negative regulation of vascular permeability ...3',5'-cGMP-inhibited cyclic-nucleotide phosphodiesterase activity / estrogen binding / positive regulation of oocyte development / regulation of ribonuclease activity / regulation of meiotic nuclear division / cellular response to cGMP / rRNA catabolic process / negative regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway / positive regulation of vascular permeability / negative regulation of vascular permeability / negative regulation of cAMP/PKA signal transduction / oocyte maturation / : / 3',5'-cyclic-nucleotide phosphodiesterase / nuclear estrogen receptor activity / 3',5'-cyclic-nucleotide phosphodiesterase activity / 3',5'-cyclic-GMP phosphodiesterase activity / 3',5'-cyclic-AMP phosphodiesterase activity / RNA nuclease activity / cellular response to transforming growth factor beta stimulus / : / apoptotic signaling pathway / lipid metabolic process / ribosome binding / G alpha (s) signalling events / 加水分解酵素; エステル加水分解酵素 / G protein-coupled receptor signaling pathway / response to xenobiotic stimulus / negative regulation of apoptotic process / metal ion binding / nucleus / membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.97 Å | |||||||||
 データ登録者 データ登録者 | Fuller JR / Garvie CW / Lemke CT | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structure of PDE3A-SLFN12 complex reveals requirements for activation of SLFN12 RNase. 著者: Colin W Garvie / Xiaoyun Wu / Malvina Papanastasiou / Sooncheol Lee / James Fuller / Gavin R Schnitzler / Steven W Horner / Andrew Baker / Terry Zhang / James P Mullahoo / Lindsay Westlake / ...著者: Colin W Garvie / Xiaoyun Wu / Malvina Papanastasiou / Sooncheol Lee / James Fuller / Gavin R Schnitzler / Steven W Horner / Andrew Baker / Terry Zhang / James P Mullahoo / Lindsay Westlake / Stephanie H Hoyt / Marcus Toetzl / Matthew J Ranaghan / Luc de Waal / Joseph McGaunn / Bethany Kaplan / Federica Piccioni / Xiaoping Yang / Martin Lange / Adrian Tersteegen / Donald Raymond / Timothy A Lewis / Steven A Carr / Andrew D Cherniack / Christopher T Lemke / Matthew Meyerson / Heidi Greulich /   要旨: DNMDP and related compounds, or velcrins, induce complex formation between the phosphodiesterase PDE3A and the SLFN12 protein, leading to a cytotoxic response in cancer cells that express elevated ...DNMDP and related compounds, or velcrins, induce complex formation between the phosphodiesterase PDE3A and the SLFN12 protein, leading to a cytotoxic response in cancer cells that express elevated levels of both proteins. The mechanisms by which velcrins induce complex formation, and how the PDE3A-SLFN12 complex causes cancer cell death, are not fully understood. Here, we show that PDE3A and SLFN12 form a heterotetramer stabilized by binding of DNMDP. Interactions between the C-terminal alpha helix of SLFN12 and residues near the active site of PDE3A are required for complex formation, and are further stabilized by interactions between SLFN12 and DNMDP. Moreover, we demonstrate that SLFN12 is an RNase, that PDE3A binding increases SLFN12 RNase activity, and that SLFN12 RNase activity is required for DNMDP response. This new mechanistic understanding will facilitate development of velcrin compounds into new cancer therapies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23494.map.gz emd_23494.map.gz | 95 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23494-v30.xml emd-23494-v30.xml emd-23494.xml emd-23494.xml | 25.3 KB 25.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23494_fsc.xml emd_23494_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23494.png emd_23494.png | 90.2 KB | ||
| マスクデータ |  emd_23494_msk_1.map emd_23494_msk_1.map | 129.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-23494.cif.gz emd-23494.cif.gz | 7.7 KB | ||
| その他 |  emd_23494_additional_1.map.gz emd_23494_additional_1.map.gz emd_23494_half_map_1.map.gz emd_23494_half_map_1.map.gz emd_23494_half_map_2.map.gz emd_23494_half_map_2.map.gz | 75.4 MB 93.1 MB 93.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23494 http://ftp.pdbj.org/pub/emdb/structures/EMD-23494 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23494 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23494 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23494_validation.pdf.gz emd_23494_validation.pdf.gz | 707.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23494_full_validation.pdf.gz emd_23494_full_validation.pdf.gz | 707.4 KB | 表示 | |
| XML形式データ |  emd_23494_validation.xml.gz emd_23494_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_23494_validation.cif.gz emd_23494_validation.cif.gz | 24.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23494 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23494 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23494 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23494 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23494.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23494.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Body output from Relion Multibody refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.063 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
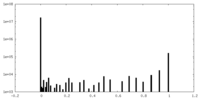
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23494_msk_1.map emd_23494_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
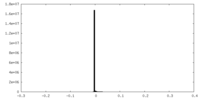
| 密度ヒストグラム |
-追加マップ: Sharpened by an automatically-determined B-factor (-117.379) and filtered...
| ファイル | emd_23494_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened by an automatically-determined B-factor (-117.379) and filtered to local resolution, using the Relion local resolution implementation. Model was primarily refined against this map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
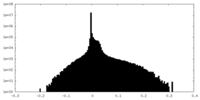
| 密度ヒストグラム |
-ハーフマップ: Body half-map 2 from Relion Multibody refinement
| ファイル | emd_23494_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Body half-map 2 from Relion Multibody refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
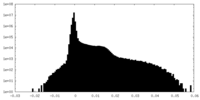
| 密度ヒストグラム |
-ハーフマップ: Body half-map 1 from Relion Multibody refinement
| ファイル | emd_23494_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Body half-map 1 from Relion Multibody refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SLFN12-PDE3A complex, PDE3A body with interacting peptide from SLFN12
| 全体 | 名称: SLFN12-PDE3A complex, PDE3A body with interacting peptide from SLFN12 |
|---|---|
| 要素 |
|
-超分子 #1: SLFN12-PDE3A complex, PDE3A body with interacting peptide from SLFN12
| 超分子 | 名称: SLFN12-PDE3A complex, PDE3A body with interacting peptide from SLFN12 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: cGMP-inhibited 3',5'-cyclic phosphodiesterase A
| 分子 | 名称: cGMP-inhibited 3',5'-cyclic phosphodiesterase A / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: 3',5'-cyclic-nucleotide phosphodiesterase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 57.352996 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GEDETECLRE PLRKASACST YAPETMMFLD KPILAPEPLV MDNLDSIMEQ LNTWNFPIFD LVENIGRKCG RILSQVSYRL FEDMGLFEA FKIPIREFMN YFHALEIGYR DIPYHNRIHA TDVLHAVWYL TTQPIPGLST VINDHGSTSD SDSDSGFTHG H MGYVFSKT ...文字列: GEDETECLRE PLRKASACST YAPETMMFLD KPILAPEPLV MDNLDSIMEQ LNTWNFPIFD LVENIGRKCG RILSQVSYRL FEDMGLFEA FKIPIREFMN YFHALEIGYR DIPYHNRIHA TDVLHAVWYL TTQPIPGLST VINDHGSTSD SDSDSGFTHG H MGYVFSKT YNVTDDKYGC LSGNIPALEL MALYVAAAMH DYDHPGRTNA FLVATSAPQA VLYNDRSVLE NHHAAAAWNL FM SRPEYNF LINLDHVEFK HFRFLVIEAI LATDLKKHFD FVAKFNGKVN DDVGIDWTNE NDRLLVCQMC IKLADINGPA KCK ELHLQW TDGIVNEFYE QGDEEASLGL PISPFMDRSA PQLANLQESF ISHIVGPLCN SYDSAGLMPG KWVEDSDESG DTDD PEEEE EEAPAPNEEE TCENNESPKK KTFKRRKIYC QITQHLLQNH KMWKKVIEEE QRLAGIENQS LDQTPQSHSS EQIQA IKEE EEEKGKPRGE EIPTQKPDQ UniProtKB: cGMP-inhibited 3',5'-cyclic phosphodiesterase 3A |
-分子 #2: Schlafen family member 12
| 分子 | 名称: Schlafen family member 12 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 67.383734 KDa |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: GGGGSMNISV DLETNYAELV LDVGRVTLGE NSRKKMKDCK LRKKQNESVS RAMCALLNSG GGVIKAEIEN EDYSYTKDGI GLDLENSFS NILLFVPEYL DFMQNGNYFL IFVKSWSLNT SGLRITTLSS NLYKRDITSA KVMNATAALE FLKDMKKTRG R LYLRPELL ...文字列: GGGGSMNISV DLETNYAELV LDVGRVTLGE NSRKKMKDCK LRKKQNESVS RAMCALLNSG GGVIKAEIEN EDYSYTKDGI GLDLENSFS NILLFVPEYL DFMQNGNYFL IFVKSWSLNT SGLRITTLSS NLYKRDITSA KVMNATAALE FLKDMKKTRG R LYLRPELL AKRPCVDIQE ENNMKALAGV FFDRTELDRK EKLTFTESTH VEIKNFSTEK LLQRIKEILP QYVSAFANTD GG YLFIGLN EDKEIIGFKA EMSDLDDLER EIEKSIRKMP VHHFCMEKKK INYSCKFLGV YDKGSLCGYV CALRVERFCC AVF AKEPDS WHVKDNRVMQ LTRKEWIQFM VEAEPKFSSS YEEVISQINT SLPAPHSWPL LEWQRQRHHC PGLSGRITYT PENL CRKLF LQHEGLKQLI CEEMDSVRKG SLIFSRSWSV DLGLQENHKV LCDALLISQD SPPVLYTFHM VQDEEFKGYS TQTAL TLKQ KLAKIGGYTK KVCVMTKIFY LSPEGMTSCQ YDLRSQVIYP ESYYFTRRKY LLKALFKALK RLKSLRDQFS FAENLY QII GIDCFQKNDK KMFKSCRRLT UniProtKB: Ribonuclease SLFN12 |
-分子 #3: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: (4~{R})-3-[4-(diethylamino)-3-[oxidanyl(oxidanylidene)-$l^{4}-aza...
| 分子 | 名称: (4~{R})-3-[4-(diethylamino)-3-[oxidanyl(oxidanylidene)-$l^{4}-azanyl]phenyl]-4-methyl-4,5-dihydro-1~{H}-pyridazin-6-one タイプ: ligand / ID: 5 / コピー数: 2 / 式: X5M |
|---|---|
| 分子量 | 理論値: 305.352 Da |
| Chemical component information | 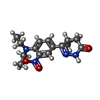 ChemComp-X5M: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 0.0038% NP-40s detergent (CAS 9016-45-9) added immediately prior to plunge freezing | |||||||||||||||
| グリッド | モデル: C-flat / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV / 詳細: 4.5 second blot time. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 3163 / 平均電子線量: 50.0 e/Å2 詳細: Exposures were collected in super-resolution mode, as movies fractionated over 40 frames |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | The atomic model (including per-residue ADP/B-factors) was refined in Phenix against the sharpened/local resolution-filtered map |
| 精密化 | 空間: REAL |
| 得られたモデル |  PDB-7lrc: |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)