+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23110 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of S. Pombe NatC complex with a Bisubstrate inhibitor and inositol hexaphosphate | |||||||||
 マップデータ マップデータ | NatC complex with a Bisubstrate inhibitor and inositol hexaphosphate | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NatB / NAA20 / NAA25 / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報N-terminal methionine Nalpha-acetyltransferase NatC / NatC complex / protein N-terminal-methionine acetyltransferase activity / protein-N-terminal amino-acid acetyltransferase activity / inositol hexakisphosphate binding / protein maturation / endoplasmic reticulum membrane / endoplasmic reticulum / RNA binding / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |    Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.16 Å | |||||||||
 データ登録者 データ登録者 | Deng S / Marmorstein R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2021 ジャーナル: Structure / 年: 2021タイトル: Molecular mechanism of N-terminal acetylation by the ternary NatC complex. 著者: Sunbin Deng / Leah Gottlieb / Buyan Pan / Julianna Supplee / Xuepeng Wei / E James Petersson / Ronen Marmorstein /  要旨: Protein N-terminal acetylation is predominantly a ribosome-associated modification, with NatA-E serving as the major enzymes. NatC is the most unusual of these enzymes, containing one Naa30 catalytic ...Protein N-terminal acetylation is predominantly a ribosome-associated modification, with NatA-E serving as the major enzymes. NatC is the most unusual of these enzymes, containing one Naa30 catalytic subunit and two auxiliary subunits, Naa35 and Naa38; and substrate selectivity profile that overlaps with NatE. Here, we report the cryoelectron microscopy structure of S. pombe NatC with a NatE/C-type bisubstrate analog and inositol hexaphosphate (IP), and associated biochemistry studies. We find that the presence of three subunits is a prerequisite for normal NatC acetylation activity in yeast and that IP binds tightly to NatC to stabilize the complex. We also describe the molecular basis for IP-mediated NatC complex stabilization and the overlapping yet distinct substrate profiles of NatC and NatE. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23110.map.gz emd_23110.map.gz | 28 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23110-v30.xml emd-23110-v30.xml emd-23110.xml emd-23110.xml | 22.2 KB 22.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23110.png emd_23110.png | 174.6 KB | ||
| マスクデータ |  emd_23110_msk_1.map emd_23110_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-23110.cif.gz emd-23110.cif.gz | 6.6 KB | ||
| その他 |  emd_23110_half_map_1.map.gz emd_23110_half_map_1.map.gz emd_23110_half_map_2.map.gz emd_23110_half_map_2.map.gz | 27.8 MB 27.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23110 http://ftp.pdbj.org/pub/emdb/structures/EMD-23110 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23110 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23110 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23110_validation.pdf.gz emd_23110_validation.pdf.gz | 958.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23110_full_validation.pdf.gz emd_23110_full_validation.pdf.gz | 958.4 KB | 表示 | |
| XML形式データ |  emd_23110_validation.xml.gz emd_23110_validation.xml.gz | 10.8 KB | 表示 | |
| CIF形式データ |  emd_23110_validation.cif.gz emd_23110_validation.cif.gz | 12.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23110 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23110 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23110 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23110 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23110.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23110.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | NatC complex with a Bisubstrate inhibitor and inositol hexaphosphate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
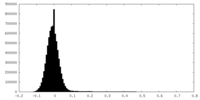
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23110_msk_1.map emd_23110_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
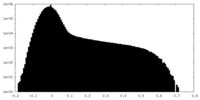
| 密度ヒストグラム |
-ハーフマップ: NatC complex with a Bisubstrate inhibitor and inositol hexaphosphate
| ファイル | emd_23110_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | NatC complex with a Bisubstrate inhibitor and inositol hexaphosphate | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
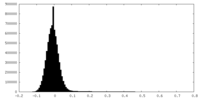
| 密度ヒストグラム |
-ハーフマップ: NatC complex with a Bisubstrate inhibitor and inositol hexaphosphate
| ファイル | emd_23110_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | NatC complex with a Bisubstrate inhibitor and inositol hexaphosphate | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : heterotimeric S.pombe NatC complex with a bisubstrate inhibitor a...
| 全体 | 名称: heterotimeric S.pombe NatC complex with a bisubstrate inhibitor and inositol hexaphosphate |
|---|---|
| 要素 |
|
-超分子 #1: heterotimeric S.pombe NatC complex with a bisubstrate inhibitor a...
| 超分子 | 名称: heterotimeric S.pombe NatC complex with a bisubstrate inhibitor and inositol hexaphosphate タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: N-alpha-acetyltransferase NatC, catalytic subunit NAA30, auxiliar...
| 超分子 | 名称: N-alpha-acetyltransferase NatC, catalytic subunit NAA30, auxiliary subunits NAA35 and NAA38 タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: CoA-Ac-MLGP bi-substrate
| 超分子 | 名称: CoA-Ac-MLGP bi-substrate / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #4 |
|---|
-分子 #1: N-alpha-acetyltransferase 30
| 分子 | 名称: N-alpha-acetyltransferase 30 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: N-terminal methionine Nalpha-acetyltransferase NatC |
|---|---|
| 由来(天然) | 生物種:  株: 972 / ATCC 24843 |
| 分子量 | 理論値: 17.72135 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVTIVPYSHQ YLKDICQLIQ KDLSEPYSKY VYRYFVHQWP EFSFVALDND RFIGAVICKQ DVHRGTTLRG YIAMLAIVKE YRGQGIATK LTQASLDVMK NRGAQEIVLE TEVDNEAAMS FYERLGFCRY KRLYRYYLNG TDAFRYILYP N UniProtKB: N-alpha-acetyltransferase 30 |
-分子 #2: N-alpha-acetyltransferase 35, NatC auxiliary subunit
| 分子 | 名称: N-alpha-acetyltransferase 35, NatC auxiliary subunit タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: 972 / ATCC 24843 |
| 分子量 | 理論値: 80.541312 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSVKESLSLL NSMQGNVKIG NVEPAKGNEG YVDNAGYVDC TKSYFEATKS LKEEQLVCDP KFTLLDSISA FEIMEPKMDS GIDYQPLRV DFSRDLSYLE ILALMDLIVS AEKEWHYGSP LSESLLCSAH VFSICKSPIS QVGDSGFSSG SGRNTTDIVL F PFVLAVIK ...文字列: MSVKESLSLL NSMQGNVKIG NVEPAKGNEG YVDNAGYVDC TKSYFEATKS LKEEQLVCDP KFTLLDSISA FEIMEPKMDS GIDYQPLRV DFSRDLSYLE ILALMDLIVS AEKEWHYGSP LSESLLCSAH VFSICKSPIS QVGDSGFSSG SGRNTTDIVL F PFVLAVIK CCDIVHREFL MGNLYDEEDI SSFSYHMSFL QNYPIEKLNY LLQSSIEYLA SEVIKFSAEL RQIIEGILNR IQ LRIGILR VYERSDIKTT IDALHLIKNL VPEIQNTVSV VDSSIKESIL KQYWDFRVQA QLVATAPVRN IPPTGIEHSY QRI LYFADD MLLILNSHTL ASSLAVYQFC LDFTRLNRTP EPYVRSSLQA LITANNAVNL RDQPTSYMLE CIREFSGLPS NFYN PNTRT VIEKNSISSA YGPLVESLIA HSTNIMVDLV RICSHNPCRF RRNLINLLPE ITVAHFEAEA LDLKFVAKSL PSNGP FSSF IYHVKLNAIE HILLSSFEQK LHQPYQWPHF FAVLDHVFSI HQTHLELHGK DRNTPPMAKT FVTYLHRILN AIKETY SGY LLLTVLCMRL NIIKTPSFTL DEKIQESYYM AHYRPLINLR QPKPLLRSEA DCIIKNLQNF STDDLIIKSN EKFTAAK NS LINVIKSGFE QNEFINPYFL QTNYLKNLLC CCITNLVSLA ILSKDHSANL KIVEIPGNPL PSLSRT UniProtKB: N-alpha-acetyltransferase 35, NatC auxiliary subunit |
-分子 #3: N-alpha-acetyltransferase 38, NatC auxiliary subunit
| 分子 | 名称: N-alpha-acetyltransferase 38, NatC auxiliary subunit タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: 972 / ATCC 24843 |
| 分子量 | 理論値: 13.258555 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MALHYFLQYD VQILCIALMF SIFRVCISTA IDFTSPKLDE FSLIMEMGEI LLTSWLNRSV HIEIFDERKF IGKFLCTDRE GAAILSNTT EYNKGFSRAL GLVVIPGKHI KSFSVRA UniProtKB: N-alpha-acetyltransferase 38, NatC auxiliary subunit |
-分子 #4: MLGP peptide
| 分子 | 名称: MLGP peptide / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 416.535 Da |
| 配列 | 文字列: MLGP |
-分子 #5: INOSITOL HEXAKISPHOSPHATE
| 分子 | 名称: INOSITOL HEXAKISPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: IHP |
|---|---|
| 分子量 | 理論値: 660.035 Da |
| Chemical component information |  ChemComp-IHP: |
-分子 #6: CARBOXYMETHYL COENZYME *A
| 分子 | 名称: CARBOXYMETHYL COENZYME *A / タイプ: ligand / ID: 6 / コピー数: 1 / 式: CMC |
|---|---|
| 分子量 | 理論値: 825.57 Da |
| Chemical component information | 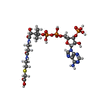 ChemComp-CMC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #0 - 平均電子線量: 1.6 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #1 - 平均電子線量: 1.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7l1k: |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)