+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23002 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
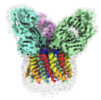
| タイトル | Cryo-EM structure of the CRAC channel Orai in an open conformation; H206A gain-of-function mutation in complex with an antibody | |||||||||
 マップデータ マップデータ | cryo-EM map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ion channel / calcium / SOCE / CRAC / eukaryotic / open structure / MEMBRANE PROTEIN / amphiboles / cryo-EM / TRANSPORT PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報store-operated calcium entry / store-operated calcium channel activity / positive regulation of calcineurin-NFAT signaling cascade / positive regulation of calcium ion transport / calcium channel complex / calcium channel regulator activity / calcium ion transmembrane transport / calcium channel activity / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Long SB / Hou X | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Cryo-EM structure of the calcium release-activated calcium channel Orai in an open conformation. 著者: Xiaowei Hou / Ian R Outhwaite / Leanne Pedi / Stephen Barstow Long /  要旨: The calcium release-activated calcium channel Orai regulates Ca entry into non-excitable cells and is required for proper immune function. While the channel typically opens following Ca release from ...The calcium release-activated calcium channel Orai regulates Ca entry into non-excitable cells and is required for proper immune function. While the channel typically opens following Ca release from the endoplasmic reticulum, certain pathologic mutations render the channel constitutively open. Previously, using one such mutation (H206A), we obtained low (6.7 Å) resolution X-ray structural information on Orai in an open conformation (Hou et al., 2018). Here we present a structure of this open conformation at 3.3 Å resolution using fiducial-assisted cryo-electron microscopy. The improved structure reveals the conformations of amino acids in the open pore, which dilates by outward movements of subunits. A ring of phenylalanine residues repositions to expose previously shielded glycine residues to the pore without significant rotational movement of the associated helices. Together with other hydrophobic amino acids, the phenylalanines act as the channel's gate. Structured M1-M2 turrets, not evident previously, form the channel's extracellular entrance. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23002.map.gz emd_23002.map.gz | 67.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23002-v30.xml emd-23002-v30.xml emd-23002.xml emd-23002.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23002.png emd_23002.png | 199.8 KB | ||
| Filedesc metadata |  emd-23002.cif.gz emd-23002.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23002 http://ftp.pdbj.org/pub/emdb/structures/EMD-23002 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23002 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23002 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23002_validation.pdf.gz emd_23002_validation.pdf.gz | 499.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23002_full_validation.pdf.gz emd_23002_full_validation.pdf.gz | 498.8 KB | 表示 | |
| XML形式データ |  emd_23002_validation.xml.gz emd_23002_validation.xml.gz | 6.4 KB | 表示 | |
| CIF形式データ |  emd_23002_validation.cif.gz emd_23002_validation.cif.gz | 7.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23002 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23002 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23002 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23002 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23002.map.gz / 形式: CCP4 / 大きさ: 72.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23002.map.gz / 形式: CCP4 / 大きさ: 72.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo-EM map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.87078 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Orai channel, H206A mutant, in complex with three Fab fragments
| 全体 | 名称: Orai channel, H206A mutant, in complex with three Fab fragments |
|---|---|
| 要素 |
|
-超分子 #1: Orai channel, H206A mutant, in complex with three Fab fragments
| 超分子 | 名称: Orai channel, H206A mutant, in complex with three Fab fragments タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-超分子 #2: Orai channel, H206A mutant
| 超分子 | 名称: Orai channel, H206A mutant / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Fab fragments
| 超分子 | 名称: Fab fragments / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Calcium release-activated calcium channel protein 1
| 分子 | 名称: Calcium release-activated calcium channel protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 24.160168 KDa |
| 組換発現 | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MSQSGEDLHS PTYLSWRKLQ LSRAKLKASS KTSALLSGFA MVAMVEVQLD HDTNVPPGML IAFAICTTLL VAVAMLALMI STCILPNIE TVSNLHSISL VHESPHERLH WYIETAWAFS TLLGLILFLL EIAILCWVKF YDLSPPAAWS ATVVLIPVMI I FMAFAIHF ...文字列: MSQSGEDLHS PTYLSWRKLQ LSRAKLKASS KTSALLSGFA MVAMVEVQLD HDTNVPPGML IAFAICTTLL VAVAMLALMI STCILPNIE TVSNLHSISL VHESPHERLH WYIETAWAFS TLLGLILFLL EIAILCWVKF YDLSPPAAWS ATVVLIPVMI I FMAFAIHF YRSLVSHKYE VTVSGIRELE MLKEQMEQDH LEHHNNIRNN GEGEEF UniProtKB: Calcium release-activated calcium channel protein 1 |
-分子 #2: 19B5 Fab heavy chain
| 分子 | 名称: 19B5 Fab heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 26.886477 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTLNMLLGLK WVFFVVFYQG VHCEVQLVES GGGLVQPKGS LKLSCAASGF TFNTYAMHWV RQAPGKGLEW VARIRTKSNN YATYYADSV KDRFTISRDD SQSMLYLQMN NLKTEDTAMY YCVRQKYGNY FDYWGQGTTL TVSSAKTTPP SVYPLAPGSA A QTNSMVTL ...文字列: MTLNMLLGLK WVFFVVFYQG VHCEVQLVES GGGLVQPKGS LKLSCAASGF TFNTYAMHWV RQAPGKGLEW VARIRTKSNN YATYYADSV KDRFTISRDD SQSMLYLQMN NLKTEDTAMY YCVRQKYGNY FDYWGQGTTL TVSSAKTTPP SVYPLAPGSA A QTNSMVTL GCLVKGYFPE PVTVTWNSGS LSSGVHTFPA VLQSDLYTLS SSVTVPSSSW PSETVTCNVA HPASSTKVDK KI VPRD |
-分子 #3: 19B5 Fab light chain
| 分子 | 名称: 19B5 Fab light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.587295 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MESQTQVFVY MLLWLSGVDG DVVMTQSQKF MSTSVGDRVS VTCKASQNVG INVAWYQQKP GQSPKALINS ASYRNSGVPD RFTGGGSGT DFTLTINNVQ SEDLAEYFCQ QCNSYPLTFG AGTKLELRRA DAAPTVSIFP PSSEQLTSGG ASVVCFLNNF Y PKDINVKW ...文字列: MESQTQVFVY MLLWLSGVDG DVVMTQSQKF MSTSVGDRVS VTCKASQNVG INVAWYQQKP GQSPKALINS ASYRNSGVPD RFTGGGSGT DFTLTINNVQ SEDLAEYFCQ QCNSYPLTFG AGTKLELRRA DAAPTVSIFP PSSEQLTSGG ASVVCFLNNF Y PKDINVKW KIDGSERQNG VLNSWTDQDS KDSTYSMSST LTLTKDEYER HNSYTCEATH KTSTSPIVKS FNRN |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 平均露光時間: 10.0 sec. / 平均電子線量: 76.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)





















