+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22653 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of full-length influenza HA with a head-binding antibody at pH 5.2, conformation A, neutral pH-like | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | influenza / virus / hemagglutinin / antibody / low pH / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Influenza A virus (A/Hong Kong/1/1968(H3N2)) (A型インフルエンザウイルス) / Influenza A virus (A/Hong Kong/1/1968(H3N2)) (A型インフルエンザウイルス) /  Influenza A virus (strain A/Hong Kong/1/1968 H3N2) (A型インフルエンザウイルス) / Influenza A virus (strain A/Hong Kong/1/1968 H3N2) (A型インフルエンザウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Gui M / Gao J / Xiang Y | |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2020 ジャーナル: PLoS Pathog / 年: 2020タイトル: Structural intermediates in the low pH-induced transition of influenza hemagglutinin. 著者: Jingjing Gao / Miao Gui / Ye Xiang /  要旨: The hemagglutinin (HA) glycoproteins of influenza viruses play a key role in binding host cell receptors and in mediating virus-host cell membrane fusion during virus infection. Upon virus entry, HA ...The hemagglutinin (HA) glycoproteins of influenza viruses play a key role in binding host cell receptors and in mediating virus-host cell membrane fusion during virus infection. Upon virus entry, HA is triggered by low pH and undergoes large structural rearrangements from a prefusion state to a postfusion state. While structures of prefusion state and postfusion state of HA have been reported, the intermediate structures remain elusive. Here, we report two distinct low pH intermediate conformations of the influenza virus HA using cryo-electron microscopy (cryo-EM). Our results show that a decrease in pH from 7.8 to 5.2 triggers the release of fusion peptides from the binding pockets and then causes a dramatic conformational change in the central helices, in which the membrane-proximal ends of the central helices unwind to an extended form. Accompanying the conformational changes of the central helices, the stem region of the HA undergoes an anticlockwise rotation of 9.5 degrees and a shift of 15 Å. The HA head, after being stabilized by an antibody, remains unchanged compared to the neutral pH state. Thus, the conformational change of the HA stem region observed in our research is likely to be independent of the HA head. These results provide new insights into the structural transition of HA during virus entry. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22653.map.gz emd_22653.map.gz | 7.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22653-v30.xml emd-22653-v30.xml emd-22653.xml emd-22653.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22653_fsc.xml emd_22653_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22653.png emd_22653.png | 116.2 KB | ||
| マスクデータ |  emd_22653_msk_1.map emd_22653_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-22653.cif.gz emd-22653.cif.gz | 6.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22653 http://ftp.pdbj.org/pub/emdb/structures/EMD-22653 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22653 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22653 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22653_validation.pdf.gz emd_22653_validation.pdf.gz | 405.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22653_full_validation.pdf.gz emd_22653_full_validation.pdf.gz | 405.5 KB | 表示 | |
| XML形式データ |  emd_22653_validation.xml.gz emd_22653_validation.xml.gz | 11.5 KB | 表示 | |
| CIF形式データ |  emd_22653_validation.cif.gz emd_22653_validation.cif.gz | 15.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22653 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22653 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22653 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22653 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22653.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22653.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.33 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
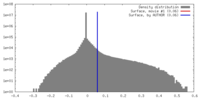
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22653_msk_1.map emd_22653_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : complex of hemagglutinin with antibody
| 全体 | 名称: complex of hemagglutinin with antibody |
|---|---|
| 要素 |
|
-超分子 #1: complex of hemagglutinin with antibody
| 超分子 | 名称: complex of hemagglutinin with antibody / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/Hong Kong/1/1968(H3N2)) (A型インフルエンザウイルス) Influenza A virus (A/Hong Kong/1/1968(H3N2)) (A型インフルエンザウイルス) |
-超分子 #2: hemagglutinin
| 超分子 | 名称: hemagglutinin / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|
-超分子 #3: antigen-binding fragment
| 超分子 | 名称: antigen-binding fragment / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|
-分子 #1: Hemagglutinin
| 分子 | 名称: Hemagglutinin / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (strain A/Hong Kong/1/1968 H3N2) (A型インフルエンザウイルス) Influenza A virus (strain A/Hong Kong/1/1968 H3N2) (A型インフルエンザウイルス)株: A/Hong Kong/1/1968 H3N2 |
| 分子量 | 理論値: 37.948812 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKTIIALSYI FCLALGQDLP GNDNSTATLC LGHHAVPNGT LVKTITDDQI EVTNATELVQ SSSTGKICNN PHRILDGIDC TLIDALLGD PHCDVFQNET WDLFVERSKA FSNCYPYDVP DYASLRSLVA SSGTLEFITE GFTWTGVTQN GGSNACKRGP G SGFFSRLN ...文字列: MKTIIALSYI FCLALGQDLP GNDNSTATLC LGHHAVPNGT LVKTITDDQI EVTNATELVQ SSSTGKICNN PHRILDGIDC TLIDALLGD PHCDVFQNET WDLFVERSKA FSNCYPYDVP DYASLRSLVA SSGTLEFITE GFTWTGVTQN GGSNACKRGP G SGFFSRLN WLTKSGSTYP VLNVTMPNND NFDKLYIWGV HHPSTNQEQT SLYVQASGRV TVSTRRSQQT IIPNIGSRPW VR GLSSRIS IYWTIVKPGD VLVINSNGNL IAPRGYFKMR TGKSSIMRSD APIDTCISEC ITPNGSIPND KPFQNVNKIT YGA CPKYVK QNTLKLATGM RNVPEKQTR UniProtKB: Hemagglutinin |
-分子 #2: Hemagglutinin
| 分子 | 名称: Hemagglutinin / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (strain A/Hong Kong/1/1968 H3N2) (A型インフルエンザウイルス) Influenza A virus (strain A/Hong Kong/1/1968 H3N2) (A型インフルエンザウイルス)株: A/Hong Kong/1/1968 H3N2 |
| 分子量 | 理論値: 28.947525 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GLFGAIAGFI ENGWEGMIDG WYGFRHQNSE GTGQAADLKS TQAAIDQING KLNRVIEKTN EKFHQIEKEF SEVEGRIQDL EKYVEDTKI DLWSYNAELL VALENQHTID LTDSEMNKLF EKTRRQLREN AEDMGNGCFK IYHKCDNACI ESIRNGTYDH D VYRDEALN ...文字列: GLFGAIAGFI ENGWEGMIDG WYGFRHQNSE GTGQAADLKS TQAAIDQING KLNRVIEKTN EKFHQIEKEF SEVEGRIQDL EKYVEDTKI DLWSYNAELL VALENQHTID LTDSEMNKLF EKTRRQLREN AEDMGNGCFK IYHKCDNACI ESIRNGTYDH D VYRDEALN NRFQIKGVEL KSGYKDWILW ISFAISCFLL CVVLLGFIMW ACQRGNIRCN ICISMGWSHP QFEKGGGARG GS GGGSWSH PQFEKGF UniProtKB: Hemagglutinin |
-分子 #3: antibody Fab light chain
| 分子 | 名称: antibody Fab light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.11241 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSVLTQPPSV SGAPGQRVTI SCTGSSSNIG AGYAVHWYQQ LPGTAPKLLI SGNSNRPSGV PDRFSGSKSG TSASLAITGL QAEDEADYY CQSYDSSLSG SVFGGGTKLT VLGRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV ...文字列: QSVLTQPPSV SGAPGQRVTI SCTGSSSNIG AGYAVHWYQQ LPGTAPKLLI SGNSNRPSGV PDRFSGSKSG TSASLAITGL QAEDEADYY CQSYDSSLSG SVFGGGTKLT VLGRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV TEQDSKDSTY SLSSTLTLSK ADYEKHKVYA CEVTHQGLSS PVTKSFNRGE C |
-分子 #4: antibody Fab heavy chain
| 分子 | 名称: antibody Fab heavy chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.278605 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSGAE VKKPGASVTV SCQVSGYTLT SYGLSWVRQA PGQGLEWVGW INTYDGQTKY VKKFQGRVTM TTHTGTNTAY MEMKSLRSD DTAVYYCARV EGVRGVMGFH YYPMDVWGQG TMVTVSSKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: QVQLVQSGAE VKKPGASVTV SCQVSGYTLT SYGLSWVRQA PGQGLEWVGW INTYDGQTKY VKKFQGRVTM TTHTGTNTAY MEMKSLRSD DTAVYYCARV EGVRGVMGFH YYPMDVWGQG TMVTVSSKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSCDK GLEVLFQ |
-分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 8 / コピー数: 9 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 5.2 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)