+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22400 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Rous sarcoma virus cleaved synaptic complex (CSC) with HIV-1 integrase strand transfer inhibitor MK-2048 | |||||||||
 マップデータ マップデータ | Contour level 0.4 set in Chimera with step level 1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | intasome / integrase-viral DNA complex / HYDROLASE-DNA-INHIBITOR complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ribonuclease H / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / RNA stem-loop binding / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / 転移酵素; リンを含む基を移すもの; 核酸を移すもの ...ribonuclease H / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / RNA stem-loop binding / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / viral nucleocapsid / DNA recombination / DNA-directed DNA polymerase / aspartic-type endopeptidase activity / 加水分解酵素; エステル加水分解酵素 / DNA-directed DNA polymerase activity / viral translational frameshifting / symbiont entry into host cell / proteolysis / DNA binding / zinc ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス) Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.21 Å | |||||||||
 データ登録者 データ登録者 | Pandey KK / Bera S | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2021 ジャーナル: Commun Biol / 年: 2021タイトル: Cryo-EM structure of the Rous sarcoma virus octameric cleaved synaptic complex intasome. 著者: Krishan K Pandey / Sibes Bera / Ke Shi / Michael J Rau / Amarachi V Oleru / James A J Fitzpatrick / Alan N Engelman / Hideki Aihara / Duane P Grandgenett /  要旨: Despite conserved catalytic integration mechanisms, retroviral intasomes composed of integrase (IN) and viral DNA possess diverse structures with variable numbers of IN subunits. To investigate ...Despite conserved catalytic integration mechanisms, retroviral intasomes composed of integrase (IN) and viral DNA possess diverse structures with variable numbers of IN subunits. To investigate intasome assembly mechanisms, we employed the Rous sarcoma virus (RSV) IN dimer that assembles a precursor tetrameric structure in transit to the mature octameric intasome. We determined the structure of RSV octameric intasome stabilized by a HIV-1 IN strand transfer inhibitor using single particle cryo-electron microscopy. The structure revealed significant flexibility of the two non-catalytic distal IN dimers along with previously unrecognized movement of the conserved intasome core, suggesting ordered conformational transitions between intermediates that may be important to capture the target DNA. Single amino acid substitutions within the IN C-terminal domain affected intasome assembly and function in vitro and infectivity of pseudotyped RSV virions. Unexpectedly, 17 C-terminal amino acids of IN were dispensable for virus infection despite regulating the transition of the tetrameric intasome to the octameric form in vitro. We speculate that this region may regulate the binding of highly flexible distal IN dimers to the intasome core to form the octameric complex. Our studies reveal key steps in the assembly of RSV intasomes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22400.map.gz emd_22400.map.gz | 203.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22400-v30.xml emd-22400-v30.xml emd-22400.xml emd-22400.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22400_fsc.xml emd_22400_fsc.xml | 13.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22400.png emd_22400.png | 72.8 KB | ||
| Filedesc metadata |  emd-22400.cif.gz emd-22400.cif.gz | 6.7 KB | ||
| その他 |  emd_22400_half_map_1.map.gz emd_22400_half_map_1.map.gz emd_22400_half_map_2.map.gz emd_22400_half_map_2.map.gz | 200.2 MB 200.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22400 http://ftp.pdbj.org/pub/emdb/structures/EMD-22400 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22400 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22400 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22400_validation.pdf.gz emd_22400_validation.pdf.gz | 916.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22400_full_validation.pdf.gz emd_22400_full_validation.pdf.gz | 916.4 KB | 表示 | |
| XML形式データ |  emd_22400_validation.xml.gz emd_22400_validation.xml.gz | 21.5 KB | 表示 | |
| CIF形式データ |  emd_22400_validation.cif.gz emd_22400_validation.cif.gz | 28 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22400 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22400 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22400 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22400 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22400.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22400.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Contour level 0.4 set in Chimera with step level 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half-map A: Contour level 0.4 set in Chimera with step level 1
| ファイル | emd_22400_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map A: Contour level 0.4 set in Chimera with step level 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map B: Contour level 0.4 set in Chimera with step level 1
| ファイル | emd_22400_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map B: Contour level 0.4 set in Chimera with step level 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cleaved synaptic complex (CSC) formed with Rous sarcoma virus int...
| 全体 | 名称: Cleaved synaptic complex (CSC) formed with Rous sarcoma virus integrase and viral DNA in presence of HIV-1 integrase strand inhibitor MK-2048 |
|---|---|
| 要素 |
|
-超分子 #1: Cleaved synaptic complex (CSC) formed with Rous sarcoma virus int...
| 超分子 | 名称: Cleaved synaptic complex (CSC) formed with Rous sarcoma virus integrase and viral DNA in presence of HIV-1 integrase strand inhibitor MK-2048 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス) Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス) |
| 分子量 | 理論値: 257 KDa |
-分子 #1: integrase
| 分子 | 名称: integrase / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO EC番号: 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; アスパラギン酸プロテアーゼ |
|---|---|
| 由来(天然) | 生物種:  Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス) Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス)株: Schmidt-Ruppin A |
| 分子量 | 理論値: 30.926582 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PLREAKDLHT ALHIGPRALS KACNISMQQA REVVQTCPHC NSAPALEAGV NPRGLGPLQI WQTDFTLEPR MAPRSWLAVT VDTASSAIV VTQHGRVTSV AVQHHWATAI AVLGRPKAIK TDNGSCFTSK STREWLARWG IAHTTGIPGN SQGQAMVERA N RLLKDKIR ...文字列: PLREAKDLHT ALHIGPRALS KACNISMQQA REVVQTCPHC NSAPALEAGV NPRGLGPLQI WQTDFTLEPR MAPRSWLAVT VDTASSAIV VTQHGRVTSV AVQHHWATAI AVLGRPKAIK TDNGSCFTSK STREWLARWG IAHTTGIPGN SQGQAMVERA N RLLKDKIR VLAEGDGFMK RIPTSKQGEL LAKAMYALNH FERGENTKTP IQKHWRPTVL TEGPPVKIRI ETGEWEKGWN VL VWGRGYA AVKNRDTDKV IWVPSRKVKP DITQKDEVTK K UniProtKB: Gag-Pol polyprotein |
-分子 #2: DNA (5'-D(*AP*AP*TP*GP*TP*TP*GP*TP*CP*TP*TP*AP*TP*GP*CP*AP*AP*T)-3')
| 分子 | 名称: DNA (5'-D(*AP*AP*TP*GP*TP*TP*GP*TP*CP*TP*TP*AP*TP*GP*CP*AP*AP*T)-3') タイプ: dna / ID: 2 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス) Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス) |
| 分子量 | 理論値: 5.5206 KDa |
| 配列 | 文字列: (DA)(DA)(DT)(DG)(DT)(DT)(DG)(DT)(DC)(DT) (DT)(DA)(DT)(DG)(DC)(DA)(DA)(DT) |
-分子 #3: DNA (5'-D(*AP*TP*TP*GP*CP*AP*TP*AP*AP*GP*AP*CP*AP*AP*CP*A)-3')
| 分子 | 名称: DNA (5'-D(*AP*TP*TP*GP*CP*AP*TP*AP*AP*GP*AP*CP*AP*AP*CP*A)-3') タイプ: dna / ID: 3 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス) Rous sarcoma virus (strain Schmidt-Ruppin A) (ラウス肉腫ウイルス) |
| 分子量 | 理論値: 4.899232 KDa |
| 配列 | 文字列: (DA)(DT)(DT)(DG)(DC)(DA)(DT)(DA)(DA)(DG) (DA)(DC)(DA)(DA)(DC)(DA) |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #5: (6S)-2-(3-chloro-4-fluorobenzyl)-8-ethyl-10-hydroxy-N,6-dimethyl-...
| 分子 | 名称: (6S)-2-(3-chloro-4-fluorobenzyl)-8-ethyl-10-hydroxy-N,6-dimethyl-1,9-dioxo-1,2,6,7,8,9-hexahydropyrazino[1',2':1,5]pyrrolo[2,3-d]pyridazine-4-carboxamide タイプ: ligand / ID: 5 / コピー数: 2 / 式: ZZX |
|---|---|
| 分子量 | 理論値: 461.874 Da |
| Chemical component information | 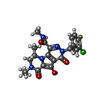 ChemComp-ZZX: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 12 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 5187 / 平均電子線量: 66.0 e/Å2 詳細: Images were collected in movie mode at 0.2 seconds per frame. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 30 / 当てはまり具合の基準: correlation coefficient |
| 得られたモデル |  PDB-7jn3: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)







































