+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22252 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CRYOEM STRUCTURE OF MYCOBACTERIUM TUBERCULOSIS ZINC METALLOPROTEASE ZMP1 IN OPEN STATE | |||||||||
 マップデータ マップデータ | ZINC METALLOPROTEASE ZMP1 IN OPEN STATE | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Open state / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌) Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌) | |||||||||
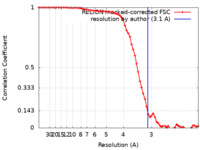
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Liang WG / Zhao M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2021 ジャーナル: Structure / 年: 2021タイトル: Structural analysis of Mycobacterium tuberculosis M13 metalloprotease Zmp1 open states. 著者: Wenguang G Liang / Jordan M Mancl / Minglei Zhao / Wei-Jen Tang /  要旨: Zinc metalloprotease 1 (Zmp1), a Mycobacterium tuberculosis 75 kDa secreted enzyme, mediates key stages of tuberculosis disease progression. The biological activity of Zmp1 presumably stems from its ...Zinc metalloprotease 1 (Zmp1), a Mycobacterium tuberculosis 75 kDa secreted enzyme, mediates key stages of tuberculosis disease progression. The biological activity of Zmp1 presumably stems from its ability to degrade bacterium- and/or host-derived peptides. The crystal structures of Zmp1 and related M13 metalloproteases, such as neprilysin and endothelin-converting enzyme-1 were determined only in the closed conformation, which cannot capture substrates or release proteolytic products. Thus, the mechanisms of substrate binding and selectivity remain elusive. Here we report two open-state cryo-EM structures of Zmp1, revealed by our SAXS analysis to be the dominant states in solution. Our structural analyses reveal how ligand binding induces a conformational switch in four linker regions to drive the rigid body motion of the D1 and D2 domains, which form the sizable catalytic chamber. Furthermore, they offer insights into the catalytic cycle and mechanism of substrate recognition of M13 metalloproteases for future therapeutic innovations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22252.map.gz emd_22252.map.gz | 20.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22252-v30.xml emd-22252-v30.xml emd-22252.xml emd-22252.xml | 22.4 KB 22.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22252_fsc.xml emd_22252_fsc.xml | 5.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22252.png emd_22252.png | 110.6 KB | ||
| マスクデータ |  emd_22252_msk_1.map emd_22252_msk_1.map | 27 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-22252.cif.gz emd-22252.cif.gz | 6.7 KB | ||
| その他 |  emd_22252_additional_1.map.gz emd_22252_additional_1.map.gz emd_22252_half_map_1.map.gz emd_22252_half_map_1.map.gz emd_22252_half_map_2.map.gz emd_22252_half_map_2.map.gz | 25.3 MB 20.7 MB 20.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22252 http://ftp.pdbj.org/pub/emdb/structures/EMD-22252 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22252 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22252 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22252_validation.pdf.gz emd_22252_validation.pdf.gz | 741.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22252_full_validation.pdf.gz emd_22252_full_validation.pdf.gz | 741.2 KB | 表示 | |
| XML形式データ |  emd_22252_validation.xml.gz emd_22252_validation.xml.gz | 12.5 KB | 表示 | |
| CIF形式データ |  emd_22252_validation.cif.gz emd_22252_validation.cif.gz | 17.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22252 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22252 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22252 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22252 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22252.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22252.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ZINC METALLOPROTEASE ZMP1 IN OPEN STATE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.063 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
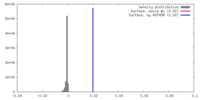
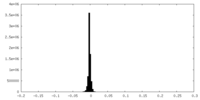
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22252_msk_1.map emd_22252_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
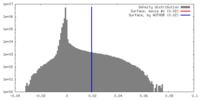
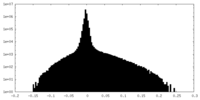
| 密度ヒストグラム |
-追加マップ: sharpen map
| ファイル | emd_22252_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpen map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
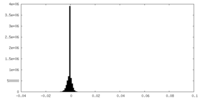
| 密度ヒストグラム |
-ハーフマップ: ZINC METALLOPROTEASE ZMP1 IN OPEN STATE
| ファイル | emd_22252_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ZINC METALLOPROTEASE ZMP1 IN OPEN STATE | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
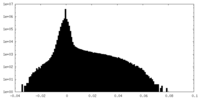
| 密度ヒストグラム |
-ハーフマップ: ZINC METALLOPROTEASE ZMP1 IN OPEN STATE
| ファイル | emd_22252_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ZINC METALLOPROTEASE ZMP1 IN OPEN STATE | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MYCOBACTERIUM TUBERCULOSIS ZINC METALLOPROTEASE
| 全体 | 名称: MYCOBACTERIUM TUBERCULOSIS ZINC METALLOPROTEASE |
|---|---|
| 要素 |
|
-超分子 #1: MYCOBACTERIUM TUBERCULOSIS ZINC METALLOPROTEASE
| 超分子 | 名称: MYCOBACTERIUM TUBERCULOSIS ZINC METALLOPROTEASE / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌) Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌)株: ATCC 25618 / H37Rv |
-分子 #1: Probable zinc metalloprotease Zmp1
| 分子 | 名称: Probable zinc metalloprotease Zmp1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌) Mycobacterium tuberculosis (strain ATCC 25618 / H37Rv) (結核菌)株: ATCC 25618 / H37Rv |
| 分子量 | 理論値: 78.132953 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRGSHHHHHH GMASMTGGQQ MGRDLYDDDD KDHPFTMTLA IPSGIDLSHI DADARPQDDL FGHVNGRWLA EHEIPADRAT DGAFRSLFD RAETQVRDLI IQASQAGAAV GTDAQRIGDL YASFLDEEAV ERAGVQPLHD ELATIDSAAD ATELAAALGT L QRAGVGGG ...文字列: MRGSHHHHHH GMASMTGGQQ MGRDLYDDDD KDHPFTMTLA IPSGIDLSHI DADARPQDDL FGHVNGRWLA EHEIPADRAT DGAFRSLFD RAETQVRDLI IQASQAGAAV GTDAQRIGDL YASFLDEEAV ERAGVQPLHD ELATIDSAAD ATELAAALGT L QRAGVGGG IGVYVDTDSK DSTRYLVHFT QSGIGLPDES YYRDEQHAAV LAAYPGHIAR MFGLVYGGES RDHAKTADRI VA LETKLAD AHWDVVKRRD ADLGYNLRTF AQLQTEGAGF DWVSWVTALG SAPDAMTELV VRQPDYLVTF ASLWASVNVE DWK CWARWR LIRARAPWLT RALVAEDFEF YGRTLTGAQQ LRDRWKRGVS LVENLMGDAV GKLYVQRHFP PDAKSRIDTL VDNL QEAYR ISISELDWMT PQTRQRALAK LNKFTAKVGY PIKWRDYSKL AIDRDDLYGN VQRGYAVNHD RELAKLFGPV DRDEW FMTP QTVNAYYNPG MNEIVFPAAI LQPPFFDPQA DEAANYGGIG AVIGHEIGHG FDDQGAKYDG DGNLVDWWTD DDRTEF AAR TKALIEQYHA YTPRDLVDHP GPPHVQGAFT IGENIGDLGG LSIALLAYQL SLNGNPAPVI DGLTGMQRVF FGWAQIW RT KSRAAEAIRR LAVDPHSPPE FRCNGVVRNV DAFYQAFDVT EDDALFLDPQ RRVRIWN UniProtKB: Probable zinc metalloprotease Zmp1 |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 6.8 詳細: 20 mM HEPES (pH 6.8), 150 mM NaCl, 0.5 mM bMe, 20 mM Trimethylamine N-oxide dihydrate |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 308 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 5.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.4000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)