+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21450 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CON-S ch.DS.SOSIP HIV trimer bound to DH845 Fab | ||||||||||||
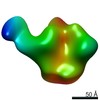
 マップデータ マップデータ | CON-S ch.DS.SOSIP HIV trimer bound to DH845 Fab | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 21.0 Å | ||||||||||||
 データ登録者 データ登録者 | Cai F / Chen WH / Wu W / Jones J / Misook C / Gohain N / Shen X / LaBranche C / Eaton A / Sutherland L ...Cai F / Chen WH / Wu W / Jones J / Misook C / Gohain N / Shen X / LaBranche C / Eaton A / Sutherland L / Hernandez G / Nelson RW / Scearce R / Seaman M / Moody MA / Santra S / Wiehe KJ / Tomaras GD / Wagh K / Korber B / Bonsignori M / Montefiori DC / Haynes BF / de Val N / Joyce GM / Saunders KO | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: HIV-1 vaccination elicits structurally and genetically-convergent neutralizing antibodies in non-human primates 著者: Cai F / Chen WH / Wu W / Jones J / Misook C / Gohain N / Shen X / LaBranche C / Eaton A / Sutherland L / Hernandez G / Nelson RW / Scearce R / Seaman M / Moody MA / Santra S / Wiehe KJ / ...著者: Cai F / Chen WH / Wu W / Jones J / Misook C / Gohain N / Shen X / LaBranche C / Eaton A / Sutherland L / Hernandez G / Nelson RW / Scearce R / Seaman M / Moody MA / Santra S / Wiehe KJ / Tomaras GD / Wagh K / Korber B / Bonsignori M / Montefiori DC / Haynes BF / de Val N / Joyce GM / Saunders KO | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21450.map.gz emd_21450.map.gz | 5.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21450-v30.xml emd-21450-v30.xml emd-21450.xml emd-21450.xml | 12.5 KB 12.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21450.png emd_21450.png | 15.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21450 http://ftp.pdbj.org/pub/emdb/structures/EMD-21450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21450 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21450_validation.pdf.gz emd_21450_validation.pdf.gz | 295.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21450_full_validation.pdf.gz emd_21450_full_validation.pdf.gz | 294.8 KB | 表示 | |
| XML形式データ |  emd_21450_validation.xml.gz emd_21450_validation.xml.gz | 5.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21450 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21450 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21450 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21450 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21450.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21450.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CON-S ch.DS.SOSIP HIV trimer bound to DH845 Fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.19 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CON-S ch.DS.SOSIP in complex with DH845 Fab
| 全体 | 名称: CON-S ch.DS.SOSIP in complex with DH845 Fab |
|---|---|
| 要素 |
|
-超分子 #1: CON-S ch.DS.SOSIP in complex with DH845 Fab
| 超分子 | 名称: CON-S ch.DS.SOSIP in complex with DH845 Fab / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK 293F cells Homo sapiens (ヒト) / 組換細胞: HEK 293F cells |
| 分子量 | 実験値: 50 KDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate 詳細: grids were glow discharged and using 0.7% UF they were stained. | |||||||||
| グリッド | モデル: C-flat / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI EAGLE (2k x 2k) / 撮影したグリッド数: 1 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.1 µm / 最小 デフォーカス(公称値): 1.1 µm / 倍率(公称値): 100000 |
| 試料ステージ | 試料ホルダーモデル: OTHER |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















