+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21156 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of Escherichia coli 2-oxoglutarate dehydrogenase E1 component sucA | |||||||||
 マップデータ マップデータ | Escherichia coli 2-oxoglutarate dehydrogenase E1 component sucA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TCA cycle / 2-oxoglutarate dehydrogenase complex (OGDH) / E1 component / sucA / AMP / Oxaloacetate (OAA) / Dimer / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報oxoglutarate dehydrogenase (succinyl-transferring) / oxoglutarate dehydrogenase (succinyl-transferring) activity / oxoglutarate dehydrogenase complex / thiamine pyrophosphate binding / tricarboxylic acid cycle / nucleotide binding / magnesium ion binding / identical protein binding / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.08 Å | |||||||||
 データ登録者 データ登録者 | Gao H | |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM Structure of Escherichia coli 2-oxoglutarate dehydrogenase E1 component sucA 著者: Gao H | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21156.map.gz emd_21156.map.gz | 28.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21156-v30.xml emd-21156-v30.xml emd-21156.xml emd-21156.xml | 11.6 KB 11.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
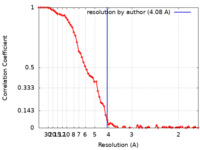
| FSC (解像度算出) |  emd_21156_fsc.xml emd_21156_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21156.png emd_21156.png | 158.5 KB | ||
| Filedesc metadata |  emd-21156.cif.gz emd-21156.cif.gz | 5.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21156 http://ftp.pdbj.org/pub/emdb/structures/EMD-21156 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21156 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21156 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21156_validation.pdf.gz emd_21156_validation.pdf.gz | 587.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21156_full_validation.pdf.gz emd_21156_full_validation.pdf.gz | 586.6 KB | 表示 | |
| XML形式データ |  emd_21156_validation.xml.gz emd_21156_validation.xml.gz | 9.6 KB | 表示 | |
| CIF形式データ |  emd_21156_validation.cif.gz emd_21156_validation.cif.gz | 12.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21156 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21156 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21156 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21156 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21156.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21156.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Escherichia coli 2-oxoglutarate dehydrogenase E1 component sucA | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.877 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Escherichia coli 2-oxoglutarate dehydrogenase E1 component sucA
| 全体 | 名称: Escherichia coli 2-oxoglutarate dehydrogenase E1 component sucA |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia coli 2-oxoglutarate dehydrogenase E1 component sucA
| 超分子 | 名称: Escherichia coli 2-oxoglutarate dehydrogenase E1 component sucA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: 2-oxoglutarate dehydrogenase E1 component
| 分子 | 名称: 2-oxoglutarate dehydrogenase E1 component / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号: oxoglutarate dehydrogenase (succinyl-transferring) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 94.942508 KDa |
| 配列 | 文字列: DTNVKQVKVL QLINAYRFRG HQHANLDPLG LYQQDKVADL DPSFHDLTEA DFQETFNVGS FASGKETMKL GELLEALKQT YCGPIGAEY MHITSTEEKR WIQQRIESGR ATFNSEEKKR FLSELTAAEG LERYLGAKFP GAKRFSLEGD ALIPMLKEMI R HAGNSGTR ...文字列: DTNVKQVKVL QLINAYRFRG HQHANLDPLG LYQQDKVADL DPSFHDLTEA DFQETFNVGS FASGKETMKL GELLEALKQT YCGPIGAEY MHITSTEEKR WIQQRIESGR ATFNSEEKKR FLSELTAAEG LERYLGAKFP GAKRFSLEGD ALIPMLKEMI R HAGNSGTR EVVLGMAHRG RLNVLVNVLG KKPQDLFDEF AGKHKEHLGT GDVKYHMGFS SDFQTDGGLV HLALAFNPSH LE IVSPVVI GSVRARLDRL DEPSSNKVLP ITIHGDAAVT GQGVVQETLN MSKARGYEVG GTVRIVINNQ VGFTTSNPLD ARS TPYMTD IGKMVQAPIF HVNADDPEAV AFVTRLALDF RNTFKRDVFI DLVCYRRHGH NEADEPSATQ KIKKHPTPRK IYAD KLEQE KVATLEDATE MVNLYRDALD AGDCVVAEWR PMNMHSFTWS PYLNHEWDEE YPNKVEMKRL QELAKRISTV PEAVE MQSR VAKIYGDRQA MAAGEKLFDW GGAENLAYAT LVDEGIPVRL SGEDSGRGTF FHRHAVIHNQ SNGSTYTPLQ HIHNGQ GAF RVWDSVLSEE AVLAFEYGYA TAEPRTLTIW EAQFGDFANG AQVVIDQFIS SGEQKWGRMC GLVMLLPHGY EGQGPEH SS ARLERYLQLC AEQNMQVCVP STPAQVYHML RRQALRGMRR PLVVMSPKSL LRHPLAVSSL EELANGTFLP AIGEIDEL D PKGVKRVVMC SGKVYYDLLE QRRKNNQHDV AIVRIEQLYP FPHKAMQEVL QQFAHVKDFV WCQEEPLNQG AWYCSQHHF REVIPFGASL RYAGRPASAS PAVGHMSVHQ KQQQDLVNDA LNVE UniProtKB: Oxoglutarate dehydrogenase (succinyl-transferring) |
-分子 #2: ADENOSINE MONOPHOSPHATE
| 分子 | 名称: ADENOSINE MONOPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: AMP |
|---|---|
| 分子量 | 理論値: 347.221 Da |
| Chemical component information |  ChemComp-AMP: |
-分子 #3: OXALOACETATE ION
| 分子 | 名称: OXALOACETATE ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: OAA |
|---|---|
| 分子量 | 理論値: 131.064 Da |
| Chemical component information |  ChemComp-OAA: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 80 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 800 / 平均露光時間: 2.0 sec. / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)