+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20581 | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ structure of BmCPV RNA dependent RNA polymerase at quiescent state | |||||||||||||||||||||||||||
 マップデータ マップデータ | Sub-particle reconstruction of BmCPV in the quiescent state | |||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
 キーワード キーワード | RdRp / VIRAL PROTEIN / TRANSFERASE | |||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral genome replication / RNA-directed RNA polymerase / RNA-directed RNA polymerase activity / RNA binding 類似検索 - 分子機能 | |||||||||||||||||||||||||||
| 生物種 |   Bombyx mori cytoplasmic polyhedrosis virus (ウイルス) Bombyx mori cytoplasmic polyhedrosis virus (ウイルス) | |||||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Cui YX / Zhang YN | |||||||||||||||||||||||||||
| 資金援助 |  中国, 中国,  米国, 8件 米国, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2019 ジャーナル: Nat Struct Mol Biol / 年: 2019タイトル: Conservative transcription in three steps visualized in a double-stranded RNA virus. 著者: Yanxiang Cui / Yinong Zhang / Kang Zhou / Jingchen Sun / Z Hong Zhou /   要旨: Endogenous RNA transcription characterizes double-stranded RNA (dsRNA) viruses in the Reoviridae, a family that is exemplified by its simple, single-shelled member cytoplasmic polyhedrosis virus (CPV) ...Endogenous RNA transcription characterizes double-stranded RNA (dsRNA) viruses in the Reoviridae, a family that is exemplified by its simple, single-shelled member cytoplasmic polyhedrosis virus (CPV). Because of the lack of in situ structures of the intermediate stages of RNA-dependent RNA polymerase (RdRp) during transcription, it is poorly understood how RdRp detects environmental cues and internal transcriptional states to initiate and coordinate repeated cycles of transcript production inside the capsid. Here, we captured five high-resolution (2.8-3.5 Å) RdRp-RNA in situ structures-representing quiescent, initiation, early elongation, elongation and abortive states-under seven experimental conditions of CPV. We observed the 'Y'-form initial RNA fork in the initiation state and the complete transcription bubble in the elongation state. These structures reveal that de novo RNA transcription involves three major conformational changes during state transitions. Our results support an ouroboros model for endogenous conservative transcription in dsRNA viruses. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20581.map.gz emd_20581.map.gz | 58.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20581-v30.xml emd-20581-v30.xml emd-20581.xml emd-20581.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20581.png emd_20581.png | 104.6 KB | ||
| Filedesc metadata |  emd-20581.cif.gz emd-20581.cif.gz | 7.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20581 http://ftp.pdbj.org/pub/emdb/structures/EMD-20581 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20581 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20581 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20581_validation.pdf.gz emd_20581_validation.pdf.gz | 745.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20581_full_validation.pdf.gz emd_20581_full_validation.pdf.gz | 744.8 KB | 表示 | |
| XML形式データ |  emd_20581_validation.xml.gz emd_20581_validation.xml.gz | 6.2 KB | 表示 | |
| CIF形式データ |  emd_20581_validation.cif.gz emd_20581_validation.cif.gz | 7.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20581 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20581 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20581 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20581 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20581.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20581.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sub-particle reconstruction of BmCPV in the quiescent state | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.062 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bombyx mori cytoplasmic polyhedrosis virus
| 全体 | 名称:   Bombyx mori cytoplasmic polyhedrosis virus (ウイルス) Bombyx mori cytoplasmic polyhedrosis virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Bombyx mori cytoplasmic polyhedrosis virus
| 超分子 | 名称: Bombyx mori cytoplasmic polyhedrosis virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 / NCBI-ID: 110829 / 生物種: Bombyx mori cytoplasmic polyhedrosis virus / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
-分子 #1: RNA-dependent RNA Polymerase
| 分子 | 名称: RNA-dependent RNA Polymerase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bombyx mori cytoplasmic polyhedrosis virus (ウイルス) Bombyx mori cytoplasmic polyhedrosis virus (ウイルス) |
| 分子量 | 理論値: 139.007125 KDa |
| 配列 | 文字列: MLPNTELYNT IFSETRKFTR ESFKEIEHLT AKLANDRVAR HDFLFNNSIA LISDYSGEDS NGNQLQATVT IPNEITNPKE YDPSDYPLA EDESFFKQGH KYDYLVTFRA GSLTNTYEPK TKMYKLHAAL DKLMHVKQRK SRFADLWREL CAVIASLDVW Y QTTNYPLR ...文字列: MLPNTELYNT IFSETRKFTR ESFKEIEHLT AKLANDRVAR HDFLFNNSIA LISDYSGEDS NGNQLQATVT IPNEITNPKE YDPSDYPLA EDESFFKQGH KYDYLVTFRA GSLTNTYEPK TKMYKLHAAL DKLMHVKQRK SRFADLWREL CAVIASLDVW Y QTTNYPLR TYVKLLFHKG DEFPFYESPS QDKIIFNDKS VASILPTFVY TCCQVGTAIM SGILTHVESI VAMNHFLHCA KD SYIDEKL KIKGIGRSWY QEALHNVGRA TVPVWSQFNE VIGHRTKTTS EPHFVSSTFI SLRAKRAELL YPEFNEYINR ALR LSKTQN DVANYYAACR AMTNDGTFLA TLTELSLDAA VFPRIEQRLV TRPAVLMSNT RHESLKQKYA NGVGSIAQSY LSSF TDEIA KRVNGIHHDE AWLNFLTTSS PGRKLTEIEK LEVGGDVAAW SNSRIVMQAV FAREYRTPER IFKSLKAPIK LVERQ QSDR RQRAISGLDN DRLFLSFMPY TIGKQIYDLN DNAAQGKQAG NAFDIGEMLY WTSQRNVLLS SIDVAGMDAS VTTNTK DIY NTFVLDVASK CTVPRFGPYY AKNMEVFEVG KRQSQVKYVN AAWQACALEA ANSQTSTSYE SEIFGQVKNA EGTYPSG RA DTSTHHTVLL QGLVRGNELK RASDGKNSCL TTIKILGDDI MEIFQGNEND THDHAVSNAS ILNESGFATT AELSQNSI V LLQQLVVNGT FWGFADRISL WTREDTKDIG RLNLAMMELN ALIDDLLFRV RRPEGLKMLG FFCGAICLRR FTLSVDNKL YDSTYNNLSK YMTLVKYDKN PDFDSTLMSL ILPLAWLFMP RGGEYPAYPF ERRDGTFTED ESMFTARGAY KRRLLYDVSN IREMIQQNS MVLDDDLLHE YGFTGALLLI DLNILDLIDE VKKEDISPVK VNELATSLEQ LGKLGEREKS RRAASDLKIR G HALSNDIV YGYGLQEKIQ KSAMATKETT VQSKRVSSRL HEVIVAKTRD YKIPTMPADA LHLYEFEVED VTVDLLPHAK HT SYSNLAY NMSFGSDGWF AFALLGGLDR SANLLRLDVA SIRGNYHKFS YDDPVFKQGY KIYKSDATLL NDFFVAISAG PKE QGILLR AFAYYSLYGN VEYHYVLSPR QLFFLSDNPV SAERLVRIPP SYYVSTQCRA LYNIFSYLHI LRSITSNQGK RLGM VLHPG LIAYVRGTSQ GAILPEADNV UniProtKB: RNA-directed RNA polymerase |
-分子 #2: Viral structural protein 4
| 分子 | 名称: Viral structural protein 4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bombyx mori cytoplasmic polyhedrosis virus (ウイルス) Bombyx mori cytoplasmic polyhedrosis virus (ウイルス) |
| 分子量 | 理論値: 63.683738 KDa |
| 配列 | 文字列: MFAIDPLKHS KLYEEYGLYL RPHQINQEIK PTTIKKKELA PTIRSIKYAS LIHSMLAKHA ARHNGTLINP RMYADMITLG NTKVTVTKG TPKAQIDTLK MNGLTVVSKS RRNNKKKPVS DTTATIDENT DDIVTYKALT EMSTLIESFR LPSGLALIIF D DEKYQSLI ...文字列: MFAIDPLKHS KLYEEYGLYL RPHQINQEIK PTTIKKKELA PTIRSIKYAS LIHSMLAKHA ARHNGTLINP RMYADMITLG NTKVTVTKG TPKAQIDTLK MNGLTVVSKS RRNNKKKPVS DTTATIDENT DDIVTYKALT EMSTLIESFR LPSGLALIIF D DEKYQSLI PNYINQLIAY TQPHIIPTWQ GIADFSDTYL RSYFKRPFEL TASNLAAPQK YNLSPMTRSI FNNTGREDAV IR KLYGYGE YVFIRYEGCL ITWTGIYGEV TMMVNLSKRD LGLDVGDDYL KEYKKLLFYG VITDAIPSGI SARSTIMKIS PHK MMNPSG GALAVLSKFL EAVVSTNVIN ATLVVYAEKG AGKTSFLSTY AEQLSLASGQ VVGHLSSDAY GRWLAKNKDV EEPS FAYDY VLSLDTDDNE SYYEQKASEL LISHGISEVA QYELLSVRKK IKMMDEMNEV LIAQLENADT HSERNFYYMV STGKT TPRT LIVEGHFNAQ DATIARTDTT VLLRTINDTT QAMRDRQRGG VVQLFLRDTY YRLLPALHTT VYPFEMLESI RRWKWV H UniProtKB: Viral structural protein 4 |
-分子 #3: P1-7-METHYLGUANOSINE-P3-ADENOSINE-5',5'-TRIPHOSPHATE
| 分子 | 名称: P1-7-METHYLGUANOSINE-P3-ADENOSINE-5',5'-TRIPHOSPHATE タイプ: ligand / ID: 3 / コピー数: 1 / 式: GTA |
|---|---|
| 分子量 | 理論値: 787.441 Da |
| Chemical component information | 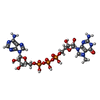 ChemComp-GTA: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















