+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2043 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram averaging reconstruction of the Ebola virus nucleocapsid | |||||||||
 マップデータ マップデータ | Reconstruction of the Ebola virus nucleocapsid | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ebola virus / nucleocapsid / subtomogram averaging | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 36.0 Å | |||||||||
 データ登録者 データ登録者 | Bharat TAM / Noda T / Riches JD / Kraehling V / Kolesnikova L / Becker S / Kawaoka Y / Briggs JAG | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2012 ジャーナル: Proc Natl Acad Sci U S A / 年: 2012タイトル: Structural dissection of Ebola virus and its assembly determinants using cryo-electron tomography. 著者: Tanmay A M Bharat / Takeshi Noda / James D Riches / Verena Kraehling / Larissa Kolesnikova / Stephan Becker / Yoshihiro Kawaoka / John A G Briggs /  要旨: Ebola virus is a highly pathogenic filovirus causing severe hemorrhagic fever with high mortality rates. It assembles heterogenous, filamentous, enveloped virus particles containing a negative-sense, ...Ebola virus is a highly pathogenic filovirus causing severe hemorrhagic fever with high mortality rates. It assembles heterogenous, filamentous, enveloped virus particles containing a negative-sense, single-stranded RNA genome packaged within a helical nucleocapsid (NC). We have used cryo-electron microscopy and tomography to visualize Ebola virus particles, as well as Ebola virus-like particles, in three dimensions in a near-native state. The NC within the virion forms a left-handed helix with an inner nucleoprotein layer decorated with protruding arms composed of VP24 and VP35. A comparison with the closely related Marburg virus shows that the N-terminal region of nucleoprotein defines the inner diameter of the Ebola virus NC, whereas the RNA genome defines its length. Binding of the nucleoprotein to RNA can assemble a loosely coiled NC-like structure; the loose coil can be condensed by binding of the viral matrix protein VP40 to the C terminus of the nucleoprotein, and rigidified by binding of VP24 and VP35 to alternate copies of the nucleoprotein. Four proteins (NP, VP24, VP35, and VP40) are necessary and sufficient to mediate assembly of an NC with structure, symmetry, variability, and flexibility indistinguishable from that in Ebola virus particles released from infected cells. Together these data provide a structural and architectural description of Ebola virus and define the roles of viral proteins in its structure and assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2043.map.gz emd_2043.map.gz | 612.1 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2043-v30.xml emd-2043-v30.xml emd-2043.xml emd-2043.xml | 9.4 KB 9.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  2043_emd_2043.jpg 2043_emd_2043.jpg emd_2043.jpg emd_2043.jpg | 73.2 KB 98.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2043 http://ftp.pdbj.org/pub/emdb/structures/EMD-2043 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2043 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2043 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2043_validation.pdf.gz emd_2043_validation.pdf.gz | 225 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2043_full_validation.pdf.gz emd_2043_full_validation.pdf.gz | 224.1 KB | 表示 | |
| XML形式データ |  emd_2043_validation.xml.gz emd_2043_validation.xml.gz | 4.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2043 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2043 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2043 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2043 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2043.map.gz / 形式: CCP4 / 大きさ: 646.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2043.map.gz / 形式: CCP4 / 大きさ: 646.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the Ebola virus nucleocapsid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 9.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Ebola virus nucleocapsid
| 全体 | 名称: Ebola virus nucleocapsid |
|---|---|
| 要素 |
|
-超分子 #1000: Ebola virus nucleocapsid
| 超分子 | 名称: Ebola virus nucleocapsid / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Zaire ebolavirus
| 超分子 | 名称: Zaire ebolavirus / タイプ: virus / ID: 1 / NCBI-ID: 186538 / 生物種: Zaire ebolavirus / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 詳細: PBS buffer with 4% paraformaldehyde |
|---|---|
| グリッド | 詳細: 300 mesh holey carbon C-Flat grid with 2micron holes. |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at high magnification. |
| 特殊光学系 | エネルギーフィルター - 名称: Gatan (GIF 2002) |
| 日付 | 2011年6月30日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC GATAN / 平均電子線量: 80 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): -6.0 µm / 最小 デフォーカス(公称値): -4.0 µm / 倍率(公称値): 27500 |
| 試料ステージ | 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -60 ° / Tilt series - Axis1 - Max angle: 60 ° |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Subtomogram averaging reconstruction was carried out without imposition of any symmetry. |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 6.3 Å 想定した対称性 - らせんパラメータ - ΔΦ: 30.5 ° アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 36.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: IMOD,AV3,Matlab |
 ムービー
ムービー コントローラー
コントローラー







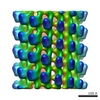





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















