+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20115 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of PilT4 from Geobacter metallireducens without adding nucleotide: C3ocococ conformation | |||||||||
 マップデータ マップデータ | PilT4 from Geobacter metallireducens | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ATPase / T4P / type iv pilus / motor / MOTOR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Geobacter metallireducens (バクテリア) / Geobacter metallireducens (バクテリア) /  Geobacter metallireducens (strain GS-15 / ATCC 53774 / DSM 7210) (バクテリア) Geobacter metallireducens (strain GS-15 / ATCC 53774 / DSM 7210) (バクテリア) | |||||||||
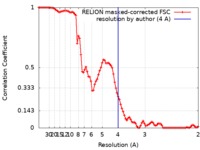
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | McCallum M / Howell PL | |||||||||
| 資金援助 |  カナダ, 1件 カナダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Multiple conformations facilitate PilT function in the type IV pilus. 著者: Matthew McCallum / Samir Benlekbir / Sheryl Nguyen / Stephanie Tammam / John L Rubinstein / Lori L Burrows / P Lynne Howell /  要旨: Type IV pilus-like systems are protein complexes that polymerize pilin fibres. They are critical for virulence in many bacterial pathogens. Pilin polymerization and depolymerization are powered by ...Type IV pilus-like systems are protein complexes that polymerize pilin fibres. They are critical for virulence in many bacterial pathogens. Pilin polymerization and depolymerization are powered by motor ATPases of the PilT/VirB11-like family. This family is thought to operate with C symmetry; however, most of these ATPases crystallize with either C or C symmetric conformations. The relevance of these conformations is unclear. Here, we determine the X-ray structures of PilT in four unique conformations and use these structures to classify the conformation of available PilT/VirB11-like family member structures. Single particle electron cryomicroscopy (cryoEM) structures of PilT reveal condition-dependent preferences for C C, and C conformations. The physiologic importance of these conformations is validated by coevolution analysis and functional studies of point mutants, identifying a rare gain-of-function mutation that favours the C conformation. With these data, we propose a comprehensive model of PilT function with broad implications for PilT/VirB11-like family members. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20115.map.gz emd_20115.map.gz | 3.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20115-v30.xml emd-20115-v30.xml emd-20115.xml emd-20115.xml | 13.2 KB 13.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20115_fsc.xml emd_20115_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20115.png emd_20115.png | 51.8 KB | ||
| Filedesc metadata |  emd-20115.cif.gz emd-20115.cif.gz | 5.5 KB | ||
| その他 |  emd_20115_additional.map.gz emd_20115_additional.map.gz | 31.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20115 http://ftp.pdbj.org/pub/emdb/structures/EMD-20115 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20115 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20115 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20115.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20115.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PilT4 from Geobacter metallireducens | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: PilT4 from Geobacter metallireducens
| ファイル | emd_20115_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PilT4 from Geobacter metallireducens | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PilT Hexamer
| 全体 | 名称: PilT Hexamer |
|---|---|
| 要素 |
|
-超分子 #1: PilT Hexamer
| 超分子 | 名称: PilT Hexamer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Geobacter metallireducens (バクテリア) Geobacter metallireducens (バクテリア) |
-分子 #1: Twitching motility pilus retraction ATPase
| 分子 | 名称: Twitching motility pilus retraction ATPase / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Geobacter metallireducens (strain GS-15 / ATCC 53774 / DSM 7210) (バクテリア) Geobacter metallireducens (strain GS-15 / ATCC 53774 / DSM 7210) (バクテリア)株: GS-15 / ATCC 53774 / DSM 7210 |
| 分子量 | 理論値: 42.883379 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MANMHQLLTE LVNRGGSDLH LTTNSPPQIR IDGKLLPLDM PPLNAVDTKQ LCYSILTEQQ KHKFEENNE LDLSFGIKGL SRFRGNVFVQ RGAVAGVFRV IPYKILSFEE LGLPPVVREL AEKPRGLVLV TGPTGSGKST T LAAIIDKI ...文字列: MGSSHHHHHH SSGLVPRGSH MANMHQLLTE LVNRGGSDLH LTTNSPPQIR IDGKLLPLDM PPLNAVDTKQ LCYSILTEQQ KHKFEENNE LDLSFGIKGL SRFRGNVFVQ RGAVAGVFRV IPYKILSFEE LGLPPVVREL AEKPRGLVLV TGPTGSGKST T LAAIIDKI NTDRHEHIVT VEDPIEYLHP HKSCVVNQRE VGADTKSFKN ALKYILRQDP DVVLVGELRD LETIEAALTL AE TGHLCFA TLHTNSAVQT INRIVDVFPS YQQPQVRAQL SFVLEGVLSQ TLLPKASGTG RVLAIEVMVP NPAIRNLIRE DKI HQIYSQ MQVGQEKFGM MTMNQCLYGL LQKRHITMDV GMGRSPDPDE LKQMLTSGVR PQAPRPPMR UniProtKB: Twitching motility pilus retraction ATPase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Homemade / 材質: GOLD |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 42.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Initial fitting in chimera, flexible fitting in Phenix-refine |
| 精密化 | プロトコル: FLEXIBLE FIT / 温度因子: 16.5 |
| 得られたモデル |  PDB-6olk: |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)