+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the human Tip60 complex | |||||||||
 マップデータ マップデータ | Composite map of the human Tip60 complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Eukaryotic transcription / Histone acetyltransferase / chromatin remodeling / Complex / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報piccolo histone acetyltransferase complex / promoter-enhancer loop anchoring activity / telomerase RNA localization to Cajal body / positive regulation of norepinephrine uptake / regulation of DNA strand elongation / positive regulation of telomere maintenance in response to DNA damage / sperm DNA condensation / histone chaperone activity / establishment of protein localization to chromatin / cellular response to cytochalasin B ...piccolo histone acetyltransferase complex / promoter-enhancer loop anchoring activity / telomerase RNA localization to Cajal body / positive regulation of norepinephrine uptake / regulation of DNA strand elongation / positive regulation of telomere maintenance in response to DNA damage / sperm DNA condensation / histone chaperone activity / establishment of protein localization to chromatin / cellular response to cytochalasin B / bBAF complex / R2TP complex / npBAF complex / nBAF complex / brahma complex / dynein axonemal particle / regulation of transepithelial transport / neural retina development / morphogenesis of a polarized epithelium / structural constituent of postsynaptic actin cytoskeleton / GBAF complex / Formation of annular gap junctions / Formation of the dystrophin-glycoprotein complex (DGC) / protein localization to adherens junction / Swr1 complex / Gap junction degradation / regulation of G0 to G1 transition / Folding of actin by CCT/TriC / dense body / Cell-extracellular matrix interactions / postsynaptic actin cytoskeleton / Tat protein binding / RPAP3/R2TP/prefoldin-like complex / protein antigen binding / chromatin-protein adaptor activity / Ino80 complex / Prefoldin mediated transfer of substrate to CCT/TriC / RSC-type complex / regulation of double-strand break repair / blastocyst formation / regulation of nucleotide-excision repair / adherens junction assembly / RHOF GTPase cycle / Adherens junctions interactions / apical protein localization / Sensory processing of sound by outer hair cells of the cochlea / box C/D snoRNP assembly / tight junction / Interaction between L1 and Ankyrins / protein folding chaperone complex / SWI/SNF complex / regulation of mitotic metaphase/anaphase transition / Sensory processing of sound by inner hair cells of the cochlea / positive regulation of T cell differentiation / regulation of norepinephrine uptake / apical junction complex / transporter regulator activity / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / positive regulation of double-strand break repair / spinal cord development / maintenance of blood-brain barrier / nitric-oxide synthase binding / negative regulation of gene expression, epigenetic / regulation of chromosome organization / establishment or maintenance of cell polarity / cortical cytoskeleton / NuA4 histone acetyltransferase complex / positive regulation of stem cell population maintenance / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / Transcriptional Regulation by E2F6 / Regulation of MITF-M-dependent genes involved in pigmentation / Recycling pathway of L1 / brush border / regulation of DNA replication / regulation of G1/S transition of mitotic cell cycle / TFIID-class transcription factor complex binding / MLL1 complex / regulation of embryonic development / kinesin binding / Telomere Extension By Telomerase / EPH-ephrin mediated repulsion of cells / negative regulation of cell differentiation / somatic stem cell population maintenance / spermatid development / regulation of synaptic vesicle endocytosis / RHO GTPases Activate WASPs and WAVEs / enzyme-substrate adaptor activity / positive regulation of myoblast differentiation / RHO GTPases activate IQGAPs / positive regulation of double-strand break repair via homologous recombination / regulation of protein localization to plasma membrane / RNA polymerase II core promoter sequence-specific DNA binding / regulation of DNA repair / cytoskeleton organization / EPHB-mediated forward signaling / Deposition of new CENPA-containing nucleosomes at the centromere / substantia nigra development / telomere maintenance / DNA helicase activity / axonogenesis 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.4 Å | |||||||||
 データ登録者 データ登録者 | Li C / Smirnova E / Schnitzler C / Crucifix C / Concordet JP / Brion A / Poterszman A / Schultz P / Papai G / Ben-Shem A | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Structure of the human TIP60-C histone exchange and acetyltransferase complex. 著者: Changqing Li / Ekaterina Smirnova / Charlotte Schnitzler / Corinne Crucifix / Jean Paul Concordet / Alice Brion / Arnaud Poterszman / Patrick Schultz / Gabor Papai / Adam Ben-Shem /  要旨: Chromatin structure is a key regulator of DNA transcription, replication and repair. In humans, the TIP60-EP400 complex (TIP60-C) is a 20-subunit assembly that affects chromatin structure through two ...Chromatin structure is a key regulator of DNA transcription, replication and repair. In humans, the TIP60-EP400 complex (TIP60-C) is a 20-subunit assembly that affects chromatin structure through two enzymatic activities: ATP-dependent exchange of histone H2A-H2B for H2A.Z-H2B, and histone acetylation. In yeast, however, these activities are performed by two independent complexes-SWR1 and NuA4, respectively. How the activities of the two complexes are merged into one supercomplex in humans, and what this association entails for the structure and mechanism of the proteins and their recruitment to chromatin, are unknown. Here we describe the structure of the endogenous human TIP60-C. We find a three-lobed architecture composed of SWR1-like (SWR1L) and NuA4-like (NuA4L) parts, which associate with a TRRAP activator-binding module. The huge EP400 subunit contains the ATPase motor, traverses the junction between SWR1L and NuA4L twice and constitutes the scaffold of the three-lobed architecture. NuA4L is completely rearranged compared with its yeast counterpart. TRRAP is flexibly tethered to NuA4L-in stark contrast to its robust connection to the completely opposite side of NuA4 in yeast. A modelled nucleosome bound to SWR1L, supported by tests of TIP60-C activity, suggests that some aspects of the histone exchange mechanism diverge from what is seen in yeast. Furthermore, a fixed actin module (as opposed to the mobile actin subcomplex in SWR1; ref. ), the flexibility of TRRAP and the weak effect of extranucleosomal DNA on exchange activity lead to a different, activator-based mode of enlisting TIP60-C to chromatin. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18611.map.gz emd_18611.map.gz | 584.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18611-v30.xml emd-18611-v30.xml emd-18611.xml emd-18611.xml | 25.9 KB 25.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18611.png emd_18611.png | 90.7 KB | ||
| Filedesc metadata |  emd-18611.cif.gz emd-18611.cif.gz | 9.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18611 http://ftp.pdbj.org/pub/emdb/structures/EMD-18611 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18611 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18611 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18611_validation.pdf.gz emd_18611_validation.pdf.gz | 508.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18611_full_validation.pdf.gz emd_18611_full_validation.pdf.gz | 507.9 KB | 表示 | |
| XML形式データ |  emd_18611_validation.xml.gz emd_18611_validation.xml.gz | 9.5 KB | 表示 | |
| CIF形式データ |  emd_18611_validation.cif.gz emd_18611_validation.cif.gz | 11.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18611 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18611 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18611 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18611 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qr1MC  8qriC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18611.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18611.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map of the human Tip60 complex | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.73 Å | ||||||||||||||||||||||||||||||||||||
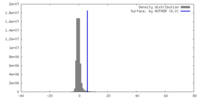
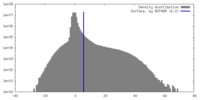
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Tip60 complex
| 全体 | 名称: Tip60 complex |
|---|---|
| 要素 |
|
-超分子 #1: Tip60 complex
| 超分子 | 名称: Tip60 complex / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 株: K562 Homo sapiens (ヒト) / 株: K562 |
-分子 #1: E1A-binding protein p400
| 分子 | 名称: E1A-binding protein p400 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 343.867312 KDa |
| 配列 | 文字列: MHHGTGPQNV QHQLQRSRAC PGSEGEEQPA HPNPPPSPAA PFAPSASPSA PQSPSYQIQQ LMNRSPATGQ NVNITLQSVG PVVGGNQQI TLAPLPLPSP TSPGFQFSAQ PRRFEHGSPS YIQVTSPLSQ QVQTQSPTQP SPGPGQALQN VRAGAPGPGL G LCSSSPTG ...文字列: MHHGTGPQNV QHQLQRSRAC PGSEGEEQPA HPNPPPSPAA PFAPSASPSA PQSPSYQIQQ LMNRSPATGQ NVNITLQSVG PVVGGNQQI TLAPLPLPSP TSPGFQFSAQ PRRFEHGSPS YIQVTSPLSQ QVQTQSPTQP SPGPGQALQN VRAGAPGPGL G LCSSSPTG GFVDASVLVR QISLSPSSGG HFVFQDGSGL TQIAQGAQVQ LQHPGTPITV RERRPSQPHT QSGGTIHHLG PQ SPAAAGG AGLQPLASPS HITTANLPPQ ISSIIQGQLV QQQQVLQGPP LPRPLGFERT PGVLLPGAGG AAGFGMTSPP PPT SPSRTA VPPGLSSLPL TSVGNTGMKK VPKKLEEIPP ASPEMAQMRK QCLDYHYQEM QALKEVFKEY LIELFFLQHF QGNM MDFLA FKKKHYAPLQ AYLRQNDLDI EEEEEEEEEE EEKSEVINDE VKVVTGKDGQ TGTPVAIATQ LPPKVSAAFS SQQQP FQQA LAGSLVAGAG STVETDLFKR QQAMPSTGMA EQSKRPRLEV GHQGVVFQHP GADAGVPLQQ LMPTAQGGMP PTPQAA QLA GQRQSQQQYD PSTGPPVQNA ASLHTPLPQL PGRLPPAGVP TAALSSALQF AQQPQVVEAQ TQLQIPVKTQ QPNVPIP AP PSSQLPIPPS QPAQLALHVP TPGKVQVQAS QLSSLPQMVA STRLPVDPAP PCPRPLPTSS TSSLAPVSGS GPGPSPAR S SPVNRPSSAT NKALSPVTSR TPGVVASAPT KPQSPAQNAT SSQDSSQDTL TEQITLENQV HQRIAELRKA GLWSQRRLP KLQEAPRPKS HWDYLLEEMQ WMATDFAQER RWKVAAAKKL VRTVVRHHEE KQLREERGKK EEQSRLRRIA ASTAREIECF WSNIEQVVE IKLRVELEEK RKKALNLQKV SRRGKELRPK GFDALQESSL DSGMSGRKRK ASISLTDDEV DDEEETIEEE E ANEGVVDH QTELSNLAKE AELPLLDLMK LYEGAFLPSS QWPRPKPDGE DTSGEEDADD CPGDRESRKD LVLIDSLFIM DQ FKAAERM NIGKPNAKDI ADVTAVAEAI LPKGSARVTT SVKFNAPSLL YGALRDYQKI GLDWLAKLYR KNLNGILADE AGL GKTVQI IAFFAHLACN EGNWGPHLVV VRSCNILKWE LELKRWCPGL KILSYIGSHR ELKAKRQEWA EPNSFHVCIT SYTQ FFRGL TAFTRVRWKC LVIDEMQRVK GMTERHWEAV FTLQSQQRLL LIDSPLHNTF LELWTMVHFL VPGISRPYLS SPLRA PSEE SQDYYHKVVI RLHRVTQPFI LRRTKRDVEK QLTKKYEHVL KCRLSNRQKA LYEDVILQPG TQEALKSGHF VNVLSI LVR LQRICNHPGL VEPRHPGSSY VAGPLEYPSA SLILKALERD FWKEADLSMF DLIGLENKIT RHEAELLSKK KIPRKLM EE ISTSAAPAAR PAAAKLKASR LFQPVQYGQK PEGRTVAFPS THPPRTAAPT TASAAPQGPL RGRPPIATFS ANPEAKAA A APFQTSQASA SAPRHQPASA SSTAASPAHP AKLRAQTTAQ ASTPGQPPPQ PQAPSHAAGQ SALPQRLVLP SQAQARLPS GEVVKIAQLA SITGPQSRVA QPETPVTLQF QGSKFTLSHS QLRQLTAGQP LQLQGSVLQI VSAPGQPYLR APGPVVMQTV SQAGAVHGA LGSKPPAGGP SPAPLTPQVG VPGRVAVNAL AVGEPGTASK PASPIGGPTQ EEKTRLLKER LDQIYLVNER R CSQAPVYG RDLLRICALP SHGRVQWRGS LDGRRGKEAG PAHSYTSSSE SPSELMLTLC RCGESLQDVI DRVAFVIPPV VA APPSLRV PRPPPLYSHR MRILRQGLRE HAAPYFQQLR QTTAPRLLQF PELRLVQFDS GKLEALAILL QKLKSEGRRV LIL SQMILM LDILEMFLNF HYLTYVRIDE NASSEQRQEL MRSFNRDRRI FCAILSTHSR TTGINLVEAD TVVFYDNDLN PVMD AKAQE WCDRIGRCKD IHIYRLVSGN SIEEKLLKNG TKDLIREVAA QGNDYSMAFL TQRTIQELFE VYSPMDDAGF PVKAE EFVV LSQEPSVTET IAPKIARPFI EALKSIEYLE EDAQKSAQEG VLGPHTDALS SDSENMPCDE EPSQLEELAD FMEQLT PIE KYALNYLELF HTSIEQEKER NSEDAVMTAV RAWEFWNLKT LQEREARLRL EQEEAELLTY TREDAYSMEY VYEDVDG QT EVMPLWTPPT PPQDDSDIYL DSVMCLMYEA TPIPEAKLPP VYVRKERKRH KTDPSAAGRK KKQRHGEAVV PPRSLFDR A TPGLLKIRRE GKEQKKNILL KQQVPFAKPL PTFAKPTAEP GQDNPEWLIS EDWALLQAVK QLLELPLNLT IVSPAHTPN WDLVSDVVNS CSRIYRSSKQ CRNRYENVII PREEGKSKNN RPLRTSQIYA QDENATHTQL YTSHFDLMKM TAGKRSPPIK PLLGMNPFQ KNPKHASVLA ESGINYDKPL PPIQVASLRA ERIAKEKKAL ADQQKAQQPA VAQPPPPQPQ PPPPPQQPPP P LPQPQAAG SQPPAGPPAV QPQPQPQPQT QPQPVQAPAK AQPAITTGGS AAVLAGTIKT SVTGTSMPTG AVSGNVIVNT IA GVPAATF QSINKRLASP VAPGALTTPG GSAPAQVVHT QPPPRAVGSP ATATPDLVSM ATTQGVRAVT SVTASAVVTT NLT PVQTPA RSLVPQVSQA TGVQLPGKTI TPAHFQLLRQ QQQQQQQQQQ QQQQQQQQQQ QQQQQQQQTT TTSQVQVPQI QGQA QSPAQ IKAVGKLTPE HLIKMQKQKL QMPPQPPPPQ AQSAPPQPTA QVQVQTSQPP QQQSPQLTTV TAPRPGALLT GTTVA NLQV ARLTRVPTSQ LQAQGQMQTQ APQPAQVALA KPPVVSVPAA VVSSPGVTTL PMNVAGISVA IGQPQKAAGQ TVVAQP VHM QQLLKLKQQA VQQQKAIQPQ AAQGPAAVQQ KITAQQITTP GAQQKVAYAA QPALKTQFLT TPISQAQKLA GAQQVQT QI QVAKLPQVVQ QQTPVASIQQ VASASQQASP QTVALTQATA AGQQVQMIPA VTATAQVVQQ KLIQQQVVTT ASAPLQTP G APNPAQVPAS SDSPSQQPKL QMRVPAVRLK TPTKPPCQ UniProtKB: E1A-binding protein p400 |
-分子 #2: Enhancer of polycomb homolog 1
| 分子 | 名称: Enhancer of polycomb homolog 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 93.589172 KDa |
| 配列 | 文字列: MSKLSFRARA LDASKPLPVF RCEDLPDLHE YASINRAVPQ MPTGMEKEEE SEHHLQRAIS AQQVYGEKRD NMVIPVPEAE SNIAYYESI YPGEFKMPKQ LIHIQPFSLD AEQPDYDLDS EDEVFVNKLK KKMDICPLQF EEMIDRLEKG SGQQPVSLQE A KLLLKEDD ...文字列: MSKLSFRARA LDASKPLPVF RCEDLPDLHE YASINRAVPQ MPTGMEKEEE SEHHLQRAIS AQQVYGEKRD NMVIPVPEAE SNIAYYESI YPGEFKMPKQ LIHIQPFSLD AEQPDYDLDS EDEVFVNKLK KKMDICPLQF EEMIDRLEKG SGQQPVSLQE A KLLLKEDD ELIREVYEYW IKKRKNCRGP SLIPSVKQEK RDGSSTNDPY VAFRRRTEKM QTRKNRKNDE ASYEKMLKLR RD LSRAVTI LEMIKRREKS KRELLHLTLE IMEKRYNLGD YNGEIMSEVM AQRQPMKPTY AIPIIPITNS SQFKHQEAMD VKE FKVNKQ DKADLIRPKR KYEKKPKVLP SSAAATPQQT SPAALPVFNA KDLNQYDFPS SDEEPLSQVL SGSSEAEEDN DPDG PFAFR RKAGCQYYAP HLDQTGNWPW TSPKDGGLGD VRYRYCLTTL TVPQRCIGFA RRRVGRGGRV LLDRAHSDYD SVFHH LDLE MLSSPQHSPV NQFANTSETN TSDKSFSKDL SQILVNIKSC RWRHFRPRTP SLHDSDNDEL SCRKLYRSIN RTGTAQ PGT QTCSTSTQSK SSSGSAHFAF TAEQYQQHQQ QLALMQKQQL AQIQQQQANS NSSTNTSQNL ASNQQKSGFR LNIQGLE RT LQGFVSKTLD SASAQFAASA LVTSEQLMGF KMKDDVVLGI GVNGVLPASG VYKGLHLSST TPTALVHTSP STAGSALL Q PSNITQTSSS HSALSHQVTA ANSATTQVLI GNNIRLTVPS SVATVNSIAP INARHIPRTL SAVPSSALKL AAAANCQVS KVPSSSSVDS VPRENHESEK PALNNIADNT VAMEVT UniProtKB: Enhancer of polycomb homolog 1 |
-分子 #3: DNA methyltransferase 1-associated protein 1
| 分子 | 名称: DNA methyltransferase 1-associated protein 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 53.090699 KDa |
| 配列 | 文字列: MATGADVRDI LELGGPEGDA ASGTISKKDI INPDKKKSKK SSETLTFKRP EGMHREVYAL LYSDKKDAPP LLPSDTGQGY RTVKAKLGS KKVRPWKWMP FTNPARKDGA MFFHWRRAAE EGKDYPFARF NKTVQVPVYS EQEYQLYLHD DAWTKAETDH L FDLSRRFD ...文字列: MATGADVRDI LELGGPEGDA ASGTISKKDI INPDKKKSKK SSETLTFKRP EGMHREVYAL LYSDKKDAPP LLPSDTGQGY RTVKAKLGS KKVRPWKWMP FTNPARKDGA MFFHWRRAAE EGKDYPFARF NKTVQVPVYS EQEYQLYLHD DAWTKAETDH L FDLSRRFD LRFVVIHDRY DHQQFKKRSV EDLKERYYHI CAKLANVRAV PGTDLKIPVF DAGHERRRKE QLERLYNRTP EQ VAEEEYL LQELRKIEAR KKEREKRSQD LQKLITAADT TAEQRRTERK APKKKLPQKK EAEKPAVPET AGIKFPDFKS AGV TLRSQR MKLPSSVGQK KIKALEQMLL ELGVELSPTP TEELVHMFNE LRSDLVLLYE LKQACANCEY ELQMLRHRHE ALAR AGVLG GPATPASGPG PASAEPAVTE PGLGPDPKDT IIDVVGAPLT PNSRKRRESA SSSSSVKKAK KP UniProtKB: DNA methyltransferase 1-associated protein 1 |
-分子 #4: Vacuolar protein sorting-associated protein 72 homolog
| 分子 | 名称: Vacuolar protein sorting-associated protein 72 homolog タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 40.658363 KDa |
| 配列 | 文字列: MSLAGGRAPR KTAGNRLSGL LEAEEEDEFY QTTYGGFTEE SGDDEYQGDQ SDTEDEVDSD FDIDEGDEPS SDGEAEEPRR KRRVVTKAY KEPLKSLRPR KVNTPAGSSQ KAREEKALLP LELQDDGSDS RKSMRQSTAE HTRQTFLRVQ ERQGQSRRRK G PHCERPLT ...文字列: MSLAGGRAPR KTAGNRLSGL LEAEEEDEFY QTTYGGFTEE SGDDEYQGDQ SDTEDEVDSD FDIDEGDEPS SDGEAEEPRR KRRVVTKAY KEPLKSLRPR KVNTPAGSSQ KAREEKALLP LELQDDGSDS RKSMRQSTAE HTRQTFLRVQ ERQGQSRRRK G PHCERPLT QEELLREAKI TEELNLRSLE TYERLEADKK KQVHKKRKCP GPIITYHSVT VPLVGEPGPK EENVDIEGLD PA PSVSALT PHAGTGPVNP PARCSRTFIT FSDDATFEEW FPQGRPPKVP VREVCPVTHR PALYRDPVTD IPYATARAFK IIR EAYKKY ITAHGLPPTA SALGPGPPPP EPLPGSGPRA LRQKIVIK UniProtKB: Vacuolar protein sorting-associated protein 72 homolog |
-分子 #5: Actin, cytoplasmic 1, N-terminally processed
| 分子 | 名称: Actin, cytoplasmic 1, N-terminally processed / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 41.78266 KDa |
| 配列 | 文字列: MDDDIAALVV DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IEHGIVTNWD DMEKIWHHT FYNELRVAPE EHPVLLTEAP LNPKANREKM TQIMFETFNT PAMYVAIQAV LSLYASGRTT GIVMDSGDGV T HTVPIYEG ...文字列: MDDDIAALVV DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IEHGIVTNWD DMEKIWHHT FYNELRVAPE EHPVLLTEAP LNPKANREKM TQIMFETFNT PAMYVAIQAV LSLYASGRTT GIVMDSGDGV T HTVPIYEG YALPHAILRL DLAGRDLTDY LMKILTERGY SFTTTAEREI VRDIKEKLCY VALDFEQEMA TAASSSSLEK SY ELPDGQV ITIGNERFRC PEALFQPSFL GMESCGIHET TFNSIMKCDV DIRKDLYANT VLSGGTTMYP GIADRMQKEI TAL APSTMK IKIIAPPERK YSVWIGGSIL ASLSTFQQMW ISKQEYDESG PSIVHRKCF UniProtKB: Actin, cytoplasmic 1 |
-分子 #6: Actin-like protein 6A
| 分子 | 名称: Actin-like protein 6A / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 47.509812 KDa |
| 配列 | 文字列: MSGGVYGGDE VGALVFDIGS YTVRAGYAGE DCPKVDFPTA IGMVVERDDG STLMEIDGDK GKQGGPTYYI DTNALRVPRE NMEAISPLK NGMVEDWDSF QAILDHTYKM HVKSEASLHP VLMSEAPWNT RAKREKLTEL MFEHYNIPAF FLCKTAVLTA F ANGRSTGL ...文字列: MSGGVYGGDE VGALVFDIGS YTVRAGYAGE DCPKVDFPTA IGMVVERDDG STLMEIDGDK GKQGGPTYYI DTNALRVPRE NMEAISPLK NGMVEDWDSF QAILDHTYKM HVKSEASLHP VLMSEAPWNT RAKREKLTEL MFEHYNIPAF FLCKTAVLTA F ANGRSTGL ILDSGATHTT AIPVHDGYVL QQGIVKSPLA GDFITMQCRE LFQEMNIELV PPYMIASKEA VREGSPANWK RK EKLPQVT RSWHNYMCNC VIQDFQASVL QVSDSTYDEQ VAAQMPTVHY EFPNGYNCDF GAERLKIPEG LFDPSNVKGL SGN TMLGVS HVVTTSVGMC DIDIRPGLYG SVIVAGGNTL IQSFTDRLNR ELSQKTPPSM RLKLIANNTT VERRFSSWIG GSIL ASLGT FQQMWISKQE YEEGGKQCVE RKCP UniProtKB: Actin-like protein 6A |
-分子 #7: RuvB-like 1
| 分子 | 名称: RuvB-like 1 / タイプ: protein_or_peptide / ID: 7 / コピー数: 3 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 50.296914 KDa |
| 配列 | 文字列: MKIEEVKSTT KTQRIASHSH VKGLGLDESG LAKQAASGLV GQENAREACG VIVELIKSKK MAGRAVLLAG PPGTGKTALA LAIAQELGS KVPFCPMVGS EVYSTEIKKT EVLMENFRRA IGLRIKETKE VYEGEVTELT PCETENPMGG YGKTISHVII G LKTAKGTK ...文字列: MKIEEVKSTT KTQRIASHSH VKGLGLDESG LAKQAASGLV GQENAREACG VIVELIKSKK MAGRAVLLAG PPGTGKTALA LAIAQELGS KVPFCPMVGS EVYSTEIKKT EVLMENFRRA IGLRIKETKE VYEGEVTELT PCETENPMGG YGKTISHVII G LKTAKGTK QLKLDPSIFE SLQKERVEAG DVIYIEANSG AVKRQGRCDT YATEFDLEAE EYVPLPKGDV HKKKEIIQDV TL HDLDVAN ARPQGGQDIL SMMGQLMKPK KTEITDKLRG EINKVVNKYI DQGIAELVPG VLFVDEVHML DIECFTYLHR ALE SSIAPI VIFASNRGNC VIRGTEDITS PHGIPLDLLD RVMIIRTMLY TPQEMKQIIK IRAQTEGINI SEEALNHLGE IGTK TTLRY SVQLLTPANL LAKINGKDSI EKEHVEEISE LFYDAKSSAK ILADQQDKYM K UniProtKB: RuvB-like 1 |
-分子 #8: RuvB-like 2
| 分子 | 名称: RuvB-like 2 / タイプ: protein_or_peptide / ID: 8 / コピー数: 3 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 51.222465 KDa |
| 配列 | 文字列: MATVTATTKV PEIRDVTRIE RIGAHSHIRG LGLDDALEPR QASQGMVGQL AARRAAGVVL EMIREGKIAG RAVLIAGQPG TGKTAIAMG MAQALGPDTP FTAIAGSEIF SLEMSKTEAL TQAFRRSIGV RIKEETEIIE GEVVEIQIDR PATGTGSKVG K LTLKTTEM ...文字列: MATVTATTKV PEIRDVTRIE RIGAHSHIRG LGLDDALEPR QASQGMVGQL AARRAAGVVL EMIREGKIAG RAVLIAGQPG TGKTAIAMG MAQALGPDTP FTAIAGSEIF SLEMSKTEAL TQAFRRSIGV RIKEETEIIE GEVVEIQIDR PATGTGSKVG K LTLKTTEM ETIYDLGTKM IESLTKDKVQ AGDVITIDKA TGKISKLGRS FTRARDYDAM GSQTKFVQCP DGELQKRKEV VH TVSLHEI DVINSRTQGF LALFSGDTGE IKSEVREQIN AKVAEWREEG KAEIIPGVLF IDEVHMLDIE SFSFLNRALE SDM APVLIM ATNRGITRIR GTSYQSPHGI PIDLLDRLLI VSTTPYSEKD TKQILRIRCE EEDVEMSEDA YTVLTRIGLE TSLR YAIQL ITAASLVCRK RKGTEVQVDD IKRVYSLFLD ESRSTQYMKE YQDAFLFNEL KGETMDTS UniProtKB: RuvB-like 2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) #0 - 平均電子線量: 40.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) #1 - 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8qr1: |
 ムービー
ムービー コントローラー
コントローラー









































 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)