+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Vipp1-F197K/L200K helical filament with lattice 1 (Vipp1-F197K/L200K_L1) | |||||||||
 マップデータ マップデータ | Primary sharpened map. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Vipp1/IM30/ESCRT-III / Membrane remodeling / Cryoelectron microscopy / Helical filament structure / LIPID BINDING PROTEIN | |||||||||
| 機能・相同性 | PspA/IM30 / PspA/IM30 family / lipid binding / plasma membrane / Membrane-associated protein Vipp1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Nostoc punctiforme (バクテリア) Nostoc punctiforme (バクテリア) | |||||||||
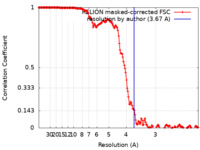
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.67 Å | |||||||||
 データ登録者 データ登録者 | Naskar S / Low HH | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2025 ジャーナル: Nat Struct Mol Biol / 年: 2025タイトル: Mechanism for Vipp1 spiral formation, ring biogenesis, and membrane repair. 著者: Souvik Naskar / Andrea Merino / Javier Espadas / Jayanti Singh / Aurelien Roux / Adai Colom / Harry H Low /    要旨: The ESCRT-III-like protein Vipp1 couples filament polymerization with membrane remodeling. It assembles planar sheets as well as 3D rings and helical polymers, all implicated in mitigating plastid- ...The ESCRT-III-like protein Vipp1 couples filament polymerization with membrane remodeling. It assembles planar sheets as well as 3D rings and helical polymers, all implicated in mitigating plastid-associated membrane stress. The architecture of Vipp1 planar sheets and helical polymers remains unknown, as do the geometric changes required to transition between polymeric forms. Here we show how cyanobacterial Vipp1 assembles into morphologically-related sheets and spirals on membranes in vitro. The spirals converge to form a central ring similar to those described in membrane budding. Cryo-EM structures of helical filaments reveal a close geometric relationship between Vipp1 helical and planar lattices. Moreover, the helical structures reveal how filaments twist-a process required for Vipp1, and likely other ESCRT-III filaments, to transition between planar and 3D architectures. Overall, our results provide a molecular model for Vipp1 ring biogenesis and a mechanism for Vipp1 membrane stabilization and repair, with implications for other ESCRT-III systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18319.map.gz emd_18319.map.gz | 315.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18319-v30.xml emd-18319-v30.xml emd-18319.xml emd-18319.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18319_fsc.xml emd_18319_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18319.png emd_18319.png | 127.5 KB | ||
| Filedesc metadata |  emd-18319.cif.gz emd-18319.cif.gz | 6 KB | ||
| その他 |  emd_18319_additional_1.map.gz emd_18319_additional_1.map.gz emd_18319_half_map_1.map.gz emd_18319_half_map_1.map.gz emd_18319_half_map_2.map.gz emd_18319_half_map_2.map.gz | 271.8 MB 275.8 MB 275.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18319 http://ftp.pdbj.org/pub/emdb/structures/EMD-18319 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18319 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18319 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18319_validation.pdf.gz emd_18319_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18319_full_validation.pdf.gz emd_18319_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_18319_validation.xml.gz emd_18319_validation.xml.gz | 22.8 KB | 表示 | |
| CIF形式データ |  emd_18319_validation.cif.gz emd_18319_validation.cif.gz | 30.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18319 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18319 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18319 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18319 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18319.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18319.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary sharpened map. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
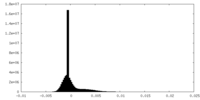
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened and unmasked raw map from 3D auto-refinement.
| ファイル | emd_18319_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened and unmasked raw map from 3D auto-refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
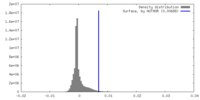
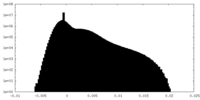
| 密度ヒストグラム |
-ハーフマップ: Unmasked raw half-map 1.
| ファイル | emd_18319_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked raw half-map 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
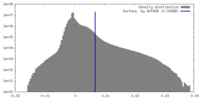
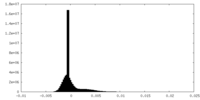
| 密度ヒストグラム |
-ハーフマップ: Unmasked raw half-map 2.
| ファイル | emd_18319_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked raw half-map 2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
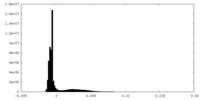
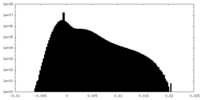
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vipp1-F197K/L200K_L1
| 全体 | 名称: Vipp1-F197K/L200K_L1 |
|---|---|
| 要素 |
|
-超分子 #1: Vipp1-F197K/L200K_L1
| 超分子 | 名称: Vipp1-F197K/L200K_L1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Vipp1 mutated at F197K and L200K, with lattice 1 (L1) helical feature. |
|---|---|
| 由来(天然) | 生物種:  Nostoc punctiforme (バクテリア) Nostoc punctiforme (バクテリア) |
-分子 #1: Phage shock protein A, PspA
| 分子 | 名称: Phage shock protein A, PspA / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Nostoc punctiforme (バクテリア) Nostoc punctiforme (バクテリア) |
| 分子量 | 理論値: 28.745461 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGLFDRIKRV VSSNLNDLVN KAEDPEKMLE QAILEMQEDL VQLRQGVAQA IAAQKRSEKQ YNDAQNEINK WQRNAQLALQ KGDENLARQ ALERKKTYTD TSAALKASLD TQSTQVETLK RNLIQLESKI SEAKTKKEML KARITTAKAQ EQLQGMVRGM N TSSAMSAF ...文字列: MGLFDRIKRV VSSNLNDLVN KAEDPEKMLE QAILEMQEDL VQLRQGVAQA IAAQKRSEKQ YNDAQNEINK WQRNAQLALQ KGDENLARQ ALERKKTYTD TSAALKASLD TQSTQVETLK RNLIQLESKI SEAKTKKEML KARITTAKAQ EQLQGMVRGM N TSSAMSAF ERMEEKVLMQ ESRAQALGEL AGADLETQFA QLEGGSDVDD ELAALKAQML PPATPVTQAQ LPPQQETTPA KS NEVVDAE LDSLRKQLDQ L UniProtKB: Membrane-associated protein Vipp1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1.7 mg/mL |
|---|---|
| 緩衝液 | pH: 8.4 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 50 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 4.6 sec. / 平均電子線量: 1.15 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.75 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / 温度因子: 73.33 |
|---|---|
| 得られたモデル |  PDB-8qbs: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)