+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1800 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle cryo-electron microscopy analysis of CD4 bound HIV-1 Env | |||||||||
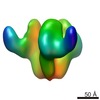
 マップデータ マップデータ | This is an image of a surface rendered CD4 bound HIV-1 Env | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV-1 spike / Membrane Fusion / Structural Transition / Cryo-EM / Single Particle | |||||||||
| 機能・相同性 | : / T-cell surface antigen CD4 / Human immunodeficiency virus 1, envelope glycoprotein Gp120 / viral envelope 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 21.0 Å | |||||||||
 データ登録者 データ登録者 | Wu SR / Loving R / Lindqvist B / Hebert H / Koeck P / Sjoberg M / Garoff H | |||||||||
 引用 引用 | ジャーナル: AIDS Res Hum Retroviruses / 年: 1990 タイトル: Escherichia coli expression, purification, and biological activity of a truncated soluble CD4. 著者: R L Garlick / R J Kirschner / F M Eckenrode / W G Tarpley / C S Tomich /  要旨: A truncated molecule containing the N-terminal 183 amino acid residues of CD4 (sCD4-183) has been produced in Escherichia coli at high levels, using the trp promoter and an AT-rich ribosome binding ...A truncated molecule containing the N-terminal 183 amino acid residues of CD4 (sCD4-183) has been produced in Escherichia coli at high levels, using the trp promoter and an AT-rich ribosome binding site to direct expression in a pBR322-derived vector. A culture has been selected which allows large-scale fermentation and production of this material as an insoluble inclusion body protein. Procedures which solubilize, refold, and purify sCD4-183 have been developed. The purified sCD4-183 binds gp120 in solution and blocks human immunodeficiency virus (HIV) infection of human peripheral blood lymphocytes in vitro. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1800.map.gz emd_1800.map.gz | 956.3 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1800-v30.xml emd-1800-v30.xml emd-1800.xml emd-1800.xml | 12.7 KB 12.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD_1800.jpg EMD_1800.jpg | 192.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1800 http://ftp.pdbj.org/pub/emdb/structures/EMD-1800 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1800 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1800 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1800_validation.pdf.gz emd_1800_validation.pdf.gz | 206.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1800_full_validation.pdf.gz emd_1800_full_validation.pdf.gz | 205.3 KB | 表示 | |
| XML形式データ |  emd_1800_validation.xml.gz emd_1800_validation.xml.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1800 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1800 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1800 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1800 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1800.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1800.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is an image of a surface rendered CD4 bound HIV-1 Env | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
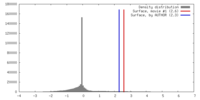
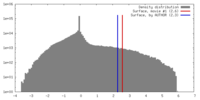
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CD4 bound HIV-1 Env
| 全体 | 名称: CD4 bound HIV-1 Env |
|---|---|
| 要素 |
|
-超分子 #1000: CD4 bound HIV-1 Env
| 超分子 | 名称: CD4 bound HIV-1 Env / タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse / 集合状態: Trimer / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 700 KDa / 理論値: 500 KDa / 手法: Blue Native PAGE |
-分子 #1: gp160deltaCTSOS
| 分子 | 名称: gp160deltaCTSOS / タイプ: protein_or_peptide / ID: 1 / Name.synonym: HIV-1 Env / コピー数: 3 / 集合状態: Trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)株: JR-FL / 別称: HIV-1 |
| 分子量 | 理論値: 420 KDa |
| 組換発現 | 生物種: 293T / 組換プラスミド: pCAGGS JRFL 160deltaCTSOS |
| 配列 | GO: viral envelope InterPro: Human immunodeficiency virus 1, envelope glycoprotein Gp120 |
-分子 #2: soluble CD4 (2 domain)
| 分子 | 名称: soluble CD4 (2 domain) / タイプ: protein_or_peptide / ID: 2 / Name.synonym: sCD4-2d 詳細: sCD4-183 was obtained through the AIDS Research and Reference Reagent Program, Division of AIDS, NIAID, NIH コピー数: 1 / 集合状態: Monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 26 KDa |
| 配列 | GO: GO: 0006948 / InterPro: T-cell surface antigen CD4 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 50 mM HEPES, 100 mM NaCl, 1.8 mM CaCl2 |
|---|---|
| 染色 | タイプ: NEGATIVE / 詳細: No stain was applied |
| グリッド | 詳細: 200 mesh Cu Holey carbon grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 99 % / チャンバー内温度: 77 K / 装置: OTHER / 詳細: Vitrification instrument: Vitrobot / 手法: Blot for 3 seconds once before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2100F |
|---|---|
| 温度 | 最低: 93 K / 最高: 96 K / 平均: 95 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected using online FFT Legacy - Electron beam tilt params: No Tilt |
| 日付 | 2009年10月23日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC CCD / デジタル化 - サンプリング間隔: 3.5 µm / 実像数: 336 / 平均電子線量: 9 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 43300 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | The 3D structures reconstructed by processing particle images using standard single particle procedure in EMAN version 1.9. |
|---|---|
| CTF補正 | 詳細: Each Digitized Image |
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 21.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN / 使用した粒子像数: 9870 |
| 最終 2次元分類 | クラス数: 131 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: O |
| 詳細 | PDBEntryID_givenInChain. Protocol: Rigid Body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)