+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a pre-dimerized murine IL-12 complete extracellular signaling complex (Class 1), obtained after local refinement. | |||||||||
 マップデータ マップデータ | Main map: Sharpened map of the murine IL12:IL12Rbeta1-DAPK1:IL12Rbeta2-Calmodulin complex (Class 1, local refinement) used for model refinement in Phenix. Additional map: deepEMhancer sharpened map. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / Cytokine / Receptor / SIGNALING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報interleukin-12 beta subunit binding / Interleukin-12 signaling / Interleukin-23 signaling / interleukin-23 receptor binding / Interleukin-35 Signalling / interleukin-12 alpha subunit binding / interleukin-12 complex / interleukin-23 complex / T-helper 1 cell activation / negative regulation of vascular endothelial growth factor signaling pathway ...interleukin-12 beta subunit binding / Interleukin-12 signaling / Interleukin-23 signaling / interleukin-23 receptor binding / Interleukin-35 Signalling / interleukin-12 alpha subunit binding / interleukin-12 complex / interleukin-23 complex / T-helper 1 cell activation / negative regulation of vascular endothelial growth factor signaling pathway / positive regulation of natural killer cell mediated cytotoxicity directed against tumor cell target / negative regulation of blood vessel endothelial cell proliferation involved in sprouting angiogenesis / cellular response to hydroperoxide / positive regulation of smooth muscle cell apoptotic process / regulation of response to tumor cell / positive regulation of autophagic cell death / DAPK1-calmodulin complex / positive regulation of T-helper 1 type immune response / interleukin-12 receptor binding / T-helper cell differentiation / interleukin-23 receptor complex / Caspase activation via Dependence Receptors in the absence of ligand / interleukin-12-mediated signaling pathway / defense response to tumor cell / positive regulation of NK T cell proliferation / positive regulation of osteoclast differentiation / regulation of NMDA receptor activity / negative regulation of interleukin-17 production / calcium/calmodulin-dependent protein kinase activity / cytokine receptor activity / natural killer cell activation / response to UV-B / positive regulation of granulocyte macrophage colony-stimulating factor production / CaM pathway / Cam-PDE 1 activation / Sodium/Calcium exchangers / Calmodulin induced events / syntaxin-1 binding / Reduction of cytosolic Ca++ levels / Activation of Ca-permeable Kainate Receptor / positive regulation of T cell differentiation / CREB1 phosphorylation through the activation of CaMKII/CaMKK/CaMKIV cascasde / Loss of phosphorylation of MECP2 at T308 / CREB1 phosphorylation through the activation of Adenylate Cyclase / CaMK IV-mediated phosphorylation of CREB / PKA activation / negative regulation of high voltage-gated calcium channel activity / Glycogen breakdown (glycogenolysis) / CLEC7A (Dectin-1) induces NFAT activation / Activation of RAC1 downstream of NMDARs / negative regulation of ryanodine-sensitive calcium-release channel activity / organelle localization by membrane tethering / negative regulation of interleukin-10 production / mitochondrion-endoplasmic reticulum membrane tethering / autophagosome membrane docking / negative regulation of calcium ion export across plasma membrane / regulation of cardiac muscle cell action potential / defense response to protozoan / presynaptic endocytosis / Synthesis of IP3 and IP4 in the cytosol / regulation of cell communication by electrical coupling involved in cardiac conduction / Phase 0 - rapid depolarisation / calcineurin-mediated signaling / Negative regulation of NMDA receptor-mediated neuronal transmission / Unblocking of NMDA receptors, glutamate binding and activation / positive regulation of interleukin-17 production / RHO GTPases activate PAKs / positive regulation of natural killer cell proliferation / positive regulation of activated T cell proliferation / Ion transport by P-type ATPases / Uptake and function of anthrax toxins / regulation of ryanodine-sensitive calcium-release channel activity / positive regulation of interleukin-10 production / Long-term potentiation / protein phosphatase activator activity / Calcineurin activates NFAT / Regulation of MECP2 expression and activity / extrinsic apoptotic signaling pathway via death domain receptors / DARPP-32 events / catalytic complex / Smooth Muscle Contraction / detection of calcium ion / negative regulation of protein secretion / regulation of cardiac muscle contraction / cell surface receptor signaling pathway via JAK-STAT / RHO GTPases activate IQGAPs / T cell proliferation / regulation of cardiac muscle contraction by regulation of the release of sequestered calcium ion / cellular response to interferon-beta / Protein methylation / calcium channel inhibitor activity / coreceptor activity / presynaptic cytosol / Activation of AMPK downstream of NMDARs / Ion homeostasis / regulation of release of sequestered calcium ion into cytosol by sarcoplasmic reticulum / eNOS activation / titin binding / Tetrahydrobiopterin (BH4) synthesis, recycling, salvage and regulation / sperm midpiece 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
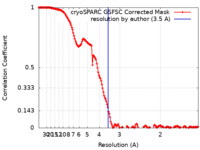
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Felix J / Bloch Y / Savvides SN | |||||||||
| 資金援助 |  ベルギー, 2件 ベルギー, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structures of complete extracellular receptor assemblies mediated by IL-12 and IL-23. 著者: Yehudi Bloch / Jan Felix / Romain Merceron / Mathias Provost / Royan Alipour Symakani / Robin De Backer / Elisabeth Lambert / Ahmad R Mehdipour / Savvas N Savvides /     要旨: Cell-surface receptor complexes mediated by pro-inflammatory interleukin (IL)-12 and IL-23, both validated therapeutic targets, are incompletely understood due to the lack of structural insights into ...Cell-surface receptor complexes mediated by pro-inflammatory interleukin (IL)-12 and IL-23, both validated therapeutic targets, are incompletely understood due to the lack of structural insights into their complete extracellular assemblies. Furthermore, there is a paucity of structural details describing the IL-12-receptor interaction interfaces, in contrast to IL-23-receptor complexes. Here we report structures of fully assembled mouse IL-12/human IL-23-receptor complexes comprising the complete extracellular segments of the cognate receptors determined by electron cryo-microscopy. The structures reveal key commonalities but also surprisingly diverse features. Most notably, whereas IL-12 and IL-23 both utilize a conspicuously presented aromatic residue on their α-subunit as a hotspot to interact with the N-terminal Ig domain of their high-affinity receptors, only IL-12 juxtaposes receptor domains proximal to the cell membrane. Collectively, our findings will help to complete our understanding of cytokine-mediated assemblies of tall cytokine receptors and will enable a cytokine-specific interrogation of IL-12/IL-23 signaling in physiology and disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17580.map.gz emd_17580.map.gz | 166 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17580-v30.xml emd-17580-v30.xml emd-17580.xml emd-17580.xml | 26.1 KB 26.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17580_fsc.xml emd_17580_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17580.png emd_17580.png | 68.5 KB | ||
| マスクデータ |  emd_17580_msk_1.map emd_17580_msk_1.map emd_17580_msk_2.map emd_17580_msk_2.map | 325 MB 325 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17580.cif.gz emd-17580.cif.gz | 8 KB | ||
| その他 |  emd_17580_additional_1.map.gz emd_17580_additional_1.map.gz emd_17580_half_map_1.map.gz emd_17580_half_map_1.map.gz emd_17580_half_map_2.map.gz emd_17580_half_map_2.map.gz | 268.8 MB 301.3 MB 301.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17580 http://ftp.pdbj.org/pub/emdb/structures/EMD-17580 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17580 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17580 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17580_validation.pdf.gz emd_17580_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17580_full_validation.pdf.gz emd_17580_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_17580_validation.xml.gz emd_17580_validation.xml.gz | 23 KB | 表示 | |
| CIF形式データ |  emd_17580_validation.cif.gz emd_17580_validation.cif.gz | 29.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17580 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17580 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17580 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17580 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pb1MC  8c7mC  8cr5C  8cr6C  8cr8C  8odxC  8odzC  8oe0C  8oe4C  8ppmC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17580.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17580.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map: Sharpened map of the murine IL12:IL12Rbeta1-DAPK1:IL12Rbeta2-Calmodulin complex (Class 1, local refinement) used for model refinement in Phenix. Additional map: deepEMhancer sharpened map. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.755 Å | ||||||||||||||||||||||||||||||||||||
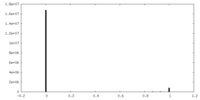
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17580_msk_1.map emd_17580_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
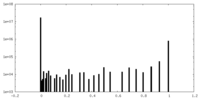
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_17580_msk_2.map emd_17580_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: DeepEMhancer sharpened map of the murine IL12:IL12Rbeta1-DAPK1:IL12Rbeta2-Calmodulin complex...
| ファイル | emd_17580_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer sharpened map of the murine IL12:IL12Rbeta1-DAPK1:IL12Rbeta2-Calmodulin complex (Class 1, local refinement) used for model building in Coot and visualization. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
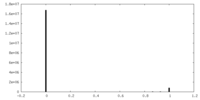
| 密度ヒストグラム |
-ハーフマップ: Half map 1 of the murine IL12:IL12Rbeta1-DAPK1:IL12Rbeta2-Calmodulin complex...
| ファイル | emd_17580_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 of the murine IL12:IL12Rbeta1-DAPK1:IL12Rbeta2-Calmodulin complex (Class 1), obtained after local refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
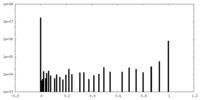
| 密度ヒストグラム |
-ハーフマップ: Half map 2 of the murine IL12:IL12Rbeta1-DAPK1:IL12Rbeta2-Calmodulin complex...
| ファイル | emd_17580_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 of the murine IL12:IL12Rbeta1-DAPK1:IL12Rbeta2-Calmodulin complex (Class 1), obtained after local refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Murine IL-12 in complex with mIL-12Rbeta1-DAPK1 and mIL-12Rbeta2-...
| 全体 | 名称: Murine IL-12 in complex with mIL-12Rbeta1-DAPK1 and mIL-12Rbeta2-Calmodulin. |
|---|---|
| 要素 |
|
-超分子 #1: Murine IL-12 in complex with mIL-12Rbeta1-DAPK1 and mIL-12Rbeta2-...
| 超分子 | 名称: Murine IL-12 in complex with mIL-12Rbeta1-DAPK1 and mIL-12Rbeta2-Calmodulin. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 216 KDa |
-分子 #1: Interleukin-12 subunit alpha
| 分子 | 名称: Interleukin-12 subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.927496 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: RVIPVSGPAR CLSQSRNLLK TTDDMVKTAR EKLKHYSCTA EDIDHEDITR DQTSTLKTCL PLELHKNESC LATRETSSTT RGSCLPPQK TSLMMTLCLG SIYEDLKMYQ TEFQAINAAL QNHNHQQIIL DKGMLVAIDE LMQSLNHNGE TLRQKPPVGE A DPYRVKMK ...文字列: RVIPVSGPAR CLSQSRNLLK TTDDMVKTAR EKLKHYSCTA EDIDHEDITR DQTSTLKTCL PLELHKNESC LATRETSSTT RGSCLPPQK TSLMMTLCLG SIYEDLKMYQ TEFQAINAAL QNHNHQQIIL DKGMLVAIDE LMQSLNHNGE TLRQKPPVGE A DPYRVKMK LCILLHAFST RVVTINRVMG YLSSAGTSDE VDGGSGGSGL NDIFEAQKIE WHEGRTKHHH HHH UniProtKB: Interleukin-12 subunit alpha |
-分子 #2: Interleukin-12 subunit beta
| 分子 | 名称: Interleukin-12 subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 35.83732 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MWELEKDVYV VEVDWTPDAP GETVNLTCDT PEEDDITWTS DQRHGVIGSG KTLTITVKEF LDAGQYTCHK GGETLSHSHL LLHKKENGI WSTEILKNFK NKTFLKCEAP NYSGRFTCSW LVQRNMDLKF NIKSSSSSPD SRAVTCGMAS LSAEKVTLDQ R DYEKYSVS ...文字列: MWELEKDVYV VEVDWTPDAP GETVNLTCDT PEEDDITWTS DQRHGVIGSG KTLTITVKEF LDAGQYTCHK GGETLSHSHL LLHKKENGI WSTEILKNFK NKTFLKCEAP NYSGRFTCSW LVQRNMDLKF NIKSSSSSPD SRAVTCGMAS LSAEKVTLDQ R DYEKYSVS CQEDVTCPTA EETLPIELAL EARQQNKYEN YSTSFFIRDI IKPDPPKNLQ MKPLKNSQVE VSWEYPDSWS TP HSYFSLK FFVRIQRKKE KMKETEEGCN QKGAFLVEKT STEVQCKGGN VCVQAQDRYY NSSCSKWACV PCRVRS UniProtKB: Interleukin-12 subunit beta |
-分子 #3: Interleukin-12 receptor subunit beta-1,Death-associated protein k...
| 分子 | 名称: Interleukin-12 receptor subunit beta-1,Death-associated protein kinase 1 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: non-specific serine/threonine protein kinase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 63.789156 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QLGASGPGDG CCVEKTSFPE GASGSPLGPR NLSCYRVSKT DYECSWQYDG PEDNVSHVLW CCFVPPNHTH TGQERCRYFS SGPDRTVQF WEQDGIPVLS KVNFWVESRL GNRTMKSQKI SQYLYNWTKT TPPLGHIKVS QSHRQLRMDW NVSEEAGAEV Q FRRRMPTT ...文字列: QLGASGPGDG CCVEKTSFPE GASGSPLGPR NLSCYRVSKT DYECSWQYDG PEDNVSHVLW CCFVPPNHTH TGQERCRYFS SGPDRTVQF WEQDGIPVLS KVNFWVESRL GNRTMKSQKI SQYLYNWTKT TPPLGHIKVS QSHRQLRMDW NVSEEAGAEV Q FRRRMPTT NWTLGDCGPQ VNSGSGVLGD IRGSMSESCL CPSENMAQEI QIRRRRRLSS GAPGGPWSDW SMPVCVPPEV LP QAKIKFL VEPLNQGGRR RLTMQGQSPQ LAVPEGCRGR PGAQVKKHLV LVRMLSCRCQ AQTSKTVPLG KKLNLSGATY DLN VLAKTR FGRSTIQKWH LPAQELTETR ALNVSVGGNM TSMQWAAQAP GTTYCLEWQP WFQHRNHTHC TLIVPEEEDP AKMV THSWS SKPTLEQEEC YRITVFASKN PKNPMLWATV LSSYYFGGNA SRAGTPRHVS VRNQTGDSVS VEWTASQLST CPGVL TQYV VRCEAEDGAW ESEWLVPPTK TQVTLDGLRS RVMYKVQVRA DTARLPGAWS HPQRFSFEGT GGSGGSGGAA RKKWKQ SVR LISLCQRLS UniProtKB: Interleukin-12 receptor subunit beta-1, Death-associated protein kinase 1 |
-分子 #4: Interleukin-12 receptor subunit beta-2,Calmodulin-1
| 分子 | 名称: Interleukin-12 receptor subunit beta-2,Calmodulin-1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 85.723344 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: NIDVCKLGTV TVQPAPVIPL GSAANISCSL NPKQGCSHYP SSNELILLKF VNDVLVENLH GKKVHDHTGH SSTFQVTNLS LGMTLFVCK LNCSNSQKKP PVPVCGVEIS VGVAPEPPQN ISCVQEGENG TVACSWNSGK VTYLKTNYTL QLSGPNNLTC Q KQCFSDNR ...文字列: NIDVCKLGTV TVQPAPVIPL GSAANISCSL NPKQGCSHYP SSNELILLKF VNDVLVENLH GKKVHDHTGH SSTFQVTNLS LGMTLFVCK LNCSNSQKKP PVPVCGVEIS VGVAPEPPQN ISCVQEGENG TVACSWNSGK VTYLKTNYTL QLSGPNNLTC Q KQCFSDNR QNCNRLDLGI NLSPDLAESR FIVRVTAIND LGNSSSLPHT FTFLDIVIPL PPWDIRINFL NASGSRGTLQ WE DEGQVVL NQLRYQPLNS TSWNMVNATN AKGKYDLRDL RPFTEYEFQI SSKLHLSGGS WSNWSESLRT RTPEEEPVGI LDI WYMKQD IDYDRQQISL FWKSLNPSEA RGKILHYQVT LQEVTKKTTL QNTTRHTSWT RVIPRTGAWT ASVSAANSKG ASAP THINI VDLCGTGLLA PHQVSAKSEN MDNILVTWQP PKKADSAVRE YIVEWRALQP GSITKFPPHW LRIPPDNMSA LISEN IKPY ICYEIRVHAL SESQGGCSSI RGDSKHKAPV SGPHITAITE KKERLFISWT HIPFPEQRGC ILHYRIYWKE RDSTAQ PEL CEIQYRRSQN SHPISSLQPR VTYVLWMTAV TAAGESPQGN EREFCPQGKA NGTGGSGGSG GLTEEQIAEF KEAFSLF DK DGDGTITTKE LGTVMRSLGQ NPTEAELQDM INEVDADGNG TIDFPEFLTM MARKMKDTDS EEEIREAFRV FDKDGNGY I SAAELRHVMT NLGEKLTDEE VDEMIREADI DGDGQVNYEE FVQMMTAK UniProtKB: Interleukin-12 receptor subunit beta-2, Calmodulin-1 |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
詳細: HEPES-buffered saline (HBS) with added calcium chloride: 25 mM HEPES, pH 7.4, 150 mM NaCl, 5 mM CaCl | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: LEICA PLUNGER / 詳細: Leica EM GP2, 5 s. blotting time.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 8145 / 平均露光時間: 3.37 sec. / 平均電子線量: 61.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 60000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8pb1: |
 ムービー
ムービー コントローラー
コントローラー










































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)