+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1714 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Asymmetric structure of the empty Prochlorococcus Cyanophage P-SSP7 | |||||||||
 マップデータ マップデータ | Asymmetric structure of the empty Prochlorococcus Cyanophage P-SSP7 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Marine Podovirus / T7-like virus / Prochlorococcus MED4 | |||||||||
| 生物種 |  Prochlorococcus phage P-SSP7 (ファージ) Prochlorococcus phage P-SSP7 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 24.0 Å | |||||||||
 データ登録者 データ登録者 | Liu X / Zhang Q / Murata K / Baker ML / Sullivan MB / Fu C / Dougherty M / Schmid MF / Osburne MS / Chisholm SW / Chiu W | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2010 ジャーナル: Nat Struct Mol Biol / 年: 2010タイトル: Structural changes in a marine podovirus associated with release of its genome into Prochlorococcus. 著者: Xiangan Liu / Qinfen Zhang / Kazuyoshi Murata / Matthew L Baker / Matthew B Sullivan / Caroline Fu / Matthew T Dougherty / Michael F Schmid / Marcia S Osburne / Sallie W Chisholm / Wah Chiu /  要旨: Podovirus P-SSP7 infects Prochlorococcus marinus, the most abundant oceanic photosynthetic microorganism. Single-particle cryo-electron microscopy yields icosahedral and asymmetrical structures of ...Podovirus P-SSP7 infects Prochlorococcus marinus, the most abundant oceanic photosynthetic microorganism. Single-particle cryo-electron microscopy yields icosahedral and asymmetrical structures of infectious P-SSP7 with 4.6-A and 9-A resolution, respectively. The asymmetric reconstruction reveals how symmetry mismatches are accommodated among five of the gene products at the portal vertex. Reconstructions of infectious and empty particles show a conformational change of the 'valve' density in the nozzle, an orientation difference in the tail fibers, a disordering of the C terminus of the portal protein and the disappearance of the core proteins. In addition, cryo-electron tomography of P-SSP7 infecting Prochlorococcus showed the same tail-fiber conformation as that in empty particles. Our observations suggest a mechanism whereby, upon binding to the host cell, the tail fibers induce a cascade of structural alterations of the portal vertex complex that triggers DNA release. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1714.map.gz emd_1714.map.gz | 22.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1714-v30.xml emd-1714-v30.xml emd-1714.xml emd-1714.xml | 11 KB 11 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  pssp7-empty-asym-EBI.png pssp7-empty-asym-EBI.png | 214.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1714 http://ftp.pdbj.org/pub/emdb/structures/EMD-1714 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1714 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1714 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1714_validation.pdf.gz emd_1714_validation.pdf.gz | 210.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1714_full_validation.pdf.gz emd_1714_full_validation.pdf.gz | 209.9 KB | 表示 | |
| XML形式データ |  emd_1714_validation.xml.gz emd_1714_validation.xml.gz | 7.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1714 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1714 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1714 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1714 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1714.map.gz / 形式: CCP4 / 大きさ: 412 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1714.map.gz / 形式: CCP4 / 大きさ: 412 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetric structure of the empty Prochlorococcus Cyanophage P-SSP7 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
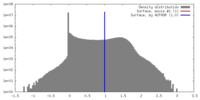
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Empty cyanophage P-SSP7
| 全体 | 名称: Empty cyanophage P-SSP7 |
|---|---|
| 要素 |
|
-超分子 #1000: Empty cyanophage P-SSP7
| 超分子 | 名称: Empty cyanophage P-SSP7 / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Prochlorococcus phage P-SSP7
| 超分子 | 名称: Prochlorococcus phage P-SSP7 / タイプ: virus / ID: 1 / Name.synonym: Cyanophage 詳細: Viral particle containing portal (gp8), capsid (gp10), adaptor (gp11), nozzle (gp12), tail fibers (gp17) proteins. NCBI-ID: 268748 / 生物種: Prochlorococcus phage P-SSP7 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes / Syn species name: Cyanophage |
|---|---|
| 宿主 | 生物種:  Prochlorococcus (バクテリア) / 別称: BACTERIA(EUBACTERIA) Prochlorococcus (バクテリア) / 別称: BACTERIA(EUBACTERIA) |
| ウイルス殻 | Shell ID: 1 / 直径: 655 Å / T番号(三角分割数): 7 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 100 mM Tris-HCl, 100 mM MgSO4, 30 mM NaCl |
| 染色 | タイプ: NEGATIVE / 詳細: No stain |
| グリッド | 詳細: 200 mesh copper grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 30 % / チャンバー内温度: 101 K / 装置: OTHER / 詳細: Vitrification instrument: FEI Vitrobot / 手法: Blot for 2 seconds 2 times before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 温度 | 最低: 4.5 K / 最高: 102 K / 平均: 101 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 400,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: JEOL エネルギーフィルター - エネルギー下限: 15.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 詳細 | MDS |
| 日付 | 2007年8月31日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 6.35 µm / 実像数: 1059 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 4.1 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダー: Side entry / 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER |
- 画像解析
画像解析
| CTF補正 | 詳細: Each micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 24.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: MPSA 詳細: The map was directly reconstructed from raw particle images without imposing any symmetry. 使用した粒子像数: 1200 |
| 最終 角度割当 | 詳細: EMAN: Z axis along 5-fold and Y axis along 2-fold asymmetry axis. |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)