+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1666 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Alpha-helical nascent polypeptide chains visualized within distinct regions of the ribosomal exit tunnel | |||||||||
 マップデータ マップデータ | 80S.DPAP-RNC | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ribosome / Protein exit tunnel / co-translational protein folding | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 7.1 Å | |||||||||
 データ登録者 データ登録者 | Bhushan S / Gartmann M / Halic M / Armache JP / Jarasch A / Mielke T / Berninghausen O / Wilson DN / Beckmann R | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2010 ジャーナル: Nat Struct Mol Biol / 年: 2010タイトル: alpha-Helical nascent polypeptide chains visualized within distinct regions of the ribosomal exit tunnel. 著者: Shashi Bhushan / Marco Gartmann / Mario Halic / Jean-Paul Armache / Alexander Jarasch / Thorsten Mielke / Otto Berninghausen / Daniel N Wilson / Roland Beckmann /  要旨: As translation proceeds, the nascent polypeptide chain passes through a tunnel in the large ribosomal subunit. Although this ribosomal exit tunnel was once thought only to be a passive conduit for ...As translation proceeds, the nascent polypeptide chain passes through a tunnel in the large ribosomal subunit. Although this ribosomal exit tunnel was once thought only to be a passive conduit for the growing nascent chain, accumulating evidence suggests that it may in fact play a more active role in regulating translation and initial protein folding events. Here we have determined single-particle cryo-electron microscopy reconstructions of eukaryotic 80S ribosomes containing nascent chains with high alpha-helical propensity located within the exit tunnel. The maps enable direct visualization of density for helices as well as allowing the sites of interaction with the tunnel wall components to be elucidated. In particular regions of the tunnel, the nascent chain adopts distinct conformations and establishes specific contacts with tunnel components, both ribosomal RNA and proteins, that have been previously implicated in nascent chain-ribosome interaction. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1666.map.gz emd_1666.map.gz | 7.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1666-v30.xml emd-1666-v30.xml emd-1666.xml emd-1666.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-1666.png EMD-1666.png | 198.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1666 http://ftp.pdbj.org/pub/emdb/structures/EMD-1666 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1666 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1666 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1666.map.gz / 形式: CCP4 / 大きさ: 185.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1666.map.gz / 形式: CCP4 / 大きさ: 185.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 80S.DPAP-RNC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
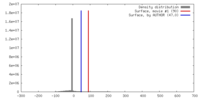
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : This map represents a wheat germ 80S ribosomal nascent chain comp...
| 全体 | 名称: This map represents a wheat germ 80S ribosomal nascent chain complex with different nascent chains |
|---|---|
| 要素 |
|
-超分子 #1000: This map represents a wheat germ 80S ribosomal nascent chain comp...
| 超分子 | 名称: This map represents a wheat germ 80S ribosomal nascent chain complex with different nascent chains タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 4.1 MDa / 理論値: 4.1 MDa / 手法: Known for 80S ribosomes |
-超分子 #1: Ribosome
| 超分子 | 名称: Ribosome / タイプ: complex / ID: 1 / Name.synonym: Ribosome / 組換発現: No / Ribosome-details: ribosome-eukaryote: ALL |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 4.1 MDa / 理論値: 4.1 MDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Hepes/KOH ph 7.5, 100 mM KOAc, 10 mM Mg(oAc)2, 1.5 mM DTT, 0.1% (w/v) Nikol |
| 染色 | タイプ: NEGATIVE / 詳細: Cryo-EM |
| グリッド | 詳細: Quantifoil grids (3/3) with 2nm carbon |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: OTHER / 詳細: Vitrification instrument: Virobot / 手法: Blot for 10 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 平均: 84 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 100,000 times magnification |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: PRIMESCAN / デジタル化 - サンプリング間隔: 4.76 µm / 実像数: 140 / 平均電子線量: 25 e/Å2 / 詳細: Scanned at 5334 dpi / Od range: 1.2 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 38000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 39000 |
| 試料ステージ | 試料ホルダー: FEI Polara cartridge system / 試料ホルダーモデル: OTHER |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Signature for particle selection and web for visual inspection |
|---|---|
| CTF補正 | 詳細: Defocus group volumes |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 7.1 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER / 使用した粒子像数: 150000 |
| 最終 角度割当 | 詳細: SPIDER |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)