+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional structure of YidC bound to the translating ribosome | |||||||||
 マップデータ マップデータ | This map shows YidC bound to the tunnel exit region of an ribosome nascent chain complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | membrane protein insertion / ribosome / translation | |||||||||
| 機能・相同性 | : / protein insertion into membrane 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 14.4 Å | |||||||||
 データ登録者 データ登録者 | Kohler R / Boehringer D / Greber B / Bingel-Erlenmeyer R / Collinson I / Schaffitzel C / Ban N | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2009 ジャーナル: Mol Cell / 年: 2009タイトル: YidC and Oxa1 form dimeric insertion pores on the translating ribosome. 著者: Rebecca Kohler / Daniel Boehringer / Basil Greber / Rouven Bingel-Erlenmeyer / Ian Collinson / Christiane Schaffitzel / Nenad Ban /  要旨: The YidC/Oxa1/Alb3 family of membrane proteins facilitates the insertion and assembly of membrane proteins in bacteria, mitochondria, and chloroplasts. Here we present the structures of both ...The YidC/Oxa1/Alb3 family of membrane proteins facilitates the insertion and assembly of membrane proteins in bacteria, mitochondria, and chloroplasts. Here we present the structures of both Escherichia coli YidC and Saccharomyces cerevisiae Oxa1 bound to E. coli ribosome nascent chain complexes determined by cryo-electron microscopy. Dimers of YidC and Oxa1 are localized above the exit of the ribosomal tunnel. Crosslinking experiments show that the ribosome specifically stabilizes the dimeric state. Functionally important and conserved transmembrane helices of YidC and Oxa1 were localized at the dimer interface by cysteine crosslinking. Both Oxa1 and YidC dimers contact the ribosome at ribosomal protein L23 and conserved rRNA helices 59 and 24, similarly to what was observed for the nonhomologous SecYEG translocon. We suggest that dimers of the YidC and Oxa1 proteins form insertion pores and share a common overall architecture with the SecY monomer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|---|
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1615.map.gz emd_1615.map.gz | 4.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1615-v30.xml emd-1615-v30.xml emd-1615.xml emd-1615.xml | 9.4 KB 9.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1615_1615.png 1615_1615.png | 175.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1615 http://ftp.pdbj.org/pub/emdb/structures/EMD-1615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1615 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1615_validation.pdf.gz emd_1615_validation.pdf.gz | 241.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1615_full_validation.pdf.gz emd_1615_full_validation.pdf.gz | 240.4 KB | 表示 | |
| XML形式データ |  emd_1615_validation.xml.gz emd_1615_validation.xml.gz | 5.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1615 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1615 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1615.map.gz / 形式: CCP4 / 大きさ: 4.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1615.map.gz / 形式: CCP4 / 大きさ: 4.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This map shows YidC bound to the tunnel exit region of an ribosome nascent chain complex | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.76 Å | ||||||||||||||||||||||||||||||||||||
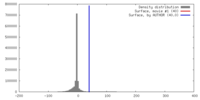
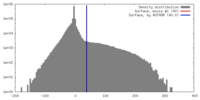
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : YidC bound to a translating ribosome
| 全体 | 名称: YidC bound to a translating ribosome |
|---|---|
| 要素 |
|
-超分子 #1000: YidC bound to a translating ribosome
| 超分子 | 名称: YidC bound to a translating ribosome / タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 2.6 MDa / 理論値: 2.6 MDa |
-超分子 #1: Ribosome nascent chain complex
| 超分子 | 名称: Ribosome nascent chain complex / タイプ: complex / ID: 1 / Name.synonym: Translating ribosome / 組換発現: No / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 2.6 MDa / 理論値: 2.6 MDa |
-分子 #1: YidC
| 分子 | 名称: YidC / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Membrane protein insertase / コピー数: 2 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 62 KDa / 理論値: 62 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: protein insertion into membrane / InterPro: INTERPRO: IPR013308 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| グリッド | 詳細: 200 mesh copper |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: Plunger |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 12.7 µm / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 16 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 50000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Each image |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 14.4 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Imagic-5 Spider / 使用した粒子像数: 24395 |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)