+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 S protein in complex with pT1696 Fab | |||||||||||||||||||||
 マップデータ マップデータ | focus map | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | PROTEIN BINDING/IMMUNE SYSTEM / PROTEIN BINDING | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / membrane fusion / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||||||||||||||
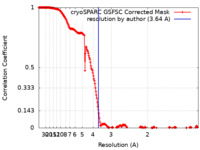
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.64 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Hansen G / Benecke T / Vollmer B / Gruenewald K / Krey T | |||||||||||||||||||||
| 資金援助 |  ドイツ, 6件 ドイツ, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: To be published ジャーナル: To be publishedタイトル: Activity of broadly neutralizing antibodies against sarbecoviruses: a trade-off between SARS-CoV-2 variants and distant coronaviruses? 著者: Stein CS / Hansen G / Ssebyatika GL / Stroeh L / Benecke T / Menz S / Waldmann J-Y / Vollmer B / Tipp S / Ochulor O / Herold E / Schwarzloh B / Mutschall D / Zischke J-Y / Cordes A / Puppe W ...著者: Stein CS / Hansen G / Ssebyatika GL / Stroeh L / Benecke T / Menz S / Waldmann J-Y / Vollmer B / Tipp S / Ochulor O / Herold E / Schwarzloh B / Mutschall D / Zischke J-Y / Cordes A / Puppe W / Schneider T / Hinrichs I / Blasczyk R / Kleine-Weber H / Hoffmann M / Hoeper M / Kaiser FK / Gonzalez-Hernandez M / Armando FK / Ciurkiewicz M / Beythien G / Poehlmann S / Baumgaertner W / Gruenewald K / Osterhaus A / Schulz T / Krey T | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16026.map.gz emd_16026.map.gz | 92.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16026-v30.xml emd-16026-v30.xml emd-16026.xml emd-16026.xml | 22.4 KB 22.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16026_fsc.xml emd_16026_fsc.xml | 21.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16026.png emd_16026.png | 30 KB | ||
| Filedesc metadata |  emd-16026.cif.gz emd-16026.cif.gz | 7.3 KB | ||
| その他 |  emd_16026_additional_1.map.gz emd_16026_additional_1.map.gz emd_16026_half_map_1.map.gz emd_16026_half_map_1.map.gz emd_16026_half_map_2.map.gz emd_16026_half_map_2.map.gz | 4.5 MB 926.6 MB 926.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16026 http://ftp.pdbj.org/pub/emdb/structures/EMD-16026 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16026 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16026 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16026_validation.pdf.gz emd_16026_validation.pdf.gz | 934.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16026_full_validation.pdf.gz emd_16026_full_validation.pdf.gz | 933.8 KB | 表示 | |
| XML形式データ |  emd_16026_validation.xml.gz emd_16026_validation.xml.gz | 8.6 KB | 表示 | |
| CIF形式データ |  emd_16026_validation.cif.gz emd_16026_validation.cif.gz | 4.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16026 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16026 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16026 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16026 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bg8MC  8becC  8bg1C  8bg2C  8bg3C  8bg4C  8bg5C  8bg6C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16026.map.gz / 形式: CCP4 / 大きさ: 104.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16026.map.gz / 形式: CCP4 / 大きさ: 104.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | focus map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.68 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: focus map, local filter
| ファイル | emd_16026_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | focus map, local filter | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16026_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16026_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 3:3 pT1696 Fab-Spike complex
| 全体 | 名称: 3:3 pT1696 Fab-Spike complex |
|---|---|
| 要素 |
|
-超分子 #1: 3:3 pT1696 Fab-Spike complex
| 超分子 | 名称: 3:3 pT1696 Fab-Spike complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: pT1696 Fab
| 超分子 | 名称: pT1696 Fab / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Spike protein S1
| 超分子 | 名称: Spike protein S1 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 140.749328 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VNLTTRTQLP PAYTNSFTRG VYYPDKVFRS SVLHSTQDLF LPFFSNVTWF HAIHVSGTNG TKRFDNPVLP FNDGVYFAST EKSNIIRGW IFGTTLDSKT QSLLIVNNAT NVVIKVCEFQ FCNDPFLGVY YHKNNKSWME SEFRVYSSAN NCTFEYVSQP F LMDLEGKQ ...文字列: VNLTTRTQLP PAYTNSFTRG VYYPDKVFRS SVLHSTQDLF LPFFSNVTWF HAIHVSGTNG TKRFDNPVLP FNDGVYFAST EKSNIIRGW IFGTTLDSKT QSLLIVNNAT NVVIKVCEFQ FCNDPFLGVY YHKNNKSWME SEFRVYSSAN NCTFEYVSQP F LMDLEGKQ GNFKNLREFV FKNIDGYFKI YSKHTPINLV RDLPQGFSAL EPLVDLPIGI NITRFQTLLA LHRSYLTPGD SS SGWTAGA AAYYVGYLQP RTFLLKYNEN GTITDAVDCA LDPLSETKCT LKSFTVEKGI YQTSNFRVQP TESIVRFPNI TNL CPFGEV FNATRFASVY AWNRKRISNC VADYSVLYNS ASFSTFKCYG VSPTKLNDLC FTNVYADSFV IRGDEVRQIA PGQT GKIAD YNYKLPDDFT GCVIAWNSNN LDSKVGGNYN YLYRLFRKSN LKPFERDIST EIYQAGSTPC NGVEGFNCYF PLQSY GFQP TNGVGYQPYR VVVLSFELLH APATVCGPKK STNLVKNKCV NFNFNGLTGT GVLTESNKKF LPFQQFGRDI ADTTDA VRD PQTLEILDIT PCSFGGVSVI TPGTNTSNQV AVLYQDVNCT EVPVAIHADQ LTPTWRVYST GSNVFQTRAG CLIGAEH VN NSYECDIPIG AGICASYQTQ TNSPGSASSV ASQSIIAYTM SLGAENSVAY SNNSIAIPTN FTISVTTEIL PVSMTKTS V DCTMYICGDS TECSNLLLQY GSFCTQLNRA LTGIAVEQDK NTQEVFAQVK QIYKTPPIKD FGGFNFSQIL PDPSKPSKR SPIEDLLFNK VTLADAGFIK QYGDCLGDIA ARDLICAQKF NGLTVLPPLL TDEMIAQYTS ALLAGTITSG WTFGAGPALQ IPFPMQMAY RFNGIGVTQN VLYENQKLIA NQFNSAIGKI QDSLSSTPSA LGKLQDVVNQ NAQALNTLVK QLSSNFGAIS S VLNDILSR LDPPEAEVQI DRLITGRLQS LQTYVTQQLI RAAEIRASAN LAATKMSECV LGQSKRVDFC GKGYHLMSFP QS APHGVVF LHVTYVPAQE KNFTTAPAIC HDGKAHFPRE GVFVSNGTHW FVTQRNFYEP QIITTDNTFV SGNCDVVIGI VNN TVYDPL QPELDSFKEE LDKYFKNHTS PDVDLGDISG INASVVNIQK EIDRLNEVAK NLNESLIDLQ ELGKYEQGSG YIPE APRDG QAYVRKDGEW VLLSTFLGRS LEVLFQGPGH HHHHHHHSAW SHPQFEKGGG SGGGGSGGSA WSHPQFEK UniProtKB: Spike glycoprotein |
-分子 #2: pT1696 Fab heavy chain
| 分子 | 名称: pT1696 Fab heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.103283 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QMQLEQSGPE VKKPGTSVKV SCKASGFTFS SAAVQWVRQA RGQRLEWMGW IVVGSGNANY AQKFQERVTI TRDMSTSIAY MELSSLRSE DTAVYYCAAP HCNRTSCLDG FDIWGQGTMV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: QMQLEQSGPE VKKPGTSVKV SCKASGFTFS SAAVQWVRQA RGQRLEWMGW IVVGSGNANY AQKFQERVTI TRDMSTSIAY MELSSLRSE DTAVYYCAAP HCNRTSCLDG FDIWGQGTMV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KRVEPKSCDK TDDDDKAGWS HP QFEKGGG SGGGSGGGSW SHPQFEK |
-分子 #3: pT1696 Fab light chain
| 分子 | 名称: pT1696 Fab light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.701223 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ASEIVLTQSP GTLSLSPGER ATLSCRASQS VSRNYLAWYQ QKPGQAPRLL IYGASSRATG IPDRFSGSGS GTDFTLNISR LEPEDFAVY YCQQYGSSPW TFGQGTKVDI KRTVAAPSVF IFPPSDEQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS G NSQESVTE ...文字列: ASEIVLTQSP GTLSLSPGER ATLSCRASQS VSRNYLAWYQ QKPGQAPRLL IYGASSRATG IPDRFSGSGS GTDFTLNISR LEPEDFAVY YCQQYGSSPW TFGQGTKVDI KRTVAAPSVF IFPPSDEQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS G NSQESVTE QDSKDSTYSL SSTLTLSKAD YEKHKVYACE VTHQGLSSPV TKSFNRGEC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.34 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)