+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1465 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional structure of vertebrate cardiac muscle myosin filaments. | |||||||||
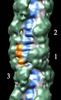
 マップデータ マップデータ | Reconstruction of cardiac myosin filaments (C-zone) under relaxing conditions. The reconstruction (filtered to 4.0 nm resolution) shows two 42.9 nm repeats. The main globular features are myosin heads, which are arranged in three crowns within each 42.9 nm repeat, following a perturbed helical path. Each crown has 3-fold rotational symmetry. Smaller features with a periodicity of about 4 nm can also be observed. Those features represent the accessory proteins titin and myosin-binding protein-C. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / ネガティブ染色法 / 解像度: 32.0 Å | |||||||||
 データ登録者 データ登録者 | Zoghbi ME / Woodhead JL / Moss RL / Craig R | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2008 ジャーナル: Proc Natl Acad Sci U S A / 年: 2008タイトル: Three-dimensional structure of vertebrate cardiac muscle myosin filaments. 著者: Maria E Zoghbi / John L Woodhead / Richard L Moss / Roger Craig /  要旨: Contraction of the heart results from interaction of the myosin and actin filaments. Cardiac myosin filaments consist of the molecular motor myosin II, the sarcomeric template protein, titin, and the ...Contraction of the heart results from interaction of the myosin and actin filaments. Cardiac myosin filaments consist of the molecular motor myosin II, the sarcomeric template protein, titin, and the cardiac modulatory protein, myosin binding protein C (MyBP-C). Inherited hypertrophic cardiomyopathy (HCM) is a disease caused mainly by mutations in these proteins. The structure of cardiac myosin filaments and the alterations caused by HCM mutations are unknown. We have used electron microscopy and image analysis to determine the three-dimensional structure of myosin filaments from wild-type mouse cardiac muscle and from a MyBP-C knockout model for HCM. Three-dimensional reconstruction of the wild-type filament reveals the conformation of the myosin heads and the organization of titin and MyBP-C at 4 nm resolution. Myosin heads appear to interact with each other intramolecularly, as in off-state smooth muscle myosin [Wendt T, Taylor D, Trybus KM, Taylor K (2001) Proc Natl Acad Sci USA 98:4361-4366], suggesting that all relaxed muscle myosin IIs may adopt this conformation. Titin domains run in an elongated strand along the filament surface, where they appear to interact with part of MyBP-C and with the myosin backbone. In the knockout filament, some of the myosin head interactions are disrupted, suggesting that MyBP-C is important for normal relaxation of the filament. These observations provide key insights into the role of the myosin filament in cardiac contraction, assembly, and disease. The techniques we have developed should be useful in studying the structural basis of other myosin-related HCM diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1465.map.gz emd_1465.map.gz | 2.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1465-v30.xml emd-1465-v30.xml emd-1465.xml emd-1465.xml | 11.9 KB 11.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1465.gif 1465.gif emd_1465.tif emd_1465.tif emd_1465_1.tif emd_1465_1.tif | 62.7 KB 3.2 MB 13.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1465 http://ftp.pdbj.org/pub/emdb/structures/EMD-1465 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1465 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1465 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1465_validation.pdf.gz emd_1465_validation.pdf.gz | 205.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1465_full_validation.pdf.gz emd_1465_full_validation.pdf.gz | 204.2 KB | 表示 | |
| XML形式データ |  emd_1465_validation.xml.gz emd_1465_validation.xml.gz | 4.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1465 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1465 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1465 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1465 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1465.map.gz / 形式: CCP4 / 大きさ: 2.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1465.map.gz / 形式: CCP4 / 大きさ: 2.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of cardiac myosin filaments (C-zone) under relaxing conditions. The reconstruction (filtered to 4.0 nm resolution) shows two 42.9 nm repeats. The main globular features are myosin heads, which are arranged in three crowns within each 42.9 nm repeat, following a perturbed helical path. Each crown has 3-fold rotational symmetry. Smaller features with a periodicity of about 4 nm can also be observed. Those features represent the accessory proteins titin and myosin-binding protein-C. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cardiac myosin filaments from mouse ventricle muscle
| 全体 | 名称: Cardiac myosin filaments from mouse ventricle muscle |
|---|---|
| 要素 |
|
-超分子 #1000: Cardiac myosin filaments from mouse ventricle muscle
| 超分子 | 名称: Cardiac myosin filaments from mouse ventricle muscle タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-分子 #1: Myosin
| 分子 | 名称: Myosin / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Myosin / 詳細: Filament is a polymer of myosin / 集合状態: polymer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 520 KDa / 理論値: 520 KDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 詳細: 100 mM NaCl, 2 mM EGTA, 5 mM MgCl2, 1 mM DTT, 10 mM Imidazole, 5 mM MgATP, 0.1 mM blebbistatin,pH 7.2 |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: A drop of filament suspension was placed on an electron microscope grid coated with a thin layer of carbon supported by a thicker holey carbon film. The grid was rinsed sequentially with 6 ...詳細: A drop of filament suspension was placed on an electron microscope grid coated with a thin layer of carbon supported by a thicker holey carbon film. The grid was rinsed sequentially with 6 drops of relaxing rinse (in mM: 140 NaAc, 1 MgAc2, 1 EGTA, 5 Imidazole, 1 sodium azide, 1 MgATP, pH 7.0 ) and 5 drops of 2% uranyl acetate. Staining was carried out at room temperature with solutions pre-warmed to 37o C. |
| グリッド | 詳細: carbon holey grids (400 mesh copper) |
| 凍結 | 凍結剤: NONE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM120T |
|---|---|
| アライメント法 | Legacy - 非点収差: corrected at 240,000 x |
| 詳細 | Grids were observed in a Philips CM120 electron microscope (FEI, Hillsboro, OR) under low dose conditions. Only filaments on thin carbon over holes were photographed . |
| 日付 | 2006年5月30日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F224 (2k x 2k) 実像数: 300 / 詳細: Images were acquired on a CDD camera at 5.7 A/pixel / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 80 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 42000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.1 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| 詳細 | The filament shows perturbations from a perfect helical structure. |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - 軸対称性: C3 (3回回転対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 32.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER 詳細: Since these filaments are not perfectly helical, we used single particle analysis for their reconstruction. Filaments were oriented vertically and the region between the 3rd and 10th 42.9 nm ...詳細: Since these filaments are not perfectly helical, we used single particle analysis for their reconstruction. Filaments were oriented vertically and the region between the 3rd and 10th 42.9 nm repeats from the bare zone (where MyBP-C is present) was computationally cut. Those selected filament regions were converted to SPIDER format (EM2EM; Image Science and Imperial College, London), and cut into segments 3x42.9 nm long in SPIDER (v11.2, Wadsworth Center, Albany, NY). Relative rotations of different filament segments were determined before back-projection by matching filament images against 2D projections of 3D models rotated around their long axis at known angles. C3 symmetry was imposed during reconstruction. A total of 2564 segments (2600 particles) were used for the reconstruction. |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
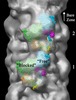
| 詳細 | Atomic structure of the myosin heads (pdb 1i84; with the modifications introduced by Woodhead et al., 2005. Nature. 436:1195)was fitted manually to the globular features of the reconstruction using UCSF Chimera. |
| 精密化 | プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera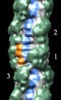


 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















