+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram averaging of Rubisco from native Halothiobacillus carboxysomes | ||||||||||||
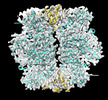
 マップデータ マップデータ | final merged map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Rubisco / carboxysome / UNKNOWN FUNCTION | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報carboxysome / ribulose-bisphosphate carboxylase / ribulose-bisphosphate carboxylase activity / reductive pentose-phosphate cycle / monooxygenase activity / magnesium ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Halothiobacillus neapolitanus (紅色硫黄細菌) Halothiobacillus neapolitanus (紅色硫黄細菌) | ||||||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||
 データ登録者 データ登録者 | Ni T / Zhu Y / Yu X / Sun Y / Liu L / Zhang P | ||||||||||||
| 資金援助 | European Union,  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure and assembly of cargo Rubisco in two native α-carboxysomes. 著者: Tao Ni / Yaqi Sun / Will Burn / Monsour M J Al-Hazeem / Yanan Zhu / Xiulian Yu / Lu-Ning Liu / Peijun Zhang /   要旨: Carboxysomes are a family of bacterial microcompartments in cyanobacteria and chemoautotrophs. They encapsulate Ribulose 1,5-bisphosphate carboxylase/oxygenase (Rubisco) and carbonic anhydrase ...Carboxysomes are a family of bacterial microcompartments in cyanobacteria and chemoautotrophs. They encapsulate Ribulose 1,5-bisphosphate carboxylase/oxygenase (Rubisco) and carbonic anhydrase catalyzing carbon fixation inside a proteinaceous shell. How Rubisco complexes pack within the carboxysomes is unknown. Using cryo-electron tomography, we determine the distinct 3D organization of Rubisco inside two distant α-carboxysomes from a marine α-cyanobacterium Cyanobium sp. PCC 7001 where Rubiscos are organized in three concentric layers, and from a chemoautotrophic bacterium Halothiobacillus neapolitanus where they form intertwining spirals. We further resolve the structures of native Rubisco as well as its higher-order assembly at near-atomic resolutions by subtomogram averaging. The structures surprisingly reveal that the authentic intrinsically disordered linker protein CsoS2 interacts with Rubiscos in native carboxysomes but functions distinctively in the two α-carboxysomes. In contrast to the uniform Rubisco-CsoS2 association in the Cyanobium α-carboxysome, CsoS2 binds only to the Rubiscos close to the shell in the Halo α-carboxysome. Our findings provide critical knowledge of the assembly principles of α-carboxysomes, which may aid in the rational design and repurposing of carboxysome structures for new functions. #1:  ジャーナル: bioRxiv / 年: 2022 ジャーナル: bioRxiv / 年: 2022タイトル: Tales of Two alpha Carboxysomes the Structure and Assembly of Cargo Rubisco 著者: Ni T / Sun Y / Seaton-Burn W / AI-Hazeem M / Zhu Y / Yu X / Liu L / Zhang P | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14590.map.gz emd_14590.map.gz | 3.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14590-v30.xml emd-14590-v30.xml emd-14590.xml emd-14590.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14590_fsc.xml emd_14590_fsc.xml | 36.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14590.png emd_14590.png | 198.6 KB | ||
| Filedesc metadata |  emd-14590.cif.gz emd-14590.cif.gz | 6.6 KB | ||
| その他 |  emd_14590_half_map_1.map.gz emd_14590_half_map_1.map.gz emd_14590_half_map_2.map.gz emd_14590_half_map_2.map.gz | 19.4 MB 19.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14590 http://ftp.pdbj.org/pub/emdb/structures/EMD-14590 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14590 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14590 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14590_validation.pdf.gz emd_14590_validation.pdf.gz | 837.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14590_full_validation.pdf.gz emd_14590_full_validation.pdf.gz | 837.2 KB | 表示 | |
| XML形式データ |  emd_14590_validation.xml.gz emd_14590_validation.xml.gz | 23.7 KB | 表示 | |
| CIF形式データ |  emd_14590_validation.cif.gz emd_14590_validation.cif.gz | 33.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14590 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14590 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14590 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14590 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zbtMC  7zc1C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14590.map.gz / 形式: CCP4 / 大きさ: 21.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14590.map.gz / 形式: CCP4 / 大きさ: 21.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | final merged map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||
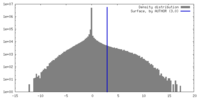
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map 2
| ファイル | emd_14590_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_14590_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : alpha carboxysomes
| 全体 | 名称: alpha carboxysomes |
|---|---|
| 要素 |
|
-超分子 #1: alpha carboxysomes
| 超分子 | 名称: alpha carboxysomes / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Halothiobacillus neapolitanus (紅色硫黄細菌) Halothiobacillus neapolitanus (紅色硫黄細菌) |
-分子 #1: Ribulose bisphosphate carboxylase large chain
| 分子 | 名称: Ribulose bisphosphate carboxylase large chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO / EC番号: ribulose-bisphosphate carboxylase |
|---|---|
| 由来(天然) | 生物種:  Halothiobacillus neapolitanus (紅色硫黄細菌) Halothiobacillus neapolitanus (紅色硫黄細菌)株: ATCC 23641 / c2 |
| 分子量 | 理論値: 52.7025 KDa |
| 組換発現 | 生物種:  Halothiobacillus neapolitanus (紅色硫黄細菌) Halothiobacillus neapolitanus (紅色硫黄細菌) |
| 配列 | 文字列: MAVKKYSAGV KEYRQTYWMP EYTPLDSDIL ACFKITPQPG VDREEAAAAV AAESSTGTWT TVWTDLLTDM DYYKGRAYRI EDVPGDDAA FYAFIAYPID LFEEGSVVNV FTSLVGNVFG FKAVRGLRLE DVRFPLAYVK TCGGPPHGIQ VERDKMNKYG R PLLGCTIK ...文字列: MAVKKYSAGV KEYRQTYWMP EYTPLDSDIL ACFKITPQPG VDREEAAAAV AAESSTGTWT TVWTDLLTDM DYYKGRAYRI EDVPGDDAA FYAFIAYPID LFEEGSVVNV FTSLVGNVFG FKAVRGLRLE DVRFPLAYVK TCGGPPHGIQ VERDKMNKYG R PLLGCTIK PKLGLSAKNY GRAVYECLRG GLDFTKDDEN INSQPFMRWR DRFLFVQDAT ETAEAQTGER KGHYLNVTAP TP EEMYKRA EFAKEIGAPI IMHDYITGGF TANTGLAKWC QDNGVLLHIH RAMHAVIDRN PNHGIHFRVL TKILRLSGGD HLH TGTVVG KLEGDRASTL GWIDLLRESF IPEDRSRGIF FDQDWGSMPG VFAVASGGIH VWHMPALVNI FGDDSVLQFG GGTL GHPWG NAAGAAANRV ALEACVEARN QGRDIEKEGK EILTAAAQHS PELKIAMETW KEIKFEFDTV DKLDTQNR UniProtKB: Ribulose bisphosphate carboxylase large chain |
-分子 #2: Ribulose bisphosphate carboxylase small subunit
| 分子 | 名称: Ribulose bisphosphate carboxylase small subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Halothiobacillus neapolitanus (紅色硫黄細菌) Halothiobacillus neapolitanus (紅色硫黄細菌)株: ATCC 23641 / c2 |
| 分子量 | 理論値: 12.866575 KDa |
| 組換発現 | 生物種:  Halothiobacillus neapolitanus (紅色硫黄細菌) Halothiobacillus neapolitanus (紅色硫黄細菌) |
| 配列 | 文字列: MAEMQDYKQS LKYETFSYLP PMNAERIRAQ IKYAIAQGWS PGIEHVEVKN SMNQYWYMWK LPFFGEQNVD NVLAEIEACR SAYPTHQVK LVAYDNYAQS LGLAFVVYRG N UniProtKB: Ribulose bisphosphate carboxylase small subunit |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 3.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)