+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AP2 on the membrane without cargo peptide | |||||||||
 マップデータ マップデータ | Relion_postprocess generated map, low-pass filtered according to local resolution, sharpened with global B factor -1800 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | adaptins / endocytosis / clathrin-mediated | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   | |||||||||
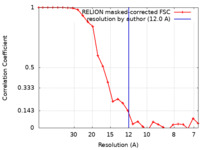
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Kovtun O / Kaufman JGG / Owen DJ / Briggs JAG | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: FCHO controls AP2's initiating role in endocytosis through a PtdIns(4,5)P-dependent switch. 著者: Nathan R Zaccai / Zuzana Kadlecova / Veronica Kane Dickson / Kseniya Korobchevskaya / Jan Kamenicky / Oleksiy Kovtun / Perunthottathu K Umasankar / Antoni G Wrobel / Jonathan G G Kaufman / ...著者: Nathan R Zaccai / Zuzana Kadlecova / Veronica Kane Dickson / Kseniya Korobchevskaya / Jan Kamenicky / Oleksiy Kovtun / Perunthottathu K Umasankar / Antoni G Wrobel / Jonathan G G Kaufman / Sally R Gray / Kun Qu / Philip R Evans / Marco Fritzsche / Filip Sroubek / Stefan Höning / John A G Briggs / Bernard T Kelly / David J Owen / Linton M Traub /      要旨: Clathrin-mediated endocytosis (CME) is the main mechanism by which mammalian cells control their cell surface proteome. Proper operation of the pivotal CME cargo adaptor AP2 requires membrane- ...Clathrin-mediated endocytosis (CME) is the main mechanism by which mammalian cells control their cell surface proteome. Proper operation of the pivotal CME cargo adaptor AP2 requires membrane-localized Fer/Cip4 homology domain-only proteins (FCHO). Here, live-cell enhanced total internal reflection fluorescence-structured illumination microscopy shows that FCHO marks sites of clathrin-coated pit (CCP) initiation, which mature into uniform-sized CCPs comprising a central patch of AP2 and clathrin corralled by an FCHO/Epidermal growth factor potential receptor substrate number 15 (Eps15) ring. We dissect the network of interactions between the FCHO interdomain linker and AP2, which concentrates, orients, tethers, and partially destabilizes closed AP2 at the plasma membrane. AP2's subsequent membrane deposition drives its opening, which triggers FCHO displacement through steric competition with phosphatidylinositol 4,5-bisphosphate, clathrin, cargo, and CME accessory factors. FCHO can now relocate toward a CCP's outer edge to engage and activate further AP2s to drive CCP growth/maturation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14526.map.gz emd_14526.map.gz | 697.6 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14526-v30.xml emd-14526-v30.xml emd-14526.xml emd-14526.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14526_fsc.xml emd_14526_fsc.xml | 2.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14526.png emd_14526.png | 63.2 KB | ||
| マスクデータ |  emd_14526_msk_1.map emd_14526_msk_1.map | 1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14526.cif.gz emd-14526.cif.gz | 6.7 KB | ||
| その他 |  emd_14526_half_map_1.map.gz emd_14526_half_map_1.map.gz emd_14526_half_map_2.map.gz emd_14526_half_map_2.map.gz | 967.9 KB 968 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14526 http://ftp.pdbj.org/pub/emdb/structures/EMD-14526 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14526 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14526 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14526.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14526.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion_postprocess generated map, low-pass filtered according to local resolution, sharpened with global B factor -1800 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.402 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14526_msk_1.map emd_14526_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: unsharpened halfmap 2
| ファイル | emd_14526_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: unsharpened halfmap 1
| ファイル | emd_14526_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Clathrin adaptor protein AP2 recruited on the membrane in the abs...
| 全体 | 名称: Clathrin adaptor protein AP2 recruited on the membrane in the absence of a cargo peptide. |
|---|---|
| 要素 |
|
-超分子 #1: Clathrin adaptor protein AP2 recruited on the membrane in the abs...
| 超分子 | 名称: Clathrin adaptor protein AP2 recruited on the membrane in the absence of a cargo peptide. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: AP-2 complex subunit alpha-2
| 分子 | 名称: AP-2 complex subunit alpha-2 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPAVSKGDGM RGLAVFISD I RNCKSKEA EI KRINKEL ANI RSKFKG DKAL DGYSK KKYVC KLLF IFLLGH DID FGHMEAV NL LSSNRYTE K QIGYLFISV LVNSNSELIR LINNAIKND L ASRNPTFM GL ALHCIAN VGS REMAEA FAGE IPKIL ...文字列: MPAVSKGDGM RGLAVFISD I RNCKSKEA EI KRINKEL ANI RSKFKG DKAL DGYSK KKYVC KLLF IFLLGH DID FGHMEAV NL LSSNRYTE K QIGYLFISV LVNSNSELIR LINNAIKND L ASRNPTFM GL ALHCIAN VGS REMAEA FAGE IPKIL VAGDT MDSV KQSAAL CLL RLYRTSP DL VPMGDWTS R VVHLLNDQH LGVVTAATSL ITTLAQKNP E EFKTSVSL AV SRLSRIV TSA STDLQD YTYY FVPAP WLSVK LLRL LQCYPP PED PAVRGRL TE CLETILNK A QEPPKSKKV QHSNAKNAVL FEAISLIIH H DSEPNLLV RA CNQLGQF LQH RETNLR YLAL ESMCT LASSE FSHE AVKTHI ETV INALKTE RD VSVRQRAV D LLYAMCDRS NAQQIVAEML SYLETADYS I REEIVLKV AI LAEKYAV DYT WYVDTI LNLI RIAGD YVSEE VWYR VIQIVI NRD DVQGYAA KT VFEALQAP A CHENLVKVG GYILGEFGNL IAGDPRSSP L IQFNLLHS KF HLCSVPT RAL LLSTYI KFVN LFPEV KATIQ DVLR SDSQLK NAD VELQQRA VE YLRLSTVA S TDILATVLE EMPPFPERES SILAKLKKK K GGSGLVPR |
-分子 #2: AP-2 complex subunit beta
| 分子 | 名称: AP-2 complex subunit beta / タイプ: protein_or_peptide / ID: 2 / 詳細: N-terminal His-tag / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHMTD SKYFTTNKK G EIFELKAE LN NEKKEKR KEA VKKVIA AMTV GKDVS SLFPD VVNC MQTDNL ELK KLVYLYL MN YAKSQPDM A IMAVNSFVK DCEDPNPLIR ALAVRTMGC I RVDKITEY LC EPLRKCL KDE DPYVRK TAAV CVAKL ...文字列: MHHHHHHMTD SKYFTTNKK G EIFELKAE LN NEKKEKR KEA VKKVIA AMTV GKDVS SLFPD VVNC MQTDNL ELK KLVYLYL MN YAKSQPDM A IMAVNSFVK DCEDPNPLIR ALAVRTMGC I RVDKITEY LC EPLRKCL KDE DPYVRK TAAV CVAKL HDINA QMVE DQGFLD SLR DLIADSN PM VVANAVAA L SEISESHPN SNLLDLNPQN INKLLTALN E CTEWGQIF IL DCLSNYN PKD DREAQS ICER VTPRL SHANS AVVL SAVKVL MKF LELLPKD SD YYNMLLKK L APPLVTLLS GEPEVQYVAL RNINLIVQK R PEILKQEI KV FFVKYND PIY VKLEKL DIMI RLASQ ANIAQ VLAE LKEYAT EVD VDFVRKA VR AIGRCAIK V EQSAERCVS TLLDLIQTKV NYVVQEAIV V IRDIFRKY PN KYESIIA TLC ENLDSL DEPD ARAAM IWIVG EYAE RIDNAD ELL ESFLEGF HD ESTQVQLT L LTAIVKLFL KKPSETQELV QQVLSLATQ D SDNPDLRD RG YIYWRLL STD PVTAKE VVLS EKPLI SEETD LIEP TLLDEL ICH IGSLASV YH KPPNAFVE G SHGIHRKHL PIHHGSTDAG DSPVGTTTA T NLEQPQVI PS QGDLLGD LLN LDLGPP VNVP QVSSM QMGAV DLLG GGLDSL VGQ SFIPSSV PA TFAPSPTP A VVSSGLNDL FELSTGIGMA PGGYVAPKA V WLPAVKAK GL EISGTFT HRQ GHIYME MNFT NKALQ HMTDF AIQF NKNSFG VIP STPLAIH TP LMPNQSID V SLPLNTLGP VMKMEPLNNL QVAVKNNID V FYFSCLIP LN VLFVEDG KME RQVFLA TWKD IPNEN ELQFQ IKEC HLNADT VSS KLQNNNV YT IAKRNVEG Q DMLYQSLKL TNGIWILAEL RIQPGNPNY T LSLKCRAP EV SQYIYQV YDS ILKN |
-分子 #3: AP-2 complex subunit mu
| 分子 | 名称: AP-2 complex subunit mu / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIGGLFIYNH KGEVLISRV Y RDDIGRNA VD AFRVNVI HAR QQVRSP VTNI ARTSF FHVKR SNIW LAAVTK QNV NAAMVFE FL YKMCDVMA A YFGKISEEN IKNNFVLIYE LLDEILDFG Y PQNSETGA LK TFITQQG IKS QHQTKE EQSQ ITSQV ...文字列: MIGGLFIYNH KGEVLISRV Y RDDIGRNA VD AFRVNVI HAR QQVRSP VTNI ARTSF FHVKR SNIW LAAVTK QNV NAAMVFE FL YKMCDVMA A YFGKISEEN IKNNFVLIYE LLDEILDFG Y PQNSETGA LK TFITQQG IKS QHQTKE EQSQ ITSQV TGQIG WRRE GIKYRR NEL FLDVLES VN LLMSPQGQ V LSAHVSGRV VMKSYLSGMP ECKFGMNDK I VIEKQGKG TA DETSKSM EQK LISEED LGKQ SIAID DCTFH QCVR LSKFDS ERS ISFIPPD GE FELMRYRT T KDIILPFRV IPLVREVGRT KLEVKVVIK S NFKPSLLA QK IEVRIPT PLN TSGVQV ICMK GKAKY KASEN AIVW KIKRMA GMK ESQISAE IE LLPTNDKK K WARPPISMN FEVPFAPSGL KVRYLKVFE P KLNYSDHD VI KWVRYIG RSG IYETRC |
-分子 #4: AP-2 complex subunit sigma
| 分子 | 名称: AP-2 complex subunit sigma / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIRFILIQNR AGKTRLAKW Y MQFDDDEK QK LIEEVHA VVT VRDAKH TNFV EFRNF KIIYR RYAG LYFCIC VDV NDNNLAY LE AIHNFVEV L NEYFHNVCE LDLVFNFYKV YTVVDEMFL A GEIRETSQ TK VLKQLLM LQS LE |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 3.17 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)