+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Bacillus pseudofirmus Mrp antiporter complex, monomer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Antiporter / Electron transport / Complex / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / monoatomic ion transmembrane transporter activity / monoatomic cation transmembrane transporter activity / : / antiporter activity / sodium ion transport / membrane => GO:0016020 / NADH dehydrogenase (ubiquinone) activity / ATP synthesis coupled electron transport / monoatomic ion transport ...: / monoatomic ion transmembrane transporter activity / monoatomic cation transmembrane transporter activity / : / antiporter activity / sodium ion transport / membrane => GO:0016020 / NADH dehydrogenase (ubiquinone) activity / ATP synthesis coupled electron transport / monoatomic ion transport / proton transmembrane transport / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Alkalihalobacillus pseudofirmus (バクテリア) / Alkalihalobacillus pseudofirmus (バクテリア) /  Alkalihalophilus pseudofirmus (バクテリア) Alkalihalophilus pseudofirmus (バクテリア) | |||||||||
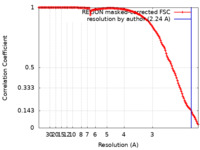
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.24 Å | |||||||||
 データ登録者 データ登録者 | Lee Y | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Ion transfer mechanisms in Mrp-type antiporters from high resolution cryoEM and molecular dynamics simulations. 著者: Yongchan Lee / Outi Haapanen / Anton Altmeyer / Werner Kühlbrandt / Vivek Sharma / Volker Zickermann /    要旨: Multiple resistance and pH adaptation (Mrp) cation/proton antiporters are essential for growth of a variety of halophilic and alkaliphilic bacteria under stress conditions. Mrp-type antiporters are ...Multiple resistance and pH adaptation (Mrp) cation/proton antiporters are essential for growth of a variety of halophilic and alkaliphilic bacteria under stress conditions. Mrp-type antiporters are closely related to the membrane domain of respiratory complex I. We determined the structure of the Mrp antiporter from Bacillus pseudofirmus by electron cryo-microscopy at 2.2 Å resolution. The structure resolves more than 99% of the sidechains of the seven membrane subunits MrpA to MrpG plus 360 water molecules, including ~70 in putative ion translocation pathways. Molecular dynamics simulations based on the high-resolution structure revealed details of the antiport mechanism. We find that switching the position of a histidine residue between three hydrated pathways in the MrpA subunit is critical for proton transfer that drives gated trans-membrane sodium translocation. Several lines of evidence indicate that the same histidine-switch mechanism operates in respiratory complex I. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14124.map.gz emd_14124.map.gz | 10.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14124-v30.xml emd-14124-v30.xml emd-14124.xml emd-14124.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14124_fsc.xml emd_14124_fsc.xml | 14 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14124.png emd_14124.png | 91.2 KB | ||
| マスクデータ |  emd_14124_msk_1.map emd_14124_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14124.cif.gz emd-14124.cif.gz | 6.9 KB | ||
| その他 |  emd_14124_half_map_1.map.gz emd_14124_half_map_1.map.gz emd_14124_half_map_2.map.gz emd_14124_half_map_2.map.gz | 194.7 MB 194.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14124 http://ftp.pdbj.org/pub/emdb/structures/EMD-14124 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14124 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14124 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14124_validation.pdf.gz emd_14124_validation.pdf.gz | 680.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14124_full_validation.pdf.gz emd_14124_full_validation.pdf.gz | 680 KB | 表示 | |
| XML形式データ |  emd_14124_validation.xml.gz emd_14124_validation.xml.gz | 21.4 KB | 表示 | |
| CIF形式データ |  emd_14124_validation.cif.gz emd_14124_validation.cif.gz | 28.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14124 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14124 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14124 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14124 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7qruMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14124.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14124.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07136 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14124_msk_1.map emd_14124_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14124_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14124_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : MrpABCDEFG complex
+超分子 #1: MrpABCDEFG complex
+分子 #1: Na+/H+ antiporter subunit D
+分子 #2: Na+/H+ antiporter subunit A
+分子 #3: Na(+)/H(+) antiporter subunit B
+分子 #4: Na(+)/H(+) antiporter subunit C
+分子 #5: Na+/H+ antiporter subunit E
+分子 #6: Na(+)/H(+) antiporter subunit F
+分子 #7: Na+/H+ antiporter subunit G1
+分子 #8: 1,2-Distearoyl-sn-glycerophosphoethanolamine
+分子 #9: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)