+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1412 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Interaction of decay-accelerating factor with coxsackievirus B3. | |||||||||
 マップデータ マップデータ | Surface rendered cvb3 complexed with DAF. Calculated map was used to scaled complexmap to 2.92 for fitting of DAF into the cryoEM density. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of lipopolysaccharide-mediated signaling pathway / negative regulation of complement activation / regulation of complement-dependent cytotoxicity / regulation of complement activation / respiratory burst / positive regulation of CD4-positive, alpha-beta T cell activation / positive regulation of CD4-positive, alpha-beta T cell proliferation / Class B/2 (Secretin family receptors) / ficolin-1-rich granule membrane / complement activation, classical pathway ...regulation of lipopolysaccharide-mediated signaling pathway / negative regulation of complement activation / regulation of complement-dependent cytotoxicity / regulation of complement activation / respiratory burst / positive regulation of CD4-positive, alpha-beta T cell activation / positive regulation of CD4-positive, alpha-beta T cell proliferation / Class B/2 (Secretin family receptors) / ficolin-1-rich granule membrane / complement activation, classical pathway / COPI-mediated anterograde transport / transport vesicle / side of membrane / endoplasmic reticulum-Golgi intermediate compartment membrane / secretory granule membrane / Regulation of Complement cascade / positive regulation of T cell cytokine production / virus receptor activity / positive regulation of cytosolic calcium ion concentration / membrane raft / Golgi membrane / innate immune response / lipid binding / Neutrophil degranulation / cell surface / extracellular exosome / extracellular region / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / coxsackievirus B3 passaged through rhabdomysarcoma cells rhabdomyosarcoma cells Homo sapiens (ヒト) / coxsackievirus B3 passaged through rhabdomysarcoma cells rhabdomyosarcoma cells | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 14.0 Å | |||||||||
 データ登録者 データ登録者 | Hafenstein S | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2007 ジャーナル: J Virol / 年: 2007タイトル: Interaction of decay-accelerating factor with coxsackievirus B3. 著者: Susan Hafenstein / Valorie D Bowman / Paul R Chipman / Carol M Bator Kelly / Feng Lin / M Edward Medof / Michael G Rossmann /  要旨: Many entero-, parecho-, and rhinoviruses use immunoglobulin (Ig)-like receptors that bind into the viral canyon and are required to initiate viral uncoating during infection. However, some of these ...Many entero-, parecho-, and rhinoviruses use immunoglobulin (Ig)-like receptors that bind into the viral canyon and are required to initiate viral uncoating during infection. However, some of these viruses use an alternative or additional receptor that binds outside the canyon. Both the coxsackievirus-adenovirus receptor (CAR), an Ig-like molecule that binds into the viral canyon, and decay-accelerating factor (DAF) have been identified as cellular receptors for coxsackievirus B3 (CVB3). A cryoelectron microscopy reconstruction of a variant of CVB3 complexed with DAF shows full occupancy of the DAF receptor in each of 60 binding sites. The DAF molecule bridges the canyon, blocking the CAR binding site and causing the two receptors to compete with one another. The binding site of DAF on CVB3 differs from the binding site of DAF on the surface of echoviruses, suggesting independent evolutionary processes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1412.map.gz emd_1412.map.gz | 2.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1412-v30.xml emd-1412-v30.xml emd-1412.xml emd-1412.xml | 11.9 KB 11.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1412.gif 1412.gif | 129.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1412 http://ftp.pdbj.org/pub/emdb/structures/EMD-1412 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1412 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1412 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1412.map.gz / 形式: CCP4 / 大きさ: 22.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1412.map.gz / 形式: CCP4 / 大きさ: 22.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Surface rendered cvb3 complexed with DAF. Calculated map was used to scaled complexmap to 2.92 for fitting of DAF into the cryoEM density. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.92 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
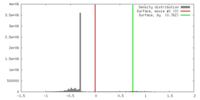
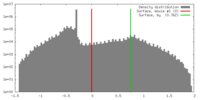
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : coxsackievirus B3, RD strain, complexed with decay-accelerating factor
| 全体 | 名称: coxsackievirus B3, RD strain, complexed with decay-accelerating factor |
|---|---|
| 要素 |
|
-超分子 #1000: coxsackievirus B3, RD strain, complexed with decay-accelerating factor
| 超分子 | 名称: coxsackievirus B3, RD strain, complexed with decay-accelerating factor タイプ: sample / ID: 1000 集合状態: one DAF binds each binding site, one per each protomer Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 7.0 MDa / 理論値: 7.0 MDa |
-超分子 #1: coxsackievirus B3 passaged through rhabdomysarcoma cells rhabdomy...
| 超分子 | 名称: coxsackievirus B3 passaged through rhabdomysarcoma cells rhabdomyosarcoma cells タイプ: virus / ID: 1 / Name.synonym: CVB3-RD 詳細: virus are saturated with one DAF bound at each possible site 生物種: coxsackievirus B3 passaged through rhabdomysarcoma cells rhabdomyosarcoma cells ウイルスタイプ: VIRION / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: CVB3-RD |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| 分子量 | 実験値: 7.0 MDa / 理論値: 7.0 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: VP1 / 直径: 300 Å / T番号(三角分割数): 1 |
| ウイルス殻 | Shell ID: 2 / 名称: VP2 / 直径: 300 Å / T番号(三角分割数): 1 |
| ウイルス殻 | Shell ID: 3 / 名称: VP3 / 直径: 300 Å / T番号(三角分割数): 1 |
-分子 #1: decay-accelerating factor
| 分子 | 名称: decay-accelerating factor / タイプ: protein_or_peptide / ID: 1 / Name.synonym: DAF 詳細: each molecule of DAF 70kD; DAF has His6 tag at c-terminus コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 実験値: 1.0 MDa / 理論値: 1.0 MDa |
| 組換発現 | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 6 / 詳細: 50mM MES |
| グリッド | 詳細: quantifoils |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 120 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: plunger / 手法: blot before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM300FEG/T |
|---|---|
| 温度 | 最低: 83 K / 最高: 83 K / 平均: 93 K |
| アライメント法 | Legacy - 非点収差: lens astigmatism was corrected at 98,000 times mag |
| 日付 | 2004年8月6日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 44 / 平均電子線量: 24 e/Å2 / 詳細: scanned at 7 microns and bin averaged to 14 / Od range: 1 / ビット/ピクセル: 8 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 300 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 倍率(補正後): 47000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.6 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 45000 |
| 試料ステージ | 試料ホルダー: side mounted nitrogen cooled / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | Virion were incubated with DAF at ratio of 4 DAF molecules per every potential binding site. |
|---|---|
| CTF補正 | 詳細: robEM |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 14.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMPFT, EM3DR / 使用した粒子像数: 2269 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera















 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)