+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-14066 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Rab GEF complex Mon1-Ccz1 | |||||||||
 マップデータ マップデータ | Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665; CtCcz1 aa1-796delta361-460). Map sharpened by Phenix local anisotropic filtering. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | guanine nucleotide exchange factor / TLD Rab GEF / longin domains / PIP binding / ENDOCYTOSIS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Mon1-Ccz1 complex / protein targeting to vacuole / multivesicular body membrane / fungal-type vacuole membrane / vesicle-mediated transport / autophagy 類似検索 - 分子機能 | |||||||||
| 生物種 |  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) | |||||||||
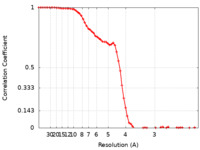
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.85 Å | |||||||||
 データ登録者 データ登録者 | Klink BU / Herrmann E | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Structure of the Mon1-Ccz1 complex reveals molecular basis of membrane binding for Rab7 activation. 著者: Björn U Klink / Eric Herrmann / Claudia Antoni / Lars Langemeyer / Stephan Kiontke / Christos Gatsogiannis / Christian Ungermann / Stefan Raunser / Daniel Kümmel /  要旨: Activation of the GTPase Rab7/Ypt7 by its cognate guanine nucleotide exchange factor (GEF) Mon1-Ccz1 marks organelles such as endosomes and autophagosomes for fusion with lysosomes/vacuoles and ...Activation of the GTPase Rab7/Ypt7 by its cognate guanine nucleotide exchange factor (GEF) Mon1-Ccz1 marks organelles such as endosomes and autophagosomes for fusion with lysosomes/vacuoles and degradation of their content. Here, we present a high-resolution cryogenic electron microscopy structure of the Mon1-Ccz1 complex that reveals its architecture in atomic detail. Mon1 and Ccz1 are arranged side by side in a pseudo-twofold symmetrical heterodimer. The three Longin domains of each Mon1 and Ccz1 are triangularly arranged, providing a strong scaffold for the catalytic center of the GEF. At the opposite side of the Ypt7-binding site, a positively charged and relatively flat patch stretches the Longin domains 2/3 of Mon1 and functions as a phosphatidylinositol phosphate-binding site, explaining how the GEF is targeted to membranes. Our work provides molecular insight into the mechanisms of endosomal Rab activation and serves as a blueprint for understanding the function of members of the Tri Longin domain Rab-GEF family. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14066.map.gz emd_14066.map.gz | 20.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14066-v30.xml emd-14066-v30.xml emd-14066.xml emd-14066.xml | 27.5 KB 27.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14066_fsc.xml emd_14066_fsc.xml | 8.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14066.png emd_14066.png | 77.3 KB | ||
| Filedesc metadata |  emd-14066.cif.gz emd-14066.cif.gz | 7.6 KB | ||
| その他 |  emd_14066_additional_1.map.gz emd_14066_additional_1.map.gz emd_14066_additional_2.map.gz emd_14066_additional_2.map.gz emd_14066_half_map_1.map.gz emd_14066_half_map_1.map.gz emd_14066_half_map_2.map.gz emd_14066_half_map_2.map.gz | 19.2 MB 20.8 MB 17 MB 17 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14066 http://ftp.pdbj.org/pub/emdb/structures/EMD-14066 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14066 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14066 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14066_validation.pdf.gz emd_14066_validation.pdf.gz | 679.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14066_full_validation.pdf.gz emd_14066_full_validation.pdf.gz | 678.7 KB | 表示 | |
| XML形式データ |  emd_14066_validation.xml.gz emd_14066_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_14066_validation.cif.gz emd_14066_validation.cif.gz | 16.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14066 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14066 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14066 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14066 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14066.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14066.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665; CtCcz1 aa1-796delta361-460). Map sharpened by Phenix local anisotropic filtering. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
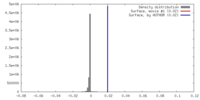
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665;...
| ファイル | emd_14066_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665; CtCcz1 aa1-796delta361-460). Map created by DeepEMhancer with high resolution training model. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
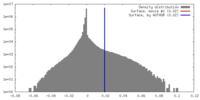
| 密度ヒストグラム |
-追加マップ: Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665;...
| ファイル | emd_14066_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665; CtCcz1 aa1-796delta361-460). Map created by Relion Postprocessing. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
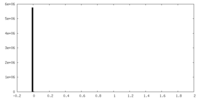
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665;...
| ファイル | emd_14066_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665; CtCcz1 aa1-796delta361-460). Unfiltered half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
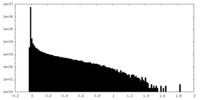
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665;...
| ファイル | emd_14066_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the Mon1-Ccz1 complex (CtMon1 aa141-665; CtCcz1 aa1-796delta361-460). Unfiltered half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rab GEF complex Mon1-Ccz1
| 全体 | 名称: Rab GEF complex Mon1-Ccz1 |
|---|---|
| 要素 |
|
-超分子 #1: Rab GEF complex Mon1-Ccz1
| 超分子 | 名称: Rab GEF complex Mon1-Ccz1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: CtMon1 aa141-665; CtCcz1 aa1-796delta361-460 |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
-分子 #1: Vacuolar fusion protein MON1
| 分子 | 名称: Vacuolar fusion protein MON1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 Chaetomium thermophilum (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 57.571832 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TGTAGDLASL LAGDGLGRKS KAWRVLRAQQ QACGEGSGEE GEITEIEGLG LGEGMEGFER ELDNIPDTLP DDERLALWKG KLKHYLILS SAGKPIWSRH GDLSLVNSTM GVVQTIISFY EGARNPLLGF TAGKVRFVIL IKGPLYFVAI SRLRESDAQL R AQLEALYM ...文字列: TGTAGDLASL LAGDGLGRKS KAWRVLRAQQ QACGEGSGEE GEITEIEGLG LGEGMEGFER ELDNIPDTLP DDERLALWKG KLKHYLILS SAGKPIWSRH GDLSLVNSTM GVVQTIISFY EGARNPLLGF TAGKVRFVIL IKGPLYFVAI SRLRESDAQL R AQLEALYM QILSTLTLPI LTNIFAHRPS TDLRGPLQGT ESLLASLADS FTKGSPSTLL SALECLRLRK SQRQAITNIF LK SRCEELL YGLLVAGGKL VSVIRPRKHS LHPSDLQLIF NMLFESGGIK GNGGENWIPL CLPAFNNTGY LYMYVSFLDD KAP DDQNQP PESSNLDASN KNSSNTPDDD LTALILISPS REAFYALQSM RTRLVSQLLS TGYLSLIRST ALSGRPSITS ILPK TPLLH FLYKSRPNVQ WCMSSLSSLT PPGATATETL LARRKLMSVY EELHAALHAR HAHLRVVYST ADEKEGEGLA CLGWS TPAF EVYCVAPGCV GRAGMAREVN RVVQWARREE ERLFILGGGV F UniProtKB: Vacuolar fusion protein MON1 |
-分子 #2: Ccz1
| 分子 | 名称: Ccz1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 Chaetomium thermophilum (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 75.780461 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTTPVSPSPS GIIPAQLGFL AIYNPALGTT DETLEDQIVY YATASTLSQA RRRHRRPRRR DRQRAQSVVK DSRPNAAGAT GDSEAVAED KDPVSKEERH ERLRQIGLAQ GMVEFAKSFS DGEPVDTIDT EKARVILVEV EEGWWILASI DLTRLPLPQI K TPTSSSAP ...文字列: MTTPVSPSPS GIIPAQLGFL AIYNPALGTT DETLEDQIVY YATASTLSQA RRRHRRPRRR DRQRAQSVVK DSRPNAAGAT GDSEAVAED KDPVSKEERH ERLRQIGLAQ GMVEFAKSFS DGEPVDTIDT EKARVILVEV EEGWWILASI DLTRLPLPQI K TPTSSSAP PPAPNLNPLP PEPAYEYSSR EVKPPSLLRA DLLRAYDLFL LHHGSSLSSL LASQGRAQLV ASLTRFWDHF LA TWNVLLH GNPACDVFGG IKLAASGELG IGVGEEERGS GEREVLEGLV ERVEGLVDVV VGRYGGPPSE KGPEEEQWLG LGG EVGEED GAVFLGVGAL DRKSLRGVVQ WMEEVYVWGE NAFGKPRRDL STGHFLLGLS ECSEEELTSS QANPKAIFVE LKPS YQHPS RKIPPEDPQP LGKVGPELPR DHTARLRPVI YVSQPFIYIL LFSEITPSPS TWPTLAESLH AQLSPLQKPL LHSTS YRPE RPVVETTSSS GTTTQHQIFD LVYDTETLTL QSTIPNIPDP FPYSATTPTG HSTGQQHHQQ SIWTRVEALQ THAQIL AIL SSGRAIPTDP SSFTHLPWEE GERTCKTARG WWIVWTRVVE HSPPDAVSLH HARDDDDNDD DASCSVLGHL RSVSSSH AA GSTSSSSGSG FGLGAIPGLG GLGGWAADGA TRLAQGIGID TRRYVEGLLT SLGR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.3 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 11916 / 平均露光時間: 15.0 sec. / 平均電子線量: 73.0 e/Å2 詳細: Images were collected in movie-mode with 4 frames per second |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 0.001 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7qla: |
 ムービー
ムービー コントローラー
コントローラー














 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)