登録情報 データベース : EMDB / ID : EMD-13831タイトル Cryo-EM structure of Ty3 retrotransposon targeting a TFIIIB-bound tRNA gene Cryo-EM map of Ty3 intasome targeting RNA Pol III transcribed gene 複合体 : Ty3 retrotransposon engaged with TFIIIB transcription factor and tRNA promoter複合体 : DNADNA : DNA (56-MER)DNA : DNA (31-MER)DNA : DNA (34-MER)DNA : DNA (9-MER)DNA : DNA (19-MER)複合体 : TFIIIB transcription factorタンパク質・ペプチド : Transposon Ty3-G Gag-Pol polyproteinタンパク質・ペプチド : Transcription factor TFIIIB component B''タンパク質・ペプチド : TATA-box-binding proteinタンパク質・ペプチド : Transcription factor IIIB 70 kDa subunit / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
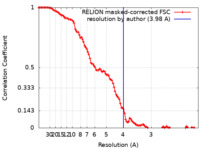
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Saccharomyces cerevisiae S288C (パン酵母)手法 / / 解像度 : 3.98 Å Abascal-Palacios G / Jochem L / Pla-Prats C / Beuron F / Vannini A 資金援助 Organization Grant number 国 Cancer Research UK CR-UK C47547/A21536 Wellcome Trust 200818/Z/16/Z
ジャーナル : Nat Commun / 年 : 2021タイトル : Structural basis of Ty3 retrotransposon integration at RNA Polymerase III-transcribed genes.著者 : Guillermo Abascal-Palacios / Laura Jochem / Carlos Pla-Prats / Fabienne Beuron / Alessandro Vannini / 要旨 : Retrotransposons are endogenous elements that have the ability to mobilise their DNA between different locations in the host genome. The Ty3 retrotransposon integrates with an exquisite specificity ... Retrotransposons are endogenous elements that have the ability to mobilise their DNA between different locations in the host genome. The Ty3 retrotransposon integrates with an exquisite specificity in a narrow window upstream of RNA Polymerase (Pol) III-transcribed genes, representing a paradigm for harmless targeted integration. Here we present the cryo-EM reconstruction at 4.0 Å of an active Ty3 strand transfer complex bound to TFIIIB transcription factor and a tRNA gene. The structure unravels the molecular mechanisms underlying Ty3 targeting specificity at Pol III-transcribed genes and sheds light into the architecture of retrotransposon machinery during integration. Ty3 intasome contacts a region of TBP, a subunit of TFIIIB, which is blocked by NC2 transcription regulator in RNA Pol II-transcribed genes. A newly-identified chromodomain on Ty3 integrase interacts with TFIIIB and the tRNA gene, defining with extreme precision the integration site position. 履歴 登録 2021年11月3日 - ヘッダ(付随情報) 公開 2022年1月26日 - マップ公開 2022年1月26日 - 更新 2024年11月13日 - 現状 2024年11月13日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 英国, 2件
英国, 2件  引用
引用 ジャーナル: Nat Commun / 年: 2021
ジャーナル: Nat Commun / 年: 2021


 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_13831.map.gz
emd_13831.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-13831-v30.xml
emd-13831-v30.xml emd-13831.xml
emd-13831.xml EMDBヘッダ
EMDBヘッダ emd_13831_fsc.xml
emd_13831_fsc.xml FSCデータファイル
FSCデータファイル emd_13831.png
emd_13831.png emd-13831.cif.gz
emd-13831.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-13831
http://ftp.pdbj.org/pub/emdb/structures/EMD-13831 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13831
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13831 emd_13831_validation.pdf.gz
emd_13831_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_13831_full_validation.pdf.gz
emd_13831_full_validation.pdf.gz emd_13831_validation.xml.gz
emd_13831_validation.xml.gz emd_13831_validation.cif.gz
emd_13831_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13831
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13831 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13831
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13831 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_13831.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_13831.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法 #1
電子顕微鏡法 #1 FIELD EMISSION GUN
FIELD EMISSION GUN
 電子顕微鏡法 #1~
電子顕微鏡法 #1~ FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)