+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | GA1 bacteriophage portal protein | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Bacteriophage / Portal protein / cryoEM / VIRAL PROTEIN | |||||||||||||||
| 機能・相同性 | Portal protein Gp10 / Portal protein Gp10 superfamily / Phage Connector (GP10) / Head-tail connector (Portal protein) 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Bacillus phage GA-1 (ファージ) Bacillus phage GA-1 (ファージ) | |||||||||||||||
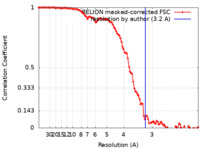
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||||||||
 データ登録者 データ登録者 | Javed A / Villanueva H | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2021 ジャーナル: Viruses / 年: 2021タイトル: Cryo-EM Structures of Two Bacteriophage Portal Proteins Provide Insights for Antimicrobial Phage Engineering. 著者: Abid Javed / Hugo Villanueva / Shadikejiang Shataer / Sara Vasciaveo / Renos Savva / Elena V Orlova /  要旨: Widespread antibiotic resistance has returned attention to bacteriophages as a means of managing bacterial pathogenesis. Synthetic biology approaches to engineer phages have demonstrated genomic ...Widespread antibiotic resistance has returned attention to bacteriophages as a means of managing bacterial pathogenesis. Synthetic biology approaches to engineer phages have demonstrated genomic editing to broaden natural host ranges, or to optimise microbicidal action. Gram positive pathogens cause serious pastoral animal and human infections that are especially lethal in newborns. Such pathogens are targeted by the obligate lytic phages of the and families. These phages have relatively small ~20 kb linear protein-capped genomes and their compact organisation, relatively few structural elements, and broad host range, are appealing from a phage-engineering standpoint. In this study, we focus on portal proteins, which are core elements for the assembly of such tailed phages. The structures of dodecameric portal complexes from phage GA1, which targets , and phage phiCPV4 that infects , were determined at resolutions of 3.3 Å and 2.9 Å, respectively. Both are found to closely resemble the related phi29 portal protein fold. However, the portal protein of phiCPV4 exhibits interesting differences in the clip domain. These structures provide new insights on structural diversity in portal proteins and will be essential for considerations in phage structural engineering. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13664.map.gz emd_13664.map.gz | 96.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13664-v30.xml emd-13664-v30.xml emd-13664.xml emd-13664.xml | 14.5 KB 14.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13664_fsc.xml emd_13664_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13664.png emd_13664.png | 97.2 KB | ||
| Filedesc metadata |  emd-13664.cif.gz emd-13664.cif.gz | 6.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13664 http://ftp.pdbj.org/pub/emdb/structures/EMD-13664 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13664 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13664 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13664_validation.pdf.gz emd_13664_validation.pdf.gz | 610.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13664_full_validation.pdf.gz emd_13664_full_validation.pdf.gz | 610.1 KB | 表示 | |
| XML形式データ |  emd_13664_validation.xml.gz emd_13664_validation.xml.gz | 11.8 KB | 表示 | |
| CIF形式データ |  emd_13664_validation.cif.gz emd_13664_validation.cif.gz | 15.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13664 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13664 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13664 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13664 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7pv2MC  7pv4C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13664.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13664.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : GA1 bacteriophage portal protein
| 全体 | 名称: GA1 bacteriophage portal protein |
|---|---|
| 要素 |
|
-超分子 #1: GA1 bacteriophage portal protein
| 超分子 | 名称: GA1 bacteriophage portal protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: A dodecamer complex made up of 12 subunits. |
|---|---|
| 由来(天然) | 生物種:  Bacillus phage GA-1 (ファージ) Bacillus phage GA-1 (ファージ) |
-分子 #1: Head-tail connector (Portal protein)
| 分子 | 名称: Head-tail connector (Portal protein) / タイプ: protein_or_peptide / ID: 1 詳細: In each subunit of the dodecamer, the DNA Tunnel loop residues (232 - 250) has not been modelled due to lack of EM density. Also missing in the pdb structure are 22 residues at the C-terminus ...詳細: In each subunit of the dodecamer, the DNA Tunnel loop residues (232 - 250) has not been modelled due to lack of EM density. Also missing in the pdb structure are 22 residues at the C-terminus and 7 residues at the N-terminus in each chain. コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Bacillus phage GA-1 (ファージ) Bacillus phage GA-1 (ファージ) |
| 分子量 | 理論値: 35.307801 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLEDGFSYKT IGEIQRRRGN LWFRTYQRYL FSLAYQMFEW QGLPKTVDPI FLEKQLHQRG FVAFYKDEMY GYLGVQGTLS GQINLYNQP NFYTASAPTY QKSFPLYWYD MGEDLNEKGQ GIVIYNNLER MPTLDILNLY AMNLAELKET IYVNQNAQKT P VIIKAGDN ...文字列: MLEDGFSYKT IGEIQRRRGN LWFRTYQRYL FSLAYQMFEW QGLPKTVDPI FLEKQLHQRG FVAFYKDEMY GYLGVQGTLS GQINLYNQP NFYTASAPTY QKSFPLYWYD MGEDLNEKGQ GIVIYNNLER MPTLDILNLY AMNLAELKET IYVNQNAQKT P VIIKAGDN DLFSMKQVYN KYEGNEPVIF AGKKFNTDDI EVLKTDAPYV ADKLTMLFKD QWNEAMTFLG LSNANTDKKE RL IQSEVES NNDQIQGSAN IYLAPRQEAC RLINEYYGLN VSVKLRKELV GNGELHNAIE GNSGMGNTV UniProtKB: Head-tail connector (Portal protein) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV / 詳細: 3ul of sample was applied on lacey carbon grids.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 95.0 K / 最高: 98.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-12 / 撮影したグリッド数: 1 / 実像数: 2249 / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)