+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13328 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Time-resolved cryo-EM structures of ATP-induced actomyosin dissociation | ||||||||||||
 マップデータ マップデータ | sharpened map of actomyosin from all timepoints combined | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  | ||||||||||||
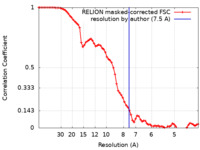
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 7.5 Å | ||||||||||||
 データ登録者 データ登録者 | Klebl DP / White HD / Sobott F / Muench SP | ||||||||||||
| 資金援助 | 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2021 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2021タイトル: On-grid and in-flow mixing for time-resolved cryo-EM. 著者: David P Klebl / Howard D White / Frank Sobott / Stephen P Muench /   要旨: Time-resolved cryo-electron microscopy (TrEM) allows the study of proteins under non-equilibrium conditions on the millisecond timescale, permitting the analysis of large-scale conformational changes ...Time-resolved cryo-electron microscopy (TrEM) allows the study of proteins under non-equilibrium conditions on the millisecond timescale, permitting the analysis of large-scale conformational changes or assembly and disassembly processes. However, the technique is developing and there have been few comparisons with other biochemical kinetic studies. Using current methods, the shortest time delay is on the millisecond timescale (∼5-10 ms), given by the delay between sample application and vitrification, and generating longer time points requires additional approaches such as using a longer delay line between the mixing element and nozzle, or an incubation step on the grid. To compare approaches, the reaction of ATP with the skeletal actomyosin S1 complex was followed on grids prepared with a 7-700 ms delay between mixing and vitrification. Classification of the cryo-EM data allows kinetic information to be derived which agrees with previous biochemical measurements, showing fast dissociation, low occupancy during steady-state hydrolysis and rebinding once ATP has been hydrolysed. However, this rebinding effect is much less pronounced when on-grid mixing is used and may be influenced by interactions with the air-water interface. Moreover, in-flow mixing results in a broader distribution of reaction times due to the range of velocities in a laminar flow profile (temporal spread), especially for longer time delays. This work shows the potential of TrEM, but also highlights challenges and opportunities for further development. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13328.map.gz emd_13328.map.gz | 1.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13328-v30.xml emd-13328-v30.xml emd-13328.xml emd-13328.xml | 35.8 KB 35.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13328_fsc.xml emd_13328_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13328.png emd_13328.png | 35.2 KB | ||
| マスクデータ |  emd_13328_msk_1.map emd_13328_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13328_additional_1.map.gz emd_13328_additional_1.map.gz emd_13328_additional_10.map.gz emd_13328_additional_10.map.gz emd_13328_additional_11.map.gz emd_13328_additional_11.map.gz emd_13328_additional_2.map.gz emd_13328_additional_2.map.gz emd_13328_additional_3.map.gz emd_13328_additional_3.map.gz emd_13328_additional_4.map.gz emd_13328_additional_4.map.gz emd_13328_additional_5.map.gz emd_13328_additional_5.map.gz emd_13328_additional_6.map.gz emd_13328_additional_6.map.gz emd_13328_additional_7.map.gz emd_13328_additional_7.map.gz emd_13328_additional_8.map.gz emd_13328_additional_8.map.gz emd_13328_additional_9.map.gz emd_13328_additional_9.map.gz emd_13328_half_map_1.map.gz emd_13328_half_map_1.map.gz emd_13328_half_map_2.map.gz emd_13328_half_map_2.map.gz | 23 MB 23.2 MB 23.2 MB 23.2 MB 23.3 MB 22.8 MB 2.2 MB 23 MB 23.3 MB 23 MB 22.9 MB 23 MB 23.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13328 http://ftp.pdbj.org/pub/emdb/structures/EMD-13328 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13328 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13328 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13328_validation.pdf.gz emd_13328_validation.pdf.gz | 423.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13328_full_validation.pdf.gz emd_13328_full_validation.pdf.gz | 423 KB | 表示 | |
| XML形式データ |  emd_13328_validation.xml.gz emd_13328_validation.xml.gz | 13.9 KB | 表示 | |
| CIF形式データ |  emd_13328_validation.cif.gz emd_13328_validation.cif.gz | 17.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13328 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13328 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13328 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13328 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13328.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13328.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map of actomyosin from all timepoints combined | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.13 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
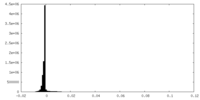
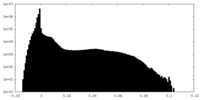
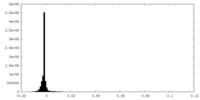
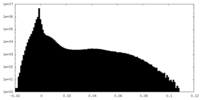
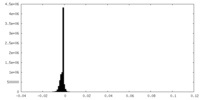
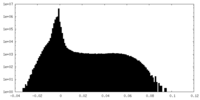
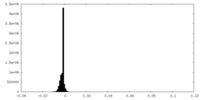
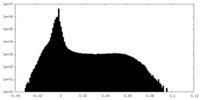
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
+マスク #1
+追加マップ: unsharpened reconstruction from all 13 ms particles
+追加マップ: unsharpened reconstruction from all 340 ms particles
+追加マップ: unsharpened reconstruction from all 400 ms particles
+追加マップ: unsharpened reconstruction from all 640 ms particles
+追加マップ: halfmap 2 of actin from all timepoints combined
+追加マップ: unsharpened map of actomyosin from all timepoints combined
+追加マップ: sharpened map of actin from all timepoints combined
+追加マップ: unsharpened map of actin from all timepoints combined
+追加マップ: halfmap 1 of actin from all timepoints combined
+追加マップ: unsharpened reconstruction from all 700 ms particles
+追加マップ: unsharpened reconstruction from all 7 ms particles
+ハーフマップ: halfmap 1 of actomyosin from all timepoints combined
+ハーフマップ: halfmap 2 of actomyosin from all timepoints combined
- 試料の構成要素
試料の構成要素
-全体 : Skeletal myosin subfragment 1 (A1 fraction) from rabbit in comple...
| 全体 | 名称: Skeletal myosin subfragment 1 (A1 fraction) from rabbit in complex with filamentous actin from rabbit |
|---|---|
| 要素 |
|
-超分子 #1: Skeletal myosin subfragment 1 (A1 fraction) from rabbit in comple...
| 超分子 | 名称: Skeletal myosin subfragment 1 (A1 fraction) from rabbit in complex with filamentous actin from rabbit タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 293 K / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 6 / 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X