[English] 日本語
 Yorodumi
Yorodumi- EMDB-13307: Cryo-EM structure of light harvesting complex 2 from Rba. sphaeroides. -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-13307 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of light harvesting complex 2 from Rba. sphaeroides. | |||||||||
 Map data Map data | A light harvesting complex 2 (LH2) from photosynthetic bacterium of Rba. spheroids. | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Photosynthesis / Purple bacteria / Light harvesting complex 2 / LH2 / Membrane protein / Cryo-EM. | |||||||||
| Function / homology |  Function and homology information Function and homology informationorganelle inner membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / : / photosynthesis, light reaction / metal ion binding / plasma membrane Similarity search - Function | |||||||||
| Biological species |  Cereibacter sphaeroides 2.4.1 (bacteria) Cereibacter sphaeroides 2.4.1 (bacteria) | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.1 Å | |||||||||
 Authors Authors | Qian P / Swainsbury DJK | |||||||||
| Funding support |  United Kingdom, 1 items United Kingdom, 1 items
| |||||||||
 Citation Citation |  Journal: Biochemistry / Year: 2021 Journal: Biochemistry / Year: 2021Title: Cryo-EM Structure of the Light-Harvesting 2 Complex at 2.1 Å. Authors: Pu Qian / David J K Swainsbury / Tristan I Croll / Pablo Castro-Hartmann / Giorgio Divitini / Kasim Sader / C Neil Hunter /   Abstract: Light-harvesting 2 (LH2) antenna complexes augment the collection of solar energy in many phototrophic bacteria. Despite its frequent role as a model for such complexes, there has been no three- ...Light-harvesting 2 (LH2) antenna complexes augment the collection of solar energy in many phototrophic bacteria. Despite its frequent role as a model for such complexes, there has been no three-dimensional (3D) structure available for the LH2 from the purple phototroph . We used cryo-electron microscopy (cryo-EM) to determine the 2.1 Å resolution structure of this LH2 antenna, which is a cylindrical assembly of nine αβ heterodimer subunits, each of which binds three bacteriochlorophyll (BChl) molecules and one carotenoid. The high resolution of this structure reveals all of the interpigment and pigment-protein interactions that promote the assembly and energy-transfer properties of this complex. Near the cytoplasmic face of the complex there is a ring of nine BChls, which absorb maximally at 800 nm and are designated as B800; each B800 is coordinated by the N-terminal carboxymethionine of LH2-α, part of a network of interactions with nearby residues on both LH2-α and LH2-β and with the carotenoid. Nine carotenoids, which are spheroidene in the strain we analyzed, snake through the complex, traversing the membrane and interacting with a ring of 18 BChls situated toward the periplasmic side of the complex. Hydrogen bonds with C-terminal aromatic residues modify the absorption of these pigments, which are red-shifted to 850 nm. Overlaps between the macrocycles of the B850 BChls ensure rapid transfer of excitation energy around this ring of pigments, which act as the donors of energy to neighboring LH2 and reaction center light-harvesting 1 (RC-LH1) complexes. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_13307.map.gz emd_13307.map.gz | 20.1 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-13307-v30.xml emd-13307-v30.xml emd-13307.xml emd-13307.xml | 16.1 KB 16.1 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_13307_fsc.xml emd_13307_fsc.xml | 13.5 KB | Display |  FSC data file FSC data file |
| Images |  emd_13307.png emd_13307.png | 63.8 KB | ||
| Filedesc metadata |  emd-13307.cif.gz emd-13307.cif.gz | 5.9 KB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13307 http://ftp.pdbj.org/pub/emdb/structures/EMD-13307 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13307 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13307 | HTTPS FTP |
-Validation report
| Summary document |  emd_13307_validation.pdf.gz emd_13307_validation.pdf.gz | 420.6 KB | Display |  EMDB validaton report EMDB validaton report |
|---|---|---|---|---|
| Full document |  emd_13307_full_validation.pdf.gz emd_13307_full_validation.pdf.gz | 420.2 KB | Display | |
| Data in XML |  emd_13307_validation.xml.gz emd_13307_validation.xml.gz | 13.2 KB | Display | |
| Data in CIF |  emd_13307_validation.cif.gz emd_13307_validation.cif.gz | 17.7 KB | Display | |
| Arichive directory |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13307 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13307 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13307 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13307 | HTTPS FTP |
-Related structure data
| Related structure data |  7pbwMC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_13307.map.gz / Format: CCP4 / Size: 209.3 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_13307.map.gz / Format: CCP4 / Size: 209.3 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | A light harvesting complex 2 (LH2) from photosynthetic bacterium of Rba. spheroids. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.66 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
- Sample components
Sample components
-Entire : Light harvesting complex 2
| Entire | Name: Light harvesting complex 2 |
|---|---|
| Components |
|
-Supramolecule #1: Light harvesting complex 2
| Supramolecule | Name: Light harvesting complex 2 / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#2 |
|---|---|
| Source (natural) | Organism:  Cereibacter sphaeroides 2.4.1 (bacteria) Cereibacter sphaeroides 2.4.1 (bacteria) |
| Molecular weight | Theoretical: 123.7 KDa |
-Macromolecule #1: Light-harvesting protein B-800/850 alpha chain
| Macromolecule | Name: Light-harvesting protein B-800/850 alpha chain / type: protein_or_peptide / ID: 1 / Number of copies: 9 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Cereibacter sphaeroides 2.4.1 (bacteria) Cereibacter sphaeroides 2.4.1 (bacteria) |
| Molecular weight | Theoretical: 5.275187 KDa |
| Sequence | String: (CXM)TNGKIWLVV KPTVGVPLFL SAAVIASVVI HAAVLTTTTW LPAYYQGSAA UniProtKB: Light-harvesting protein B-800/850 alpha chain |
-Macromolecule #2: Light-harvesting protein B-800/850 beta chain
| Macromolecule | Name: Light-harvesting protein B-800/850 beta chain / type: protein_or_peptide / ID: 2 / Number of copies: 9 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Cereibacter sphaeroides 2.4.1 (bacteria) Cereibacter sphaeroides 2.4.1 (bacteria) |
| Molecular weight | Theoretical: 4.99084 KDa |
| Sequence | String: LNKVWPSGLT VAEAEEVHKQ LILGTRVFGG MALIAHFLAA AATPWLG UniProtKB: Light-harvesting protein B-800/850 beta chain |
-Macromolecule #3: BACTERIOCHLOROPHYLL A
| Macromolecule | Name: BACTERIOCHLOROPHYLL A / type: ligand / ID: 3 / Number of copies: 27 / Formula: BCL |
|---|---|
| Molecular weight | Theoretical: 911.504 Da |
| Chemical component information |  ChemComp-BCL: |
-Macromolecule #4: SPHEROIDENE
| Macromolecule | Name: SPHEROIDENE / type: ligand / ID: 4 / Number of copies: 9 / Formula: 7OT |
|---|---|
| Molecular weight | Theoretical: 568.914 Da |
| Chemical component information | 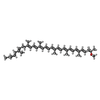 ChemComp-7OT: |
-Macromolecule #5: LAURYL DIMETHYLAMINE-N-OXIDE
| Macromolecule | Name: LAURYL DIMETHYLAMINE-N-OXIDE / type: ligand / ID: 5 / Number of copies: 36 / Formula: LDA |
|---|---|
| Molecular weight | Theoretical: 229.402 Da |
| Chemical component information |  ChemComp-LDA: |
-Macromolecule #6: CALCIUM ION
| Macromolecule | Name: CALCIUM ION / type: ligand / ID: 6 / Number of copies: 9 / Formula: CA |
|---|---|
| Molecular weight | Theoretical: 40.078 Da |
-Macromolecule #7: water
| Macromolecule | Name: water / type: ligand / ID: 7 / Number of copies: 230 / Formula: HOH |
|---|---|
| Molecular weight | Theoretical: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 4.5 mg/mL |
|---|---|
| Buffer | pH: 7.8 / Component - Concentration: 20.0 mmol / Component - Formula: C8H18N2O4S / Component - Name: Hepes Details: The final sample buffer: 20 mM Hepes, pH 7.8, 0.1%LDAO |
| Grid | Model: Quantifoil R1.2/1.3 / Material: COPPER / Mesh: 300 / Support film - Material: CARBON / Support film - topology: HOLEY ARRAY / Support film - Film thickness: 30 / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 60 sec. / Pretreatment - Atmosphere: AIR / Details: easiGlow was used for glow discharge. |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 277 K / Instrument: FEI VITROBOT MARK IV |
| Details | Sample was solubilised and purified with detergent of LDAO |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN |
|---|---|
| Specialist optics | Energy filter - Name: GIF Bioquantum / Energy filter - Slit width: 20 eV |
| Image recording | Film or detector model: GATAN K3 BIOQUANTUM (6k x 4k) / Number grids imaged: 1 / Number real images: 3138 / Average exposure time: 1.37 sec. / Average electron dose: 41.6 e/Å2 Details: A total doe 41.6 was fractioned into 40 frames, resulting in an electron fluency of 1.04 e/A2/frame. |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.2 µm / Nominal defocus min: 0.8 µm / Nominal magnification: 130000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
 Movie
Movie Controller
Controller









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)























