+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | polysome, ribosome pair (conformation 3) in Mycoplasma pneumoniae cells | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Mycoplasma pneumoniae (strain ATCC 29342 / M129) (バクテリア) Mycoplasma pneumoniae (strain ATCC 29342 / M129) (バクテリア) | |||||||||
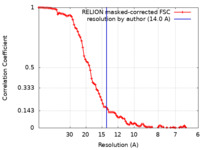
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 14.0 Å | |||||||||
 データ登録者 データ登録者 | Xue L / Lenz S / Rappsilber J / Mahamid J | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Visualizing translation dynamics at atomic detail inside a bacterial cell. 著者: Liang Xue / Swantje Lenz / Maria Zimmermann-Kogadeeva / Dimitry Tegunov / Patrick Cramer / Peer Bork / Juri Rappsilber / Julia Mahamid /    要旨: Translation is the fundamental process of protein synthesis and is catalysed by the ribosome in all living cells. Here we use advances in cryo-electron tomography and sub-tomogram analysis to ...Translation is the fundamental process of protein synthesis and is catalysed by the ribosome in all living cells. Here we use advances in cryo-electron tomography and sub-tomogram analysis to visualize the structural dynamics of translation inside the bacterium Mycoplasma pneumoniae. To interpret the functional states in detail, we first obtain a high-resolution in-cell average map of all translating ribosomes and build an atomic model for the M. pneumoniae ribosome that reveals distinct extensions of ribosomal proteins. Classification then resolves 13 ribosome states that differ in their conformation and composition. These recapitulate major states that were previously resolved in vitro, and reflect intermediates during active translation. On the basis of these states, we animate translation elongation inside native cells and show how antibiotics reshape the cellular translation landscapes. During translation elongation, ribosomes often assemble in defined three-dimensional arrangements to form polysomes. By mapping the intracellular organization of translating ribosomes, we show that their association into polysomes involves a local coordination mechanism that is mediated by the ribosomal protein L9. We propose that an extended conformation of L9 within polysomes mitigates collisions to facilitate translation fidelity. Our work thus demonstrates the feasibility of visualizing molecular processes at atomic detail inside cells. #1:  ジャーナル: Biorxiv / 年: 2021 ジャーナル: Biorxiv / 年: 2021タイトル: Visualizing translation dynamics at atomic detail inside a bacterial cell 著者: Xue L / Lenz S / Zimmermann-Kogadeeva M / Tegunov D / Cramer P / Bork P / Rappsilber J / Mahamid J | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13289.map.gz emd_13289.map.gz | 4.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13289-v30.xml emd-13289-v30.xml emd-13289.xml emd-13289.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13289_fsc.xml emd_13289_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13289.png emd_13289.png | 57.6 KB | ||
| マスクデータ |  emd_13289_msk_1.map emd_13289_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13289_half_map_1.map.gz emd_13289_half_map_1.map.gz emd_13289_half_map_2.map.gz emd_13289_half_map_2.map.gz | 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13289 http://ftp.pdbj.org/pub/emdb/structures/EMD-13289 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13289 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13289 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13289_validation.pdf.gz emd_13289_validation.pdf.gz | 800.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13289_full_validation.pdf.gz emd_13289_full_validation.pdf.gz | 800.2 KB | 表示 | |
| XML形式データ |  emd_13289_validation.xml.gz emd_13289_validation.xml.gz | 15.7 KB | 表示 | |
| CIF形式データ |  emd_13289_validation.cif.gz emd_13289_validation.cif.gz | 20.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13289 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13289 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13289 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13289 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7oocC  7oodC  7p6zC  7pahC  7paiC  7pajC  7pakC  7palC  7pamC  7panC  7paoC  7paqC  7parC  7pasC  7patC  7pauC  7ph9C  7phaC  7phbC  7phcC  7pi8C  7pi9C  7piaC  7pibC  7picC  7pioC  7pipC  7piqC  7pirC  7pisC  7pitC C: 同じ文献を引用 ( |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13289.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13289.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_13289_msk_1.map emd_13289_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13289_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13289_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : cryo-electron tomograms of untreated Mycoplasma pneumoniae cells
| 全体 | 名称: cryo-electron tomograms of untreated Mycoplasma pneumoniae cells |
|---|---|
| 要素 |
|
-超分子 #1: cryo-electron tomograms of untreated Mycoplasma pneumoniae cells
| 超分子 | 名称: cryo-electron tomograms of untreated Mycoplasma pneumoniae cells タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#55 詳細: The tomograms were collected with intact Mycoplasma pneumoniae cells. The sub-tomograms extracted in silico from cellular tomograms are with large box size to accommodate two adjacent ...詳細: The tomograms were collected with intact Mycoplasma pneumoniae cells. The sub-tomograms extracted in silico from cellular tomograms are with large box size to accommodate two adjacent ribosomes (one ribosome pair) with polysomes. |
|---|---|
| 由来(天然) | 生物種:  Mycoplasma pneumoniae (strain ATCC 29342 / M129) (バクテリア) Mycoplasma pneumoniae (strain ATCC 29342 / M129) (バクテリア) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE / 装置: HOMEMADE PLUNGER 詳細: back-side blotting for 2-3 second before plunging using a manual plunger without an environmental chamber. |
| 詳細 | Mycoplasma pneumoniae M129 cells grown on gold Quantifoil grids at 37 degrees Celsius before plunge freezing. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 3.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.75 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー









































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)