+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of human enterovirus 70 native virion | |||||||||
 マップデータ マップデータ | Native EV70 virion | |||||||||
 試料 試料 | Enterovirus D70 != Human enterovirus 70 (strain J670/71) Enterovirus D70
| |||||||||
 キーワード キーワード | EV70 / enterovirus 70 / virion / native / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport / DNA replication / RNA helicase activity / endocytosis involved in viral entry into host cell / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / virion attachment to host cell / host cell nucleus / structural molecule activity / ATP hydrolysis activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human enterovirus 70 (strain J670/71) (エンテロウイルス) Human enterovirus 70 (strain J670/71) (エンテロウイルス) | |||||||||
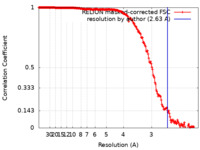
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.63 Å | |||||||||
 データ登録者 データ登録者 | Fuzik T / Plevka P | |||||||||
| 資金援助 |  チェコ, 1件 チェコ, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2022 ジャーナル: J Virol / 年: 2022タイトル: Structure of Human Enterovirus 70 and Its Inhibition by Capsid-Binding Compounds. 著者: Tibor Füzik / Jana Moravcová / Sergei Kalynych / Pavel Plevka /  要旨: Enterovirus 70 (EV70) is a human pathogen belonging to the family . EV70 is transmitted by eye secretions and causes acute hemorrhagic conjunctivitis, a serious eye disease. Despite the severity of ...Enterovirus 70 (EV70) is a human pathogen belonging to the family . EV70 is transmitted by eye secretions and causes acute hemorrhagic conjunctivitis, a serious eye disease. Despite the severity of the disease caused by EV70, its structure is unknown. Here, we present the structures of the EV70 virion, altered particle, and empty capsid determined by cryo-electron microscopy. The capsid of EV70 is composed of the subunits VP1, VP2, VP3, and VP4. The partially collapsed hydrophobic pocket located in VP1 of the EV70 virion is not occupied by a pocket factor, which is commonly present in other enteroviruses. Nevertheless, we show that the pocket can be targeted by the antiviral compounds WIN51711 and pleconaril, which block virus infection. The inhibitors prevent genome release by stabilizing EV70 particles. Knowledge of the structures of complexes of EV70 with inhibitors will enable the development of capsid-binding therapeutics against this virus. Globally distributed enterovirus 70 (EV70) causes local outbreaks of acute hemorrhagic conjunctivitis. The discharge from infected eyes enables the high-efficiency transmission of EV70 in overcrowded areas with low hygienic standards. Currently, only symptomatic treatments are available. We determined the structures of EV70 in its native form, the genome release intermediate, and the empty capsid resulting from genome release. Furthermore, we elucidated the structures of EV70 in complex with two inhibitors that block virus infection, and we describe the mechanism of their binding to the virus capsid. These results enable the development of therapeutics against EV70. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13022.map.gz emd_13022.map.gz | 42.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13022-v30.xml emd-13022-v30.xml emd-13022.xml emd-13022.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13022_fsc.xml emd_13022_fsc.xml | 18 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13022.png emd_13022.png | 329.5 KB | ||
| Filedesc metadata |  emd-13022.cif.gz emd-13022.cif.gz | 6.4 KB | ||
| その他 |  emd_13022_half_map_1.map.gz emd_13022_half_map_1.map.gz emd_13022_half_map_2.map.gz emd_13022_half_map_2.map.gz | 411.2 MB 411.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13022 http://ftp.pdbj.org/pub/emdb/structures/EMD-13022 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13022 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13022 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13022_validation.pdf.gz emd_13022_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13022_full_validation.pdf.gz emd_13022_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_13022_validation.xml.gz emd_13022_validation.xml.gz | 25.9 KB | 表示 | |
| CIF形式データ |  emd_13022_validation.cif.gz emd_13022_validation.cif.gz | 33.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13022 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13022 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13022 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13022 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13022.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13022.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Native EV70 virion | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.063 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_13022_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
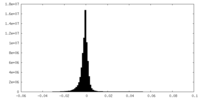
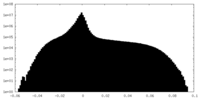
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13022_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
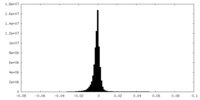
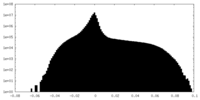
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Enterovirus D70
| 全体 | 名称: Enterovirus D70 |
|---|---|
| 要素 |
|
-超分子 #1: Human enterovirus 70 (strain J670/71)
| 超分子 | 名称: Human enterovirus 70 (strain J670/71) / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / 詳細: isolated from infected hTERT RPE1 cells / NCBI-ID: 31915 / 生物種: Human enterovirus 70 (strain J670/71) / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 分子量 | 理論値: 5.7 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid / 直径: 330.0 Å / T番号(三角分割数): 1 |
-分子 #1: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human enterovirus 70 (strain J670/71) (エンテロウイルス) Human enterovirus 70 (strain J670/71) (エンテロウイルス)株: J670/71 |
| 分子量 | 理論値: 34.049793 KDa |
| 配列 | 文字列: AATTQIGEIV KTVANTVESE IKAELGVIPS LNAVETGATS NTEPEEAIQT RTVINMHGTA ECLVENFLGR SALVCMRSFE YKNHSTSTS SIQKNFFIWT LNTRELVQIR RKMELFTYLR FDTEITIVPT LRLFSSSNVS FSGLPNLTLQ AMYVPTGARK P SSQDSFEW ...文字列: AATTQIGEIV KTVANTVESE IKAELGVIPS LNAVETGATS NTEPEEAIQT RTVINMHGTA ECLVENFLGR SALVCMRSFE YKNHSTSTS SIQKNFFIWT LNTRELVQIR RKMELFTYLR FDTEITIVPT LRLFSSSNVS FSGLPNLTLQ AMYVPTGARK P SSQDSFEW QSACNPSVFF KINDPPARLT IPFMSINSAY ANFYDGFAGF EKKATVLYGI NPANTMGNLC LRVVNSYQPV QY TLTVRVY MKPKHIKAWA PRAPRTMPYT NILNNNYAGR SAAPNAPTAI VSHRSTIKTM PNDINLTTA UniProtKB: Genome polyprotein |
-分子 #2: Capsid protein VP2
| 分子 | 名称: Capsid protein VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human enterovirus 70 (strain J670/71) (エンテロウイルス) Human enterovirus 70 (strain J670/71) (エンテロウイルス)株: J670/71 |
| 分子量 | 理論値: 27.536107 KDa |
| 配列 | 文字列: SPSAEACGYS DRVLQLKLGN SSIVTQEAAN ICCAYGEWPT YLPDNEAVAI DKPTQPETST DRFYTLKSKK WESNSTGWWW KLPDALNQI GMFGQNVQYH YLYRSGFLCH VQCNATKFHQ GTLLIVAIPE HQIGKKGTGT SASFAEVMKG AEGGVFEQPY L LDDGTSLA ...文字列: SPSAEACGYS DRVLQLKLGN SSIVTQEAAN ICCAYGEWPT YLPDNEAVAI DKPTQPETST DRFYTLKSKK WESNSTGWWW KLPDALNQI GMFGQNVQYH YLYRSGFLCH VQCNATKFHQ GTLLIVAIPE HQIGKKGTGT SASFAEVMKG AEGGVFEQPY L LDDGTSLA CALVYPHQWI NLRTNNSATI VLPWMNSAPM DFALRHNNWT LAVIPVCPLA GGTGNTNTYV PITISIAPMC AE YNGLRNA ITQ UniProtKB: Genome polyprotein |
-分子 #3: Capsid protein VP3
| 分子 | 名称: Capsid protein VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human enterovirus 70 (strain J670/71) (エンテロウイルス) Human enterovirus 70 (strain J670/71) (エンテロウイルス)株: J670/71 |
| 分子量 | 理論値: 26.717535 KDa |
| 配列 | 文字列: GVPTCLLPGS NQFLTTDDHS SAPAFPDFSP TPEMHIPGQV HSMLEIVQIE SMMEINNVND ASGVERLRVQ ISAQSDMDQL LFNIPLDIQ LEGPLRNTLL GNISRYYTHW SGSLEMTFMF CGSFMTTGKL IICYTPPGGS SPTDRMQAML ATHVVWDFGL Q SSITIIIP ...文字列: GVPTCLLPGS NQFLTTDDHS SAPAFPDFSP TPEMHIPGQV HSMLEIVQIE SMMEINNVND ASGVERLRVQ ISAQSDMDQL LFNIPLDIQ LEGPLRNTLL GNISRYYTHW SGSLEMTFMF CGSFMTTGKL IICYTPPGGS SPTDRMQAML ATHVVWDFGL Q SSITIIIP WISGSHYRMF NTDAKAINAN VGYVTCFMQT NLVAPVGAAD QCYIVGMVAA KKDFNLRLMR DSPDIGQSAI LP EQA UniProtKB: Genome polyprotein |
-分子 #4: Capsid protein VP4
| 分子 | 名称: Capsid protein VP4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human enterovirus 70 (strain J670/71) (エンテロウイルス) Human enterovirus 70 (strain J670/71) (エンテロウイルス)株: J670/71 |
| 分子量 | 理論値: 7.18676 KDa |
| 配列 | 文字列: GAQVSRQQTG THENANVATG GSSITYNQIN FYKDSYAASA SKQDFSQDPS KFTEPVAEAL KAGAPVLK UniProtKB: Genome polyprotein |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 150 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - 画像ごとのフレーム数: 1-16 / 撮影したグリッド数: 2 / 実像数: 6698 / 平均露光時間: 1.0 sec. / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Y (Sec.)
Y (Sec.) X (Row.)
X (Row.) Z (Col.)
Z (Col.)