+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12772 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle cryo-EM reconstruction of a 40-mer assembly of recombinant yeast Hsp26 mutant S47ET48E. | |||||||||
 マップデータ マップデータ | Cryo-EM single particle preliminary reconstruction of yeast Hsp26 mutant S47ET48E | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytoplasmic stress granule / unfolded protein binding / protein folding / cellular response to heat / mRNA binding / mitochondrion / identical protein binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
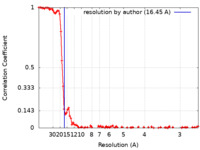
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 16.45 Å | |||||||||
 データ登録者 データ登録者 | Muehlhofer M / Peters C / Kriehuber T / Kreuzeder M / Kazman P / Rodina N / Reif B / Haslbeck M / Weinkauf S / Buchner J | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Phosphorylation activates the yeast small heat shock protein Hsp26 by weakening domain contacts in the oligomer ensemble. 著者: Moritz Mühlhofer / Carsten Peters / Thomas Kriehuber / Marina Kreuzeder / Pamina Kazman / Natalia Rodina / Bernd Reif / Martin Haslbeck / Sevil Weinkauf / Johannes Buchner /  要旨: Hsp26 is a small heat shock protein (sHsp) from S. cerevisiae. Its chaperone activity is activated by oligomer dissociation at heat shock temperatures. Hsp26 contains 9 phosphorylation sites in ...Hsp26 is a small heat shock protein (sHsp) from S. cerevisiae. Its chaperone activity is activated by oligomer dissociation at heat shock temperatures. Hsp26 contains 9 phosphorylation sites in different structural elements. Our analysis of phospho-mimetic mutations shows that phosphorylation activates Hsp26 at permissive temperatures. The cryo-EM structure of the Hsp26 40mer revealed contacts between the conserved core domain of Hsp26 and the so-called thermosensor domain in the N-terminal part of the protein, which are targeted by phosphorylation. Furthermore, several phosphorylation sites in the C-terminal extension, which link subunits within the oligomer, are sensitive to the introduction of negative charges. In all cases, the intrinsic inhibition of chaperone activity is relieved and the N-terminal domain becomes accessible for substrate protein binding. The weakening of domain interactions within and between subunits by phosphorylation to activate the chaperone activity in response to proteotoxic stresses independent of heat stress could be a general regulation principle of sHsps. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12772.map.gz emd_12772.map.gz | 43.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12772-v30.xml emd-12772-v30.xml emd-12772.xml emd-12772.xml | 11.9 KB 11.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12772_fsc.xml emd_12772_fsc.xml | 13.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12772.png emd_12772.png | 124.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12772 http://ftp.pdbj.org/pub/emdb/structures/EMD-12772 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12772 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12772 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12772_validation.pdf.gz emd_12772_validation.pdf.gz | 338.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12772_full_validation.pdf.gz emd_12772_full_validation.pdf.gz | 337.7 KB | 表示 | |
| XML形式データ |  emd_12772_validation.xml.gz emd_12772_validation.xml.gz | 11.8 KB | 表示 | |
| CIF形式データ |  emd_12772_validation.cif.gz emd_12772_validation.cif.gz | 15.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12772 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12772 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12772 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12772 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12772.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12772.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM single particle preliminary reconstruction of yeast Hsp26 mutant S47ET48E | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.33 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : 40mer complex of Hsp26 S47ET48E mutant
| 全体 | 名称: 40mer complex of Hsp26 S47ET48E mutant |
|---|---|
| 要素 |
|
-超分子 #1: 40mer complex of Hsp26 S47ET48E mutant
| 超分子 | 名称: 40mer complex of Hsp26 S47ET48E mutant / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 955.2 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 294 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 3042 / 平均露光時間: 10.0 sec. / 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)