+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12451 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the cytochrome bd oxidase from M. tuberculosis at 2.5 A resolution | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Terminal oxidase Oxidoreductase Oxygen reductase bd oxidase / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytochrome complex / aerobic electron transport chain / oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor / electron transfer activity / heme binding / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||||||||
 データ登録者 データ登録者 | Safarian S / Wu D | |||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  ニュージーランド, 4件 ニュージーランド, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: The cryo-EM structure of the bd oxidase from M. tuberculosis reveals a unique structural framework and enables rational drug design to combat TB. 著者: Schara Safarian / Helen K Opel-Reading / Di Wu / Ahmad R Mehdipour / Kiel Hards / Liam K Harold / Melanie Radloff / Ian Stewart / Sonja Welsch / Gerhard Hummer / Gregory M Cook / Kurt L ...著者: Schara Safarian / Helen K Opel-Reading / Di Wu / Ahmad R Mehdipour / Kiel Hards / Liam K Harold / Melanie Radloff / Ian Stewart / Sonja Welsch / Gerhard Hummer / Gregory M Cook / Kurt L Krause / Hartmut Michel /   要旨: New drugs are urgently needed to combat the global TB epidemic. Targeting simultaneously multiple respiratory enzyme complexes of Mycobacterium tuberculosis is regarded as one of the most effective ...New drugs are urgently needed to combat the global TB epidemic. Targeting simultaneously multiple respiratory enzyme complexes of Mycobacterium tuberculosis is regarded as one of the most effective treatment options to shorten drug administration regimes, and reduce the opportunity for the emergence of drug resistance. During infection and proliferation, the cytochrome bd oxidase plays a crucial role for mycobacterial pathophysiology by maintaining aerobic respiration at limited oxygen concentrations. Here, we present the cryo-EM structure of the cytochrome bd oxidase from M. tuberculosis at 2.5 Å. In conjunction with atomistic molecular dynamics (MD) simulation studies we discovered a previously unknown MK-9-binding site, as well as a unique disulfide bond within the Q-loop domain that defines an inactive conformation of the canonical quinol oxidation site in Actinobacteria. Our detailed insights into the long-sought atomic framework of the cytochrome bd oxidase from M. tuberculosis will form the basis for the design of highly specific drugs to act on this enzyme. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12451.map.gz emd_12451.map.gz | 39.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12451-v30.xml emd-12451-v30.xml emd-12451.xml emd-12451.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12451_fsc.xml emd_12451_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12451.png emd_12451.png | 135.4 KB | ||
| マスクデータ |  emd_12451_msk_1.map emd_12451_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12451.cif.gz emd-12451.cif.gz | 7 KB | ||
| その他 |  emd_12451_additional_1.map.gz emd_12451_additional_1.map.gz emd_12451_half_map_1.map.gz emd_12451_half_map_1.map.gz emd_12451_half_map_2.map.gz emd_12451_half_map_2.map.gz | 2.9 MB 44.2 MB 44.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12451 http://ftp.pdbj.org/pub/emdb/structures/EMD-12451 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12451 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12451 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12451_validation.pdf.gz emd_12451_validation.pdf.gz | 685.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12451_full_validation.pdf.gz emd_12451_full_validation.pdf.gz | 685 KB | 表示 | |
| XML形式データ |  emd_12451_validation.xml.gz emd_12451_validation.xml.gz | 14.4 KB | 表示 | |
| CIF形式データ |  emd_12451_validation.cif.gz emd_12451_validation.cif.gz | 21.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12451 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12451 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12451 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12451 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12451.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12451.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.837 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12451_msk_1.map emd_12451_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
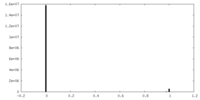
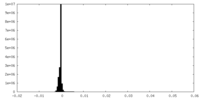
| 密度ヒストグラム |
-追加マップ: Sharpened Postprocess map
| ファイル | emd_12451_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened Postprocess map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
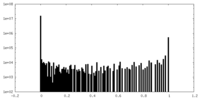
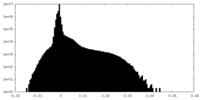
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_12451_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
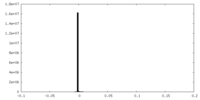
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12451_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
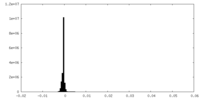
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of the cytochrome bd oxidase from M. tuberculos...
| 全体 | 名称: Cryo-EM structure of the cytochrome bd oxidase from M. tuberculosis at 2.5 A resolution |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of the cytochrome bd oxidase from M. tuberculos...
| 超分子 | 名称: Cryo-EM structure of the cytochrome bd oxidase from M. tuberculosis at 2.5 A resolution タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) |
| 分子量 | 理論値: 91.5 KDa |
-分子 #1: Probable integral membrane cytochrome D ubiquinol oxidase (Subuni...
| 分子 | 名称: Probable integral membrane cytochrome D ubiquinol oxidase (Subunit II) CydB (Cytochrome BD-I oxidase subunit II) タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) |
| 分子量 | 理論値: 37.650957 KDa |
| 組換発現 | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 配列 | 文字列: MVLQELWFGV IAALFLGFFI LEGFDFGVGM LMAPFAHVGM GDPETHRRTA LNTIGPVWDG NEVWLITAGA AIFAAFPGWY ATVFSALYL PLLAILFGMI LRAVAIEWRG KIDDPKWRTG ADFGIAAGSW LPALLWGVAF AILVRGLPVD ANGHVALSIP D VLNAYTLL ...文字列: MVLQELWFGV IAALFLGFFI LEGFDFGVGM LMAPFAHVGM GDPETHRRTA LNTIGPVWDG NEVWLITAGA AIFAAFPGWY ATVFSALYL PLLAILFGMI LRAVAIEWRG KIDDPKWRTG ADFGIAAGSW LPALLWGVAF AILVRGLPVD ANGHVALSIP D VLNAYTLL GGLATAGLFS LYGAVFIALK TSGPIRDDAY RFAVWLSLPV AGLVAGFGLW TQLAYGKDWT WLVLAVAGCA QA AATVLVW RRVSDGWAFM CTLIVVAAVV VLLFGALYPN LVPSTLNPQW SLTIHNASST PYTLKIMTWV TAFFAPLTVA YQT WTYWVF RQRISAERIP PPTGLARRAP UniProtKB: Probable integral membrane cytochrome D ubiquinol oxidase (Subunit II) CydB (Cytochrome BD-I oxidase subunit II) |
-分子 #2: Probable integral membrane cytochrome D ubiquinol oxidase (Subuni...
| 分子 | 名称: Probable integral membrane cytochrome D ubiquinol oxidase (Subunit I) CydA (Cytochrome BD-I oxidase subunit I) タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis H37Rv (結核菌) / 株: ATCC 25618 / H37Rv Mycobacterium tuberculosis H37Rv (結核菌) / 株: ATCC 25618 / H37Rv |
| 分子量 | 理論値: 53.863098 KDa |
| 組換発現 | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 配列 | 文字列: MNVVDISRWQ FGITTVYHFI FVPLTIGLAP LIAVMQTLWV VTDNPAWYRL TKFFGKLFLI NFAIGVATGI VQEFQFGMNW SEYSRFVGD VFGAPLAMEG LAAFFFESTF IGLWIFGWNR LPRLVHLACI WIVAIAVNVS AFFIIAANSF MQHPVGAHYN P TTGRAELS ...文字列: MNVVDISRWQ FGITTVYHFI FVPLTIGLAP LIAVMQTLWV VTDNPAWYRL TKFFGKLFLI NFAIGVATGI VQEFQFGMNW SEYSRFVGD VFGAPLAMEG LAAFFFESTF IGLWIFGWNR LPRLVHLACI WIVAIAVNVS AFFIIAANSF MQHPVGAHYN P TTGRAELS SIVVLLTNNT AQAAFTHTVS GALLTAGTFV AAVSAWWLVR SSTTHADSDT QAMYRPATIL GCWVALAATA GL LFTGDHQ GKLMFQQQPM KMASAESLCD TQTDPNFSVL TVGRQNNCDS LTRVIEVPYV LPFLAEGRIS GVTLQGIRDL QQE YQQRFG PNDYRPNLFV TYWSFRMMIG LMAIPVLFAL IALWLTRGGQ IPNQRWFSWL ALLTMPAPFL ANSAGWVFTE MGRQ PWVVV PNPTGDQLVR LTVKAGVSDH SATVVATSLL MFTLVYAVLA VIWCWLLKRY IVEGPLEHDA EPAAHGAPRD DEVAP LSFA Y UniProtKB: Probable integral membrane cytochrome D ubiquinol oxidase (Subunit I) CydA (Cytochrome BD-I oxidase subunit I) |
-分子 #3: OXYGEN MOLECULE
| 分子 | 名称: OXYGEN MOLECULE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: OXY |
|---|---|
| 分子量 | 理論値: 31.999 Da |
| Chemical component information |  ChemComp-O2: |
-分子 #4: MENAQUINONE-9
| 分子 | 名称: MENAQUINONE-9 / タイプ: ligand / ID: 4 / コピー数: 1 / 式: MQ9 |
|---|---|
| 分子量 | 理論値: 785.233 Da |
| Chemical component information | 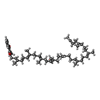 ChemComp-MQ9: |
-分子 #5: CIS-HEME D HYDROXYCHLORIN GAMMA-SPIROLACTONE
| 分子 | 名称: CIS-HEME D HYDROXYCHLORIN GAMMA-SPIROLACTONE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: HDD |
|---|---|
| 分子量 | 理論値: 632.487 Da |
| Chemical component information |  ChemComp-HDD: |
-分子 #6: HEME B/C
| 分子 | 名称: HEME B/C / タイプ: ligand / ID: 6 / コピー数: 2 / 式: HEB |
|---|---|
| 分子量 | 理論値: 618.503 Da |
| Chemical component information |  ChemComp-HEB: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 42 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 / 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 詳細: 15 mA | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot force 20. | |||||||||
| 詳細 | Monodisperse sample of cytochrome bd oxidase reconstituted in lipid nanodiscs 1D1 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 12070 / 平均露光時間: 5.0 sec. / 平均電子線量: 15.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 96000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 70 / 当てはまり具合の基準: Correlation coefficient | ||||||
| 得られたモデル |  PDB-7nkz: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)