+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11871 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | yeast THO-Sub2 complex dimer | |||||||||
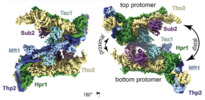
 マップデータ マップデータ | dimer map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | yeast THO complex S. cerevisiae THO-Sub2 the transcription-export (TREX) complex / RNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nucleoplasmic THO complex / cellular response to azide / THO complex / THO complex part of transcription export complex / positive regulation of transcription elongation by RNA polymerase I / transcription export complex / Cdc73/Paf1 complex / mRNA 3'-end processing / positive regulation of transcription by RNA polymerase I / subtelomeric heterochromatin formation ...nucleoplasmic THO complex / cellular response to azide / THO complex / THO complex part of transcription export complex / positive regulation of transcription elongation by RNA polymerase I / transcription export complex / Cdc73/Paf1 complex / mRNA 3'-end processing / positive regulation of transcription by RNA polymerase I / subtelomeric heterochromatin formation / mRNA export from nucleus / transcription-coupled nucleotide-excision repair / stress granule assembly / spliceosomal complex / transcription elongation by RNA polymerase II / helicase activity / mRNA splicing, via spliceosome / mRNA processing / DNA recombination / molecular adaptor activity / nucleic acid binding / chromosome, telomeric region / RNA helicase activity / RNA helicase / mRNA binding / ATP hydrolysis activity / RNA binding / ATP binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | Schuller SK / Schuller JM | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Structural insights into the nucleic acid remodeling mechanisms of the yeast THO-Sub2 complex. 著者: Sandra K Schuller / Jan M Schuller / J Rajan Prabu / Marc Baumgärtner / Fabien Bonneau / Jérôme Basquin / Elena Conti /  要旨: The yeast THO complex is recruited to active genes and interacts with the RNA-dependent ATPase Sub2 to facilitate the formation of mature export-competent messenger ribonucleoprotein particles and to ...The yeast THO complex is recruited to active genes and interacts with the RNA-dependent ATPase Sub2 to facilitate the formation of mature export-competent messenger ribonucleoprotein particles and to prevent the co-transcriptional formation of RNA:DNA-hybrid-containing structures. How THO-containing complexes function at the mechanistic level is unclear. Here, we elucidated a 3.4 Å resolution structure of THO-Sub2 by cryo-electron microscopy. THO subunits Tho2 and Hpr1 intertwine to form a platform that is bound by Mft1, Thp2, and Tex1. The resulting complex homodimerizes in an asymmetric fashion, with a Sub2 molecule attached to each protomer. The homodimerization interfaces serve as a fulcrum for a seesaw-like movement concomitant with conformational changes of the Sub2 ATPase. The overall structural architecture and topology suggest the molecular mechanisms of nucleic acid remodeling during mRNA biogenesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11871.map.gz emd_11871.map.gz | 97.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11871-v30.xml emd-11871-v30.xml emd-11871.xml emd-11871.xml | 23.4 KB 23.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11871.png emd_11871.png | 377.9 KB | ||
| Filedesc metadata |  emd-11871.cif.gz emd-11871.cif.gz | 8.2 KB | ||
| その他 |  emd_11871_additional_1.map.gz emd_11871_additional_1.map.gz emd_11871_additional_2.map.gz emd_11871_additional_2.map.gz | 87.2 MB 87 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11871 http://ftp.pdbj.org/pub/emdb/structures/EMD-11871 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11871 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11871 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11871_validation.pdf.gz emd_11871_validation.pdf.gz | 496.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11871_full_validation.pdf.gz emd_11871_full_validation.pdf.gz | 495.8 KB | 表示 | |
| XML形式データ |  emd_11871_validation.xml.gz emd_11871_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  emd_11871_validation.cif.gz emd_11871_validation.cif.gz | 7.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11871 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11871 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11871 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11871 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11871.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11871.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dimer map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.38 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
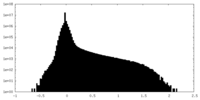
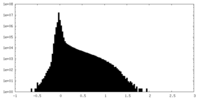
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Map Dimer Bottom Local 3.69 Angstrom
| ファイル | emd_11871_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map Dimer_Bottom Local_3.69 Angstrom | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
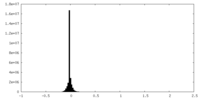
| 密度ヒストグラム |
-追加マップ: Map Dimer Bottom Local 4.01 Angstrom
| ファイル | emd_11871_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map Dimer_Bottom Local_4.01 Angstrom | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
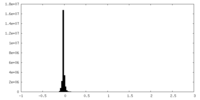
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : yeast THO-Sub2 complex dimer
| 全体 | 名称: yeast THO-Sub2 complex dimer |
|---|---|
| 要素 |
|
-超分子 #1: yeast THO-Sub2 complex dimer
| 超分子 | 名称: yeast THO-Sub2 complex dimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 862 kDa/nm |
-分子 #1: THO complex subunit 2
| 分子 | 名称: THO complex subunit 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 184.450016 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GAASMAEQTL LSKLNALSQK VIPPASPSQA SILTEEVIRN WPERSKTLCS DFTALESNDE KEDWLRTLFI ELFDFINKND ENSPLKLSD VASFTNELVN HERQVSQASI VGKMFIAVSS TVPNINDLTT ISLCKLIPSL HEELFKFSWI SSKLLNKEQT T LLRHLLKK ...文字列: GAASMAEQTL LSKLNALSQK VIPPASPSQA SILTEEVIRN WPERSKTLCS DFTALESNDE KEDWLRTLFI ELFDFINKND ENSPLKLSD VASFTNELVN HERQVSQASI VGKMFIAVSS TVPNINDLTT ISLCKLIPSL HEELFKFSWI SSKLLNKEQT T LLRHLLKK SKYELKKYNL LVENSVGYGQ LVALLILAYY DPDNFSKVSA YLKEIYHIMG KYSLDSIRTL DVILNVSSQF IT EGYKFFI ALLRKSDSWP SSHVANNSNY SSLNEGGNMI AANIISFNLS QYNEEVDKEN YERYMDMCCI LLKNGFVNFY SIW DNVKPE MEFLQEYIQN LETELEEEST KGVENPLAMA AALSTENETD EDNALVVNDD VNMKDKISEE TNADIESKGK QKTQ QDILL FGKIKLLERL LIHGCVIPVI HVLKQYPKVL YVSESLSRYL GRVFEYLLNP LYTSMTSSGE SKDMATALMI TRIDN GILA HKPRLIHKYK THEPFESLEL NSSYVFYYSE WNSNLTPFAS VNDLFENSHI YLSIIGPYLG RIPTLLSKIS RIGVAD IQK NHGSESLHVT IDKWIDYVRK FIFPATSLLQ NNPIATSEVY ELMKFFPFEK RYFIYNEMMT KLSQDILPLK VSFNKAE RE AKSILKALSI DTIAKESRRF AKLISTNPLA SLVPAVKQIE NYDKVSELVV YTTKYFNDFA YDVLQFVLLL RLTYNRPA V QFDGVNQAMW VQRLSIFIAG LAKNCPNMDI SNIITYILKT LHNGNIIAVS ILKELIITVG GIRDLNEVNM KQLLMLNSG SPLKQYARHL IYDFRDDNSV ISSRLTSFFT DQSAISEIIL LLYTLNLKAN TQNSHYKILS TRCDEMNTLL WSFIELIKHC LKGKAFEEN VLPFVELNNR FHLSTPWTFH IWRDYLDNQL NSNENFSIDE LIEGAEFSDV DLTKISKDLF TTFWRLSLYD I HFDKSLYD ERKNALSGEN TGHMSNRKKH LIQNQIKDIL VTGISHQRAF KKTSEFISEK SNVWNKDCGE DQIKIFLQNC VV PRVLFSP SDALFSSFFI FMAFRTENLM SILNTCITSN ILKTLLFCCT SSEAGNLGLF FTDVLKKLEK MRLNGDFNDQ ASR KLYEWH SVITEQVIDL LSEKNYMSIR NGIEFMKHVT SVFPVVKAHI QLVYTTLEEN LINEEREDIK LPSSALIGHL KARL KDALE LDEFCTLTEE EAEQKRIREM ELEEIKNYET ACQNEQKQVA LRKQLELNKS QRLQNDPPKS VASGSAGLNS KDRYT YSRN EPVIPTKPSS SQWSYSKVTR HVDDINHYLA TNHLQKAISL VENDDETRNL RKLSKQNMPI FDFRNSTLEI FERYFR TLI QNPQNPDFAE KIDSLKRYIK NISREPYPDT TSSYSEAAAP EYTKRSSRYS GNAGGKDGYG SSNYRGPSND RSAPKNI KP ISSYAHKRSE LPTRPSKSKT YNDRSRALRP TGPDRGDGFD QRDNRLREEY KKNSSQRSQL RFPEKPFQEG KDSSKANP Y QASSYKRDSP SENEEKPNKR FKKDETIRNK FQTQDYRNTR DSGAAHRANE NQRYNGNRKS NTQALPQGPK GGNYVSRYQ R UniProtKB: THO complex subunit 2 |
-分子 #2: THO complex subunit HPR1
| 分子 | 名称: THO complex subunit HPR1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 84.544047 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSNTEELIQN SIGFLQKTFK ALPVSFDSIR HEPLPSSMLH ASVLNFEWEP LEKNISAIHD RDSLIDIILK RFIIDSMTNA IEDEEENNL EKGLLNSCIG LDFVYNSRFN RSNPASWGNT FFELFSTIID LLNSPSTFLK FWPYAESRIE WFKMNTSVEP V SLGESNLI ...文字列: MSNTEELIQN SIGFLQKTFK ALPVSFDSIR HEPLPSSMLH ASVLNFEWEP LEKNISAIHD RDSLIDIILK RFIIDSMTNA IEDEEENNL EKGLLNSCIG LDFVYNSRFN RSNPASWGNT FFELFSTIID LLNSPSTFLK FWPYAESRIE WFKMNTSVEP V SLGESNLI SYKQPLYEKL RHWNDILAKL ENNDILNTVK HYNMKYKLEN FLSELLPINE ESNFNRSASI SALQESDNEW NR SARERES NRSSDVIFAA DYNFVFYHLI ICPIEFAFSD LEYKNDVDRS LSPLLDAILE IEENFYSKIK MNNRTRYSLE EAL NTEYYA NYDVMTPKLP VYMKHSNAMK MDRNEFWANL QNIKESDDYT LRPTIMDISL SNTTCLYKQL TQEDDDYYRK QFIL QLCFT TNLIRNLISS DETRNFYKSC YLRENPLSDI DFENLDEVNK KRGLNLCSYI CDNRVLKFYK IKDPDFYRVI RKLMS SDEK FTTAKIDGFK EFQNFRISKE KIPPPAFDET FKKFTFIKMG NKLINNVWKI PTGLDKIEQE VKKPEGVYEA AQAKWE SKI SSETSGGEAK DEIIRQWQTL RFLRSRYLFD FDKVNEKTGV DGLFEEPRKV EALDDSFKEK LLYKINQEHR KKLQDAR EY KIGKERKKRA LEEEASFPER EQKIKSQRIN SASQTEGDEL KSEQTQPKGE ISEENTKIKS SEVSSQDPDS GVAGEFAP UniProtKB: THO complex subunit HPR1 |
-分子 #3: TEX1 isoform 1
| 分子 | 名称: TEX1 isoform 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 42.315168 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSTIGAVDIL NQKTITSEVA ASVTSKYLQS TFSKGNTSHI EDKRFIHVSS RSHSRFTSTP ITPNEILSLK FHVSGSSMAY SRMDGSLTV WFIKDASFDK SVEVYIPDCC GSDKLATDLS WNPTSLNQIA VVSNSSEISL LLINEKSLTA SKLRTLSLGS K TKVNTCLY ...文字列: MSTIGAVDIL NQKTITSEVA ASVTSKYLQS TFSKGNTSHI EDKRFIHVSS RSHSRFTSTP ITPNEILSLK FHVSGSSMAY SRMDGSLTV WFIKDASFDK SVEVYIPDCC GSDKLATDLS WNPTSLNQIA VVSNSSEISL LLINEKSLTA SKLRTLSLGS K TKVNTCLY DPLGNWLLAA TKSEKIYLFD VKKDHSSVCS LNISDISQED NDVVYSLAWS NGGSHIFIGF KSGYLAILKA KH GILEVCT KIKAHTGPIT EIKMDPWGRN FITGSIDGNC YVWNMKSLCC ELIINDLNSA VTTLDVCHLG KILGICTEDE MVY FYDLNS GNLLHSKSLA NYKTDPVLKF YPDKSWYIMS GKNDTLSNHF VKNEKNLITY WKDM UniProtKB: TEX1 isoform 1 |
-分子 #4: EM14S01-3B_G0007820.mRNA.1.CDS.1
| 分子 | 名称: EM14S01-3B_G0007820.mRNA.1.CDS.1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.521004 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GAASDKKGSY VGIHSTGFKD FLLKPELSRA IIDCGFEHPS EVQQHTIPQS IHGTDVLCQA KSGLGKTAVF VLSTLQQLDP VPGEVAVVV ICNARELAYQ IRNEYLRFSK YMPDVKTAVF YGGTPISKDA ELLKNKDTAP HIVVATPGRL KALVREKYID L SHVKNFVI ...文字列: GAASDKKGSY VGIHSTGFKD FLLKPELSRA IIDCGFEHPS EVQQHTIPQS IHGTDVLCQA KSGLGKTAVF VLSTLQQLDP VPGEVAVVV ICNARELAYQ IRNEYLRFSK YMPDVKTAVF YGGTPISKDA ELLKNKDTAP HIVVATPGRL KALVREKYID L SHVKNFVI DECDKVLEEL DMRRDVQEIF RATPRDKQVM MFSATLSQEI RPICRRFLQN PLEIFVDDEA KLTLHGLQQY YI KLEEREK NRKLAQLLDD LEFNQVIIFV KSTTRANELT KLLNASNFPA ITVHGHMKQE ERIARYKAFK DFEKRICVST DVF GRGIDI ERINLAINYD LTNEADQYLH RVGRAGRFGT KGLAISFVSS KEDEEVLAKI QERFDVKIAE FPEEGIDPST YLNN UniProtKB: EM14S01-3B_G0007820.mRNA.1.CDS.1 |
-分子 #5: BJ4_G0025130.mRNA.1.CDS.1
| 分子 | 名称: BJ4_G0025130.mRNA.1.CDS.1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 30.340264 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTKEEGRTYF ESLCEEEQSL QESQTHLLNI LDILSVLADP RSSDDLLTES LKKLPDLHRE LINSSIRLRY DKYQTREAQL LEDTKTGRD VAAGVQNPKS ISEYYSTFEH LNRDTLRYIN LLKRLSVDLA KQVEVSDPSV TVYEMDKWVP SEKLQGILEQ Y CAPDTDIR ...文字列: MTKEEGRTYF ESLCEEEQSL QESQTHLLNI LDILSVLADP RSSDDLLTES LKKLPDLHRE LINSSIRLRY DKYQTREAQL LEDTKTGRD VAAGVQNPKS ISEYYSTFEH LNRDTLRYIN LLKRLSVDLA KQVEVSDPSV TVYEMDKWVP SEKLQGILEQ Y CAPDTDIR GVDAQIKNYL DQIKMARAKF GLENKYSLKE RLSTLTKELN HWRKEWDDIE MLMFGDDAHS MKKMIQKIDS LK SEINAPS ESYPVDKEGD IVLE UniProtKB: BJ4_G0002130.mRNA.1.CDS.1 |
-分子 #6: THO complex subunit MFT1
| 分子 | 名称: THO complex subunit MFT1 / タイプ: protein_or_peptide / ID: 6 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.055012 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPLSQKQIDQ VRTKVHYSEV DTPFNKYLDI LGKVTKLTGS IINGTLSNDD SKIEKLTEQN ISQLKESAHL RFLDLQSSID TKKVADENW ETCQQETLAK LENLKDKLPD IKSIHSKLLL RIGKLQGLYD SVQVINREVE GLSEGRTSLV VTRAEWEKEL G TDLVKFLI ...文字列: MPLSQKQIDQ VRTKVHYSEV DTPFNKYLDI LGKVTKLTGS IINGTLSNDD SKIEKLTEQN ISQLKESAHL RFLDLQSSID TKKVADENW ETCQQETLAK LENLKDKLPD IKSIHSKLLL RIGKLQGLYD SVQVINREVE GLSEGRTSLV VTRAEWEKEL G TDLVKFLI EKNYLKLVDP GLKKDSSEER YRIYDDFSKG PKELESINAS MKSDIENVRQ EVSSYKEKWL RDAEIFGKIT SI FKEELLK RDGLLNEAEG DNIDEDYESD EDEERKERFK RQRSMVEVNT IENVDEKEES DHEYDDQEDE ENEEEDDMEV DVE DIKEDN EVDGESSQQE DNSRQGNNEE TDKETGVIEE PDAVNDAEEA DSDHSSRKLG GTTSDFSASS SVEEVK UniProtKB: THO complex subunit MFT1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)