+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11613 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (monomer) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glutamate dehydrogenase / glutamate dehydrogenase (NAD+) activity / L-glutamate catabolic process / L-aspartate:2-oxoglutarate aminotransferase activity 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.59 Å | |||||||||
 データ登録者 データ登録者 | Lazaro M / Melero R / Huet C / Lopez-Alonso JP / Delgado S / Dodu A / Bruch EM / Abriata LA / Alzari PM / Valle M / Lisa MN | |||||||||
| 資金援助 |  スペイン, 1件 スペイン, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2021 ジャーナル: Commun Biol / 年: 2021タイトル: 3D architecture and structural flexibility revealed in the subfamily of large glutamate dehydrogenases by a mycobacterial enzyme. 著者: Melisa Lázaro / Roberto Melero / Charlotte Huet / Jorge P López-Alonso / Sandra Delgado / Alexandra Dodu / Eduardo M Bruch / Luciano A Abriata / Pedro M Alzari / Mikel Valle / María-Natalia Lisa /     要旨: Glutamate dehydrogenases (GDHs) are widespread metabolic enzymes that play key roles in nitrogen homeostasis. Large glutamate dehydrogenases composed of 180 kDa subunits (L-GDHs) contain long N- ...Glutamate dehydrogenases (GDHs) are widespread metabolic enzymes that play key roles in nitrogen homeostasis. Large glutamate dehydrogenases composed of 180 kDa subunits (L-GDHs) contain long N- and C-terminal segments flanking the catalytic core. Despite the relevance of L-GDHs in bacterial physiology, the lack of structural data for these enzymes has limited the progress of functional studies. Here we show that the mycobacterial L-GDH (mL-GDH) adopts a quaternary structure that is radically different from that of related low molecular weight enzymes. Intersubunit contacts in mL-GDH involve a C-terminal domain that we propose as a new fold and a flexible N-terminal segment comprising ACT-like and PAS-type domains that could act as metabolic sensors for allosteric regulation. These findings uncover unique aspects of the structure-function relationship in the subfamily of L-GDHs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11613.map.gz emd_11613.map.gz | 11.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11613-v30.xml emd-11613-v30.xml emd-11613.xml emd-11613.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11613.png emd_11613.png | 145.9 KB | ||
| マスクデータ |  emd_11613_msk_1.map emd_11613_msk_1.map | 209.3 MB |  マスクマップ マスクマップ | |
| その他 |  emd_11613_half_map_1.map.gz emd_11613_half_map_1.map.gz emd_11613_half_map_2.map.gz emd_11613_half_map_2.map.gz | 166 MB 166.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11613 http://ftp.pdbj.org/pub/emdb/structures/EMD-11613 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11613 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11613 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11613_validation.pdf.gz emd_11613_validation.pdf.gz | 417.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11613_full_validation.pdf.gz emd_11613_full_validation.pdf.gz | 416.8 KB | 表示 | |
| XML形式データ |  emd_11613_validation.xml.gz emd_11613_validation.xml.gz | 15 KB | 表示 | |
| CIF形式データ |  emd_11613_validation.cif.gz emd_11613_validation.cif.gz | 17.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11613 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11613 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11613 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11613 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11613.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11613.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11613_msk_1.map emd_11613_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
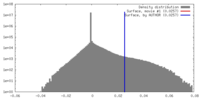
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11613_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
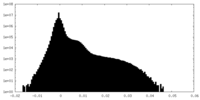
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11613_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
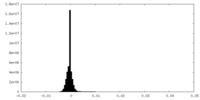
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM map of the large glutamate dehydrogenase composed of 180 ...
| 全体 | 名称: Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (monomer) |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM map of the large glutamate dehydrogenase composed of 180 ...
| 超分子 | 名称: Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (monomer) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 705 KDa |
-分子 #1: Large Glutamate Dehydrogenase
| 分子 | 名称: Large Glutamate Dehydrogenase / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HMHHHHHENL YFQGAASMIR RLSVAFLSTY RGPQADAPGV TSTGPLAVAA HDDLVSDDLV AAHYRLASMR APGETKAAVY PGDAGSGAAL QIVTDQAPML VDSVTVLLHR HGIAYTAIMN PVFRVRRGLD GELLDVRPAA EAAPGDGADE CWILVPITAA ADGEALTEAT ...文字列: HMHHHHHENL YFQGAASMIR RLSVAFLSTY RGPQADAPGV TSTGPLAVAA HDDLVSDDLV AAHYRLASMR APGETKAAVY PGDAGSGAAL QIVTDQAPML VDSVTVLLHR HGIAYTAIMN PVFRVRRGLD GELLDVRPAA EAAPGDGADE CWILVPITAA ADGEALTEAT RLVPGILAEA RQIGLDSGAM IAALHGLAND LATDLEGHFP NAERKEVAAL LRWLADGHFV LLGYQQCVVG DGNAEVDPAS RLGVLRLRND VLPPLTDSDD LLVLAQATMP SYLRYGAYPY IVVVRESPGA SRVIEHRFVG LFTVAAMNAN ALEIPLISRR VEEALAMAHR DPSHPGQLLR DIIQTIPRPE LFALSSKQLL EMALAVVDLG SRRRTLLFLR ADHLAHFVSC LVYLPRDRYT TAVRLEMQDI LVRELGGAGI DYSARVSESP WAVVHFTVRL PEGTAADSVD TSLENESRIQ DLLTEATRNW GDRMISAAAA ASISPAALEH YAHAFPEDYK QAFAPQDAIA DISLIEALQD DSVKLVLADT AEDRVWKLTW YLGGHSASLS ELLPMLQSMG VVVLEERPFT LRRTDGLPVW IYQFKISPHP SIPHAPDAEA QRDTAQRFAD AVTAIWHGRV EIDRFNELVM RAGLTWQQVV VLRAYAKYLR QAGFPYSQSH IESVLNENPH TTRSLIDLFE ALFDPSQETD GRRDAQGAAA AVAADIDALV SLDTDRVLRA FANLIEATLR TNYFVARPDS ARARNVLAFK LNPLVIKELP LPRPKFEIFV YSPRVEGVHL RFGFVARGGL RWSDRREDFR TEILGLVKAQ AVKNAVIVPV GAKGGFVVKR PPTLTGDAAA DREATRAEGV ECYRLFISGL LDVTDNVDKA TGAVVTPPEV VRRDGEDAYL VVAADKGTAT FSDIANEVAK SYGFWLGDAF ASGGSIGYDH KAMGITAKGA WESVKRHFRE MGVDTQTQDF TVVGIGDMSG DVFGNGMLLS KHIRLVAAFD HRDIFLDPNP DAGRSWDERK RLFDLPRSSW ADYDKSLISE GGGVYSRQQK SIPISPQVRT ALGLDADVEE LTPPALIKAI LKAPVDLLWN GGIGTYIKAE TEADADVGDR ANDQIRVCGN QVRAKVIGEG GNLGVTALGR IEFDLAGGRI NTDALDNSAG VDCSDHEVNI KILIDSAVTA GKVTPEERTE LLLSMTDEVG ELVLADNRDQ NDLMGTSRAN AASLLSVHAR MIKDLVDNRG LNRELEALPS EKEIRRRADA GIGLTSPELA TLMAHVKLAL KDDVLASDLP DQEVFASRLP YYFPTRLREE LHGEIRSHQL RREIITTMLV NDLVDTAGIS YAYRITEDVG VGPVDAVRSY VAINAIFGIG DVWRRIRAAG DAGVPTSVTD RMTLDLRRLV DRAGRWLLNY RPQPLAVGAE INRFGAKVAA LTPRMSEWLR GDDKAIVSKE AGDFASHGVP EDLAYHIATG LYQYSLLDVI DIADIVDREP DEVADTYFAL MDHLGADALL TAVSRLSRDD RWHSLARLAI RDDIYGSLRA LCFDVLAVGE PDENGEEKIA EWETTNSSRV TRARRTLTEI YKDGEQDLAT LSVAARQIRS MTRTSGTGTT G |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 6 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-20 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.2600000000000002 µm 最小 デフォーカス(補正後): 0.67 µm / 倍率(補正後): 47170 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: OTHER |
|---|
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)