+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1129 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Nucleotide-dependent bending flexibility of tubulin regulates microtubule assembly. | |||||||||
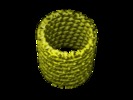
 マップデータ マップデータ | This is the reconstruction of the inner layer of the GDP-tubulin double-layered tube. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Wang HW / Nogales E | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2005 ジャーナル: Nature / 年: 2005タイトル: Nucleotide-dependent bending flexibility of tubulin regulates microtubule assembly. 著者: Hong-Wei Wang / Eva Nogales /  要旨: The atomic structure of tubulin in a polymerized, straight protofilament is clearly distinct from that in a curved conformation bound to a cellular depolymerizer. The nucleotide contents are ...The atomic structure of tubulin in a polymerized, straight protofilament is clearly distinct from that in a curved conformation bound to a cellular depolymerizer. The nucleotide contents are identical, and in both cases the conformation of the GTP-containing, intra-dimer interface is indistinguishable from the GDP-containing, inter-dimer contact. Here we present two structures corresponding to the start and end points in the microtubule polymerization and hydrolysis cycles that illustrate the consequences of nucleotide state on longitudinal and lateral assembly. In the absence of depolymerizers, GDP-bound tubulin shows distinctive intra-dimer and inter-dimer interactions and thus distinguishes the GTP and GDP interfaces. A cold-stable tubulin polymer with the non-hydrolysable GTP analogue GMPCPP, containing semi-conserved lateral interactions, supports a model in which the straightening of longitudinal interfaces happens sequentially, starting with a conformational change after GTP binding that straightens the dimer enough for the formation of lateral contacts into a non-tubular intermediate. Closure into a microtubule does not require GTP hydrolysis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1129.map.gz emd_1129.map.gz | 522.6 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1129-v30.xml emd-1129-v30.xml emd-1129.xml emd-1129.xml | 9.3 KB 9.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1129.gif 1129.gif | 36.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1129 http://ftp.pdbj.org/pub/emdb/structures/EMD-1129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1129 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1129.map.gz / 形式: CCP4 / 大きさ: 11 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1129.map.gz / 形式: CCP4 / 大きさ: 11 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is the reconstruction of the inner layer of the GDP-tubulin double-layered tube. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : GDP-tubulin
| 全体 | 名称: GDP-tubulin |
|---|---|
| 要素 |
|
-超分子 #1000: GDP-tubulin
| 超分子 | 名称: GDP-tubulin / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-分子 #1: tubulin
| 分子 | 名称: tubulin / タイプ: protein_or_peptide / ID: 1 / Name.synonym: tubulin / コピー数: 1 / 集合状態: dimer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 110 KDa / 理論値: 110 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 2.0 mg/mL |
|---|---|
| 緩衝液 | pH: 6.8 / 詳細: 80 mM PIPES, 1 mM MgCl2, 1mM GDP, 30 mM MnCl2 |
| グリッド | 詳細: 400 Quantifoil |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 90 K / 装置: OTHER / 詳細: Vitrification instrument: Vitrobot 手法: Blot for 1.8 seconds before plunging with Vitrobot offset as -2 mm |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 温度 | 平均: 90 K |
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at 200K mag |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 12.7 µm / 実像数: 200 / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 14 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 50000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | helices were formed from tubular crystals |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 12.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: MRC and home-made programs / 詳細: final maps were calculated from 19 tube images |
| CTF補正 | 詳細: each image |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















