+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11242 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Membrane domain of open complex I during turnover | |||||||||
 マップデータ マップデータ | Oversampled, local resolution-filtered map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | complex / respiration / NADH / proton pump / mitochondria / iron-sulphur cluster / oxidoreductase / membrane protein / ELECTRON TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / acyl binding / ubiquinone binding / electron transport coupled proton transport / acyl carrier activity / NADH:ubiquinone reductase (H+-translocating) / mitochondrial respiratory chain complex I assembly / mitochondrial electron transport, NADH to ubiquinone / respiratory chain complex I ...: / : / acyl binding / ubiquinone binding / electron transport coupled proton transport / acyl carrier activity / NADH:ubiquinone reductase (H+-translocating) / mitochondrial respiratory chain complex I assembly / mitochondrial electron transport, NADH to ubiquinone / respiratory chain complex I / membrane => GO:0016020 / NADH dehydrogenase (ubiquinone) activity / ATP synthesis coupled electron transport / reactive oxygen species metabolic process / electron transport chain / mitochondrial intermembrane space / mitochondrial inner membrane / mitochondrial matrix / mitochondrion 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
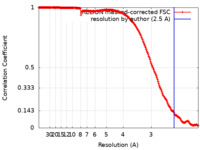
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Kampjut D / Sazanov LA | |||||||||
| 資金援助 | European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2020 ジャーナル: Science / 年: 2020タイトル: The coupling mechanism of mammalian respiratory complex I. 著者: Domen Kampjut / Leonid A Sazanov /  要旨: Mitochondrial complex I couples NADH:ubiquinone oxidoreduction to proton pumping by an unknown mechanism. Here, we present cryo-electron microscopy structures of ovine complex I in five different ...Mitochondrial complex I couples NADH:ubiquinone oxidoreduction to proton pumping by an unknown mechanism. Here, we present cryo-electron microscopy structures of ovine complex I in five different conditions, including turnover, at resolutions up to 2.3 to 2.5 angstroms. Resolved water molecules allowed us to experimentally define the proton translocation pathways. Quinone binds at three positions along the quinone cavity, as does the inhibitor rotenone that also binds within subunit ND4. Dramatic conformational changes around the quinone cavity couple the redox reaction to proton translocation during open-to-closed state transitions of the enzyme. In the induced deactive state, the open conformation is arrested by the ND6 subunit. We propose a detailed molecular coupling mechanism of complex I, which is an unexpected combination of conformational changes and electrostatic interactions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11242.map.gz emd_11242.map.gz | 173 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11242-v30.xml emd-11242-v30.xml emd-11242.xml emd-11242.xml | 47.7 KB 47.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11242_fsc.xml emd_11242_fsc.xml | 17.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11242.png emd_11242.png | 72.9 KB | ||
| Filedesc metadata |  emd-11242.cif.gz emd-11242.cif.gz | 11.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11242 http://ftp.pdbj.org/pub/emdb/structures/EMD-11242 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11242 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11242 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11242_validation.pdf.gz emd_11242_validation.pdf.gz | 189.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11242_full_validation.pdf.gz emd_11242_full_validation.pdf.gz | 189.1 KB | 表示 | |
| XML形式データ |  emd_11242_validation.xml.gz emd_11242_validation.xml.gz | 500 B | 表示 | |
| CIF形式データ |  emd_11242_validation.cif.gz emd_11242_validation.cif.gz | 449 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11242 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11242 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11242 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11242 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6zkaMC  6zk9C  6zkbC  6zkcC  6zkdC  6zkeC  6zkfC  6zkgC  6zkhC  6zkiC  6zkjC  6zkkC  6zklC  6zkmC  6zknC  6zkoC  6zkpC  6zkqC  6zkrC  6zksC  6zktC  6zkuC  6zkvC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11242.map.gz / 形式: CCP4 / 大きさ: 189.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11242.map.gz / 形式: CCP4 / 大きさ: 189.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Oversampled, local resolution-filtered map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
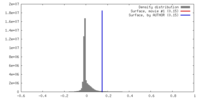
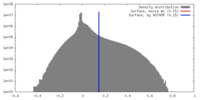
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Membrane domain of open complex I during turnover
+超分子 #1: Membrane domain of open complex I during turnover
+分子 #1: NADH-ubiquinone oxidoreductase chain 3
+分子 #2: NADH-ubiquinone oxidoreductase chain 1
+分子 #3: NADH-ubiquinone oxidoreductase chain 6
+分子 #4: NADH-ubiquinone oxidoreductase chain 4L
+分子 #5: NADH-ubiquinone oxidoreductase chain 5
+分子 #6: NADH-ubiquinone oxidoreductase chain 4
+分子 #7: NADH-ubiquinone oxidoreductase chain 2
+分子 #8: Mitochondrial complex I, B14.7 subunit
+分子 #9: NADH:ubiquinone oxidoreductase subunit B5
+分子 #10: Acyl carrier protein
+分子 #11: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 8
+分子 #12: Mitochondrial complex I, PDSW subunit
+分子 #13: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 10, mi...
+分子 #14: NADH:ubiquinone oxidoreductase subunit S5
+分子 #15: NADH:ubiquinone oxidoreductase subunit A3
+分子 #16: NADH:ubiquinone oxidoreductase subunit B3
+分子 #17: NADH dehydrogenase [ubiquinone] 1 subunit C2
+分子 #18: NADH:ubiquinone oxidoreductase subunit B4
+分子 #19: Mitochondrial complex I, B16.6 subunit
+分子 #20: Mitochondrial complex I, B17 subunit
+分子 #21: NADH:ubiquinone oxidoreductase subunit B7
+分子 #22: NADH:ubiquinone oxidoreductase subunit B9
+分子 #23: NADH:ubiquinone oxidoreductase subunit B2
+分子 #24: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 8, mito...
+分子 #25: Mitochondrial complex I, ESSS subunit
+分子 #26: Mitochondrial complex I, KFYI subunit
+分子 #27: Mitochondrial complex I, MNLL subunit
+分子 #28: Mitochondrial complex I, MWFE subunit
+分子 #29: Mitochondrial complex I, ND4L subunit
+分子 #30: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE
+分子 #31: 2-decyl-5,6-dimethoxy-3-methylcyclohexa-2,5-diene-1,4-dione
+分子 #32: 1,2-Distearoyl-sn-glycerophosphoethanolamine
+分子 #33: CARDIOLIPIN
+分子 #34: S-[2-({N-[(2S)-2-hydroxy-3,3-dimethyl-4-(phosphonooxy)butanoyl]-b...
+分子 #35: ADENOSINE MONOPHOSPHATE
+分子 #36: MYRISTIC ACID
+分子 #37: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R0.6/1 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 100.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)