+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11221 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the regulatory linker of ALC1 bound to the nucleosome's acidic patch: hexasome class. | |||||||||
 マップデータ マップデータ | Map filtered by local resolution from RELION LocalRes procedure. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ALC1 / CHD1L / chromatin remodeler / DNA damage response / nucleosome / hexasome / NUCLEAR PROTEIN / GENE REGULATION / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報poly-ADP-D-ribose modification-dependent protein binding / ATP-dependent chromatin remodeler activity / histone reader activity / site of DNA damage / nucleosome binding / DNA helicase activity / Dual Incision in GG-NER / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / Formation of Incision Complex in GG-NER / structural constituent of chromatin ...poly-ADP-D-ribose modification-dependent protein binding / ATP-dependent chromatin remodeler activity / histone reader activity / site of DNA damage / nucleosome binding / DNA helicase activity / Dual Incision in GG-NER / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / Formation of Incision Complex in GG-NER / structural constituent of chromatin / nucleosome / heterochromatin formation / nucleosome assembly / site of double-strand break / chromatin remodeling / protein heterodimerization activity / DNA repair / nucleotide binding / DNA damage response / ATP hydrolysis activity / DNA binding / nucleoplasm / ATP binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Bacic L / Gaullier G / Deindl S | |||||||||
| 資金援助 |  スウェーデン, 1件 スウェーデン, 1件
| |||||||||
 引用 引用 | ジャーナル: Protein Sci / 年: 2018 タイトル: UCSF ChimeraX: Meeting modern challenges in visualization and analysis. 著者: Thomas D Goddard / Conrad C Huang / Elaine C Meng / Eric F Pettersen / Gregory S Couch / John H Morris / Thomas E Ferrin /  要旨: UCSF ChimeraX is next-generation software for the visualization and analysis of molecular structures, density maps, 3D microscopy, and associated data. It addresses challenges in the size, scope, and ...UCSF ChimeraX is next-generation software for the visualization and analysis of molecular structures, density maps, 3D microscopy, and associated data. It addresses challenges in the size, scope, and disparate types of data attendant with cutting-edge experimental methods, while providing advanced options for high-quality rendering (interactive ambient occlusion, reliable molecular surface calculations, etc.) and professional approaches to software design and distribution. This article highlights some specific advances in the areas of visualization and usability, performance, and extensibility. ChimeraX is free for noncommercial use and is available from http://www.rbvi.ucsf.edu/chimerax/ for Windows, Mac, and Linux. #2:  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2018 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2018タイトル: ISOLDE: a physically realistic environment for model building into low-resolution electron-density maps. 著者: Croll TI #3:  ジャーナル: IUCrJ / 年: 2020 ジャーナル: IUCrJ / 年: 2020タイトル: Estimation of high-order aberrations and anisotropic magnification from cryo-EM data sets in 著者: Zivanov J / Nakane T / Scheres SHW #4:  ジャーナル: J. Struct. Biol. / 年: 2012 ジャーナル: J. Struct. Biol. / 年: 2012タイトル: RELION: implementation of a Bayesian approach to cryo-EM structure determination. 著者: Scheres SH | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11221.map.gz emd_11221.map.gz | 67.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11221-v30.xml emd-11221-v30.xml emd-11221.xml emd-11221.xml | 41.1 KB 41.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11221_fsc.xml emd_11221_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11221.png emd_11221.png | 246 KB | ||
| マスクデータ |  emd_11221_msk_1.map emd_11221_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-11221.cif.gz emd-11221.cif.gz | 8.2 KB | ||
| その他 |  emd_11221_additional_1.map.gz emd_11221_additional_1.map.gz emd_11221_additional_2.map.gz emd_11221_additional_2.map.gz emd_11221_additional_3.map.gz emd_11221_additional_3.map.gz emd_11221_half_map_1.map.gz emd_11221_half_map_1.map.gz emd_11221_half_map_2.map.gz emd_11221_half_map_2.map.gz | 80.5 MB 96.5 MB 10.3 MB 80.9 MB 80.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11221 http://ftp.pdbj.org/pub/emdb/structures/EMD-11221 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11221 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11221 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11221_validation.pdf.gz emd_11221_validation.pdf.gz | 887.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11221_full_validation.pdf.gz emd_11221_full_validation.pdf.gz | 886.8 KB | 表示 | |
| XML形式データ |  emd_11221_validation.xml.gz emd_11221_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_11221_validation.cif.gz emd_11221_validation.cif.gz | 23.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11221 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11221 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11221 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11221 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6zhyMC  6zhxC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10465 (タイトル: Single-particle cryo-EM dataset of the complex between a nucleosome and the regulatory linker of ALC1 EMPIAR-10465 (タイトル: Single-particle cryo-EM dataset of the complex between a nucleosome and the regulatory linker of ALC1Data size: 6.8 TB Data #1: Unaligned movies of the nucleosome - ALC1-linker complex - session_1_disk_1 [micrographs - multiframe] Data #2: Unaligned movies of the nucleosome - ALC1-linker complex - session_1_disk_2 [micrographs - multiframe] Data #3: Unaligned movies of the nucleosome - ALC1-linker complex - session_1_disk_3 [micrographs - multiframe] Data #4: Unaligned movies of the nucleosome - ALC1-linker complex - session_2_grid_1 [micrographs - multiframe] Data #5: Unaligned movies of the nucleosome - ALC1-linker complex - session_2_grid_2 [micrographs - multiframe] Data #6: Particles of the high-resolution nucleosome class [picked particles - multiframe - processed] Data #7: Particles of the hexasome class [picked particles - multiframe - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11221.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11221.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map filtered by local resolution from RELION LocalRes procedure. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.654 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11221_msk_1.map emd_11221_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
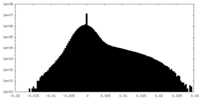
| 密度ヒストグラム |
-追加マップ: Map from final 3D refinement in RELION.
| ファイル | emd_11221_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map from final 3D refinement in RELION. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map from RELION PostProcess.
| ファイル | emd_11221_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map from RELION PostProcess. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map from RELION PostProcess with masking.
| ファイル | emd_11221_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map from RELION PostProcess with masking. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 1 from final 3D refinement in RELION.
| ファイル | emd_11221_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 from final 3D refinement in RELION. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 1 from final 3D refinement in RELION.
| ファイル | emd_11221_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 from final 3D refinement in RELION. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : A hexasome (nucleosome lacking one of the two H2A-H2B dimers) wit...
+超分子 #1: A hexasome (nucleosome lacking one of the two H2A-H2B dimers) wit...
+超分子 #2: Histones
+超分子 #3: DNA
+超分子 #4: Chromodomain-helicase-DNA-binding protein 1-like
+分子 #1: Histone H3
+分子 #2: Histone H4
+分子 #3: Histone H2A type 1
+分子 #4: Histone H2B 1.1
+分子 #7: Chromodomain-helicase-DNA-binding protein 1-like
+分子 #5: DNA (110-MER) Widom 601 sequence
+分子 #6: DNA (110-MER) Widom 601 sequence
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER / 詳細: current 20 mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Blot time 2.5 s, blot force 0. Two sample applications and blots were performed before vitrification.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 3 / 実像数: 19897 / 平均電子線量: 50.4 e/Å2 詳細: Movies were collected in counting mode. Total exposure was fractionated over 60 movie frames. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT 当てはまり具合の基準: Real-space CC between model and map | ||||||||||||||||||||
| 得られたモデル |  PDB-6zhy: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)