+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11111 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human Adenoviurs-F 41 at pH 4.0. | |||||||||||||||
 マップデータ マップデータ | Full unmasked Map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 生物種 |  Human adenovirus 41 (ヒトアデノウイルス) Human adenovirus 41 (ヒトアデノウイルス) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.0 Å | |||||||||||||||
 データ登録者 データ登録者 | Carlson L-A / Rafie K | |||||||||||||||
| 資金援助 |  スウェーデン, 4件 スウェーデン, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: The structure of enteric human adenovirus 41-A leading cause of diarrhea in children. 著者: K Rafie / A Lenman / J Fuchs / A Rajan / N Arnberg / L-A Carlson /   要旨: Human adenovirus (HAdV) types F40 and F41 are a prominent cause of diarrhea and diarrhea-associated mortality in young children worldwide. These enteric HAdVs differ notably in tissue tropism and ...Human adenovirus (HAdV) types F40 and F41 are a prominent cause of diarrhea and diarrhea-associated mortality in young children worldwide. These enteric HAdVs differ notably in tissue tropism and pathogenicity from respiratory and ocular adenoviruses, but the structural basis for this divergence has been unknown. Here, we present the first structure of an enteric HAdV-HAdV-F41-determined by cryo-electron microscopy to a resolution of 3.8 Å. The structure reveals extensive alterations to the virion exterior as compared to nonenteric HAdVs, including a unique arrangement of capsid protein IX. The structure also provides new insights into conserved aspects of HAdV architecture such as a proposed location of core protein V, which links the viral DNA to the capsid, and assembly-induced conformational changes in the penton base protein. Our findings provide the structural basis for adaptation of enteric HAdVs to a fundamentally different tissue tropism. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11111.map.gz emd_11111.map.gz | 5.6 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11111-v30.xml emd-11111-v30.xml emd-11111.xml emd-11111.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11111.png emd_11111.png | 198.5 KB | ||
| その他 |  emd_11111_additional_1.map.gz emd_11111_additional_1.map.gz emd_11111_half_map_1.map.gz emd_11111_half_map_1.map.gz emd_11111_half_map_2.map.gz emd_11111_half_map_2.map.gz | 1.1 GB 5.7 GB 5.7 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11111 http://ftp.pdbj.org/pub/emdb/structures/EMD-11111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11111 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11111_validation.pdf.gz emd_11111_validation.pdf.gz | 513.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11111_full_validation.pdf.gz emd_11111_full_validation.pdf.gz | 513.3 KB | 表示 | |
| XML形式データ |  emd_11111_validation.xml.gz emd_11111_validation.xml.gz | 30 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11111 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11111 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11111.map.gz / 形式: CCP4 / 大きさ: 6.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11111.map.gz / 形式: CCP4 / 大きさ: 6.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Full unmasked Map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.041 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
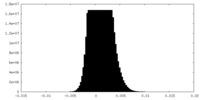
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Full masked Map
| ファイル | emd_11111_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Full masked Map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
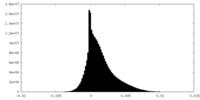
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_11111_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
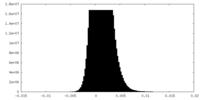
| 密度ヒストグラム |
-ハーフマップ: Half Map
| ファイル | emd_11111_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
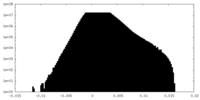
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human adenovirus 41
| 全体 | 名称:  Human adenovirus 41 (ヒトアデノウイルス) Human adenovirus 41 (ヒトアデノウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human adenovirus 41
| 超分子 | 名称: Human adenovirus 41 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: The HAdVF-41 virus was propagated and isolated from A549 cells. NCBI-ID: 10524 / 生物種: Human adenovirus 41 / ウイルスタイプ: VIRION / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| ウイルス殻 | Shell ID: 1 / 直径: 900.0 Å |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.6 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 4 構成要素:
詳細: The base buffer was PBS, prepared by using a commercial PBS tablet. Medicargo, PBS tablets, 09-9400-100 The last two components were at pH 3.4 before addition to the virus sample (pH 7.4) to ...詳細: The base buffer was PBS, prepared by using a commercial PBS tablet. Medicargo, PBS tablets, 09-9400-100 The last two components were at pH 3.4 before addition to the virus sample (pH 7.4) to reduce the pH of the virus solution to pH 4.0. | ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 0.037 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 295.15 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | The sample was monodisperse with a preferred orientation in the vitrious ice. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 2-20 / 撮影したグリッド数: 1 / 実像数: 3730 / 平均電子線量: 2.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.7 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)