+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1109 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural insights into the activity of enhancer-binding proteins. | |||||||||
 マップデータ マップデータ | this is a volume map of PspF in complex with sigma54 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | RNA polymerase sigma factor 54 / AAA+ ATPase domain 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Rappas M / Schumacher J / Beuron F / Niwa H / Bordes P / Wigneshweraraj S / Keetch CA / Robinson CV / Buck M / Zhang X | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2005 ジャーナル: Science / 年: 2005タイトル: Structural insights into the activity of enhancer-binding proteins. 著者: Mathieu Rappas / Jorg Schumacher / Fabienne Beuron / Hajime Niwa / Patricia Bordes / Sivaramesh Wigneshweraraj / Catherine A Keetch / Carol V Robinson / Martin Buck / Xiaodong Zhang /  要旨: Activators of bacterial sigma54-RNA polymerase holoenzyme are mechanochemical proteins that use adenosine triphosphate (ATP) hydrolysis to activate transcription. We have determined by cryogenic ...Activators of bacterial sigma54-RNA polymerase holoenzyme are mechanochemical proteins that use adenosine triphosphate (ATP) hydrolysis to activate transcription. We have determined by cryogenic electron microscopy (cryo-EM) a 20 angstrom resolution structure of an activator, phage shock protein F [PspF(1-275)], which is bound to an ATP transition state analog in complex with its basal factor, sigma54. By fitting the crystal structure of PspF(1-275) at 1.75 angstroms into the EM map, we identified two loops involved in binding sigma54. Comparing enhancer-binding structures in different nucleotide states and mutational analysis led us to propose nucleotide-dependent conformational changes that free the loops for association with sigma54. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1109.map.gz emd_1109.map.gz | 621.9 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1109-v30.xml emd-1109-v30.xml emd-1109.xml emd-1109.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1109.gif 1109.gif | 23.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1109 http://ftp.pdbj.org/pub/emdb/structures/EMD-1109 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1109 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1109 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1109_validation.pdf.gz emd_1109_validation.pdf.gz | 187.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1109_full_validation.pdf.gz emd_1109_full_validation.pdf.gz | 186.2 KB | 表示 | |
| XML形式データ |  emd_1109_validation.xml.gz emd_1109_validation.xml.gz | 5.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1109 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1109 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1109 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1109 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1109.map.gz / 形式: CCP4 / 大きさ: 12.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1109.map.gz / 形式: CCP4 / 大きさ: 12.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | this is a volume map of PspF in complex with sigma54 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
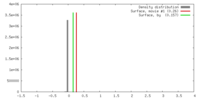
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : PspF AAA domain
| 全体 | 名称: PspF AAA domain |
|---|---|
| 要素 |
|
-超分子 #1000: PspF AAA domain
| 超分子 | 名称: PspF AAA domain / タイプ: sample / ID: 1000 集合状態: one hexamer of PspF binds to a monomer of sigma54 Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 240 KDa / 理論値: 240 KDa / 手法: mass spec |
-分子 #1: PspF AAA domain
| 分子 | 名称: PspF AAA domain / タイプ: protein_or_peptide / ID: 1 / Name.synonym: pspf AAA domain / コピー数: 6 / 集合状態: hexamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 31 KDa / 理論値: 31 KDa |
| 組換発現 | 生物種: Escherichia coli B834 / 組換プラスミド: pET28 bplus |
| 配列 | InterPro: AAA+ ATPase domain |
-分子 #2: sigma54
| 分子 | 名称: sigma54 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: sigma54 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 54 KDa / 理論値: 54 KDa |
| 組換発現 | 生物種: B834 / 組換プラスミド: pET28 bplus |
| 配列 | InterPro: RNA polymerase sigma factor 54 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 10 mM Tris HCl, pH8, 50 mM NaCl, 1 mM DTT, 0.1 mM EDTA, 5% glycerol |
| 染色 | タイプ: NEGATIVE 詳細: Grids with native sample quench frozen in liquid ethane cooled at -186 C |
| グリッド | 詳細: Holey carbon grids from Agar |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 87.15 K / Timed resolved state: vitrified 30msec after / 手法: blot for 2 seconds |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG/UT |
|---|---|
| 温度 | 平均: 103 K |
| 詳細 | weak beam illumination |
| 日付 | 2002年4月4日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - サンプリング間隔: 2 µm / 実像数: 9 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 14 |
| 電子線 | 加速電圧: 160 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: OTHER / Cs: 1.2 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.4 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: FSC 3 SIGMA CUT-OFF / ソフトウェア - 名称: IMAGIC-5 詳細: Final maps were calculate from 123 class averages from one dataset 使用した粒子像数: 3895 |
| 最終 2次元分類 | クラス数: 123 |
-原子モデル構築 1
| 詳細 | Protocol: Cross correlation coefficient between projections of fitted model and those from the EM reconstruction. The monomer was manually fitted using the program "O"; the model was p6 symmetrised and individually subunits were readjusted manually using "O" |
|---|---|
| 精密化 | プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera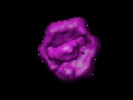







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)