+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10900 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-ET/ subtomogram averaging of primary cilia reveals actin filaments in the axoneme | |||||||||
 マップデータ マップデータ | cryo-subtomogram averaging of primary cilia actin filaments | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 45.0 Å | |||||||||
 データ登録者 データ登録者 | Kiesel P / Alvarez Viar G / Tsoy N / Maraspini R / Honigmann A / Pigino G | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: The molecular structure of mammalian primary cilia revealed by cryo-electron tomography. 著者: Petra Kiesel / Gonzalo Alvarez Viar / Nikolai Tsoy / Riccardo Maraspini / Peter Gorilak / Vladimir Varga / Alf Honigmann / Gaia Pigino /    要旨: Primary cilia are microtubule-based organelles that are important for signaling and sensing in eukaryotic cells. Unlike the thoroughly studied motile cilia, the three-dimensional architecture and ...Primary cilia are microtubule-based organelles that are important for signaling and sensing in eukaryotic cells. Unlike the thoroughly studied motile cilia, the three-dimensional architecture and molecular composition of primary cilia are largely unexplored. Yet, studying these aspects is necessary to understand how primary cilia function in health and disease. We developed an enabling method for investigating the structure of primary cilia isolated from MDCK-II cells at molecular resolution by cryo-electron tomography. We show that the textbook '9 + 0' arrangement of microtubule doublets is only present at the primary cilium base. A few microns out, the architecture changes into an unstructured bundle of EB1-decorated microtubules and actin filaments, putting an end to a long debate on the presence or absence of actin filaments in primary cilia. Our work provides a plethora of insights into the molecular structure of primary cilia and offers a methodological framework to study these important organelles. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: The molecular structure of primary cilia revealed by cryo-electron tomography 著者: Kiesel P / Alvarez Viar G / Tsoy N / Maraspini R / Honigmann A / Pigino G | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10900.map.gz emd_10900.map.gz | 24.2 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10900-v30.xml emd-10900-v30.xml emd-10900.xml emd-10900.xml | 10.8 KB 10.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10900.png emd_10900.png | 30.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10900 http://ftp.pdbj.org/pub/emdb/structures/EMD-10900 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10900 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10900 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10900_validation.pdf.gz emd_10900_validation.pdf.gz | 175.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10900_full_validation.pdf.gz emd_10900_full_validation.pdf.gz | 175 KB | 表示 | |
| XML形式データ |  emd_10900_validation.xml.gz emd_10900_validation.xml.gz | 4.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10900 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10900 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10900 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10900 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10900.map.gz / 形式: CCP4 / 大きさ: 26.4 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10900.map.gz / 形式: CCP4 / 大きさ: 26.4 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo-subtomogram averaging of primary cilia actin filaments | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 9.422 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
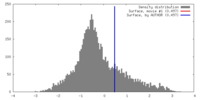
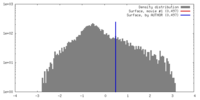
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cryo-subtomogram averaging of actin filaments in primary cilia
| 全体 | 名称: Cryo-subtomogram averaging of actin filaments in primary cilia |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-subtomogram averaging of actin filaments in primary cilia
| 超分子 | 名称: Cryo-subtomogram averaging of actin filaments in primary cilia タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.25 |
|---|---|
| グリッド | モデル: Quantifoil R3.5/1 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 99 % / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 1.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)