+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10890 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-electron density map of the P140-P110 heterodimer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | P110/P140 Nap Mycoplasma genitalium surface adhesin / CELL ADHESION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Mycoplasma genitalium G37 (バクテリア) / Mycoplasma genitalium G37 (バクテリア) /  Mycoplasma genitalium (strain ATCC 33530 / G-37 / NCTC 10195) (バクテリア) Mycoplasma genitalium (strain ATCC 33530 / G-37 / NCTC 10195) (バクテリア) | |||||||||
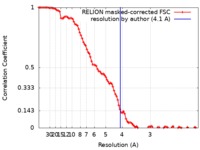
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Scheffer MP / Aparicio D | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structure and mechanism of the Nap adhesion complex from the human pathogen Mycoplasma genitalium. 著者: David Aparicio / Margot P Scheffer / Marina Marcos-Silva / David Vizarraga / Lasse Sprankel / Mercè Ratera / Miriam S Weber / Anja Seybert / Sergi Torres-Puig / Luis Gonzalez-Gonzalez / ...著者: David Aparicio / Margot P Scheffer / Marina Marcos-Silva / David Vizarraga / Lasse Sprankel / Mercè Ratera / Miriam S Weber / Anja Seybert / Sergi Torres-Puig / Luis Gonzalez-Gonzalez / Julian Reitz / Enrique Querol / Jaume Piñol / Oscar Q Pich / Ignacio Fita / Achilleas S Frangakis /   要旨: Mycoplasma genitalium is a human pathogen adhering to host target epithelial cells and causing urethritis, cervicitis and pelvic inflammatory disease. Essential for infectivity is a transmembrane ...Mycoplasma genitalium is a human pathogen adhering to host target epithelial cells and causing urethritis, cervicitis and pelvic inflammatory disease. Essential for infectivity is a transmembrane adhesion complex called Nap comprising proteins P110 and P140. Here we report the crystal structure of P140 both alone and in complex with the N-terminal domain of P110. By cryo-electron microscopy (cryo-EM) and tomography (cryo-ET) we find closed and open Nap conformations, determined at 9.8 and 15 Å, respectively. Both crystal structures and the cryo-EM structure are found in a closed conformation, where the sialic acid binding site in P110 is occluded. By contrast, the cryo-ET structure shows an open conformation, where the binding site is accessible. Structural information, in combination with functional studies, suggests a mechanism for attachment and release of M. genitalium to and from the host cell receptor, in which Nap conformations alternate to sustain motility and guarantee infectivity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10890.map.gz emd_10890.map.gz | 47.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10890-v30.xml emd-10890-v30.xml emd-10890.xml emd-10890.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10890_fsc.xml emd_10890_fsc.xml | 9.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10890.png emd_10890.png | 193.7 KB | ||
| Filedesc metadata |  emd-10890.cif.gz emd-10890.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10890 http://ftp.pdbj.org/pub/emdb/structures/EMD-10890 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10890 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10890 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10890_validation.pdf.gz emd_10890_validation.pdf.gz | 237 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10890_full_validation.pdf.gz emd_10890_full_validation.pdf.gz | 236.1 KB | 表示 | |
| XML形式データ |  emd_10890_validation.xml.gz emd_10890_validation.xml.gz | 11 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10890 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10890 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10890 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10890 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10890.map.gz / 形式: CCP4 / 大きさ: 75.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10890.map.gz / 形式: CCP4 / 大きさ: 75.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : P140-P110 heterodimer
| 全体 | 名称: P140-P110 heterodimer |
|---|---|
| 要素 |
|
-超分子 #1: P140-P110 heterodimer
| 超分子 | 名称: P140-P110 heterodimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Mycoplasma genitalium G37 (バクテリア) Mycoplasma genitalium G37 (バクテリア) |
-分子 #1: Mgp-operon protein 3
| 分子 | 名称: Mgp-operon protein 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycoplasma genitalium (strain ATCC 33530 / G-37 / NCTC 10195) (バクテリア) Mycoplasma genitalium (strain ATCC 33530 / G-37 / NCTC 10195) (バクテリア) |
| 分子量 | 理論値: 84.816969 KDa |
| 組換発現 | 生物種:  Mycoplasma genitalium G37 (バクテリア) Mycoplasma genitalium G37 (バクテリア) |
| 配列 | 文字列: TFLVKEDSKN VTAYTPFATP ITDSKSDLVS LAQLDSSYQI ADQTIHNTNL FVLFKSRDVK VKYESSGSNN ISFDSTSQGE KPSYVVEFT NSTGIKWTMV KKYQLDVPNV SSDMNQVLKN LILEQPLTKY TLNSSLAKEK GKTQREVHLG SGQANQWTSQ R NQHDLNNN ...文字列: TFLVKEDSKN VTAYTPFATP ITDSKSDLVS LAQLDSSYQI ADQTIHNTNL FVLFKSRDVK VKYESSGSNN ISFDSTSQGE KPSYVVEFT NSTGIKWTMV KKYQLDVPNV SSDMNQVLKN LILEQPLTKY TLNSSLAKEK GKTQREVHLG SGQANQWTSQ R NQHDLNNN PSPNASTGFK LTTGNAYRKL SESWPIYEPI DGTKQGKGKD SSGWSSTEEN EAKNDAPSVS SGTFNKYLNT KQ ALESIGI LFDDQTPRNV ITQLYYASTS KLAVTNNHIV VMGNSFLPSM WYWVVERSAQ ENASNKPTWF ANTNLDWGED KQK QFVENQ LGYKETTSTN SHNFHSKSFT QPAYLISGID SVNDQIIFSG FKAGSVGYDS SSSSSSSTKD QALAWSTTTS LDSK TGYKD LVTNDTGLNG PINGSFSIQD TFSFVVPYSG NHTNNGTTGP IKTAYPVKKD QKSTVKINSL INATPLNSYG DEGIG VFDA LGLNYNFKSN QERLPSRTDQ IFVYGIVSPN ELRSAKSSAD STGSDTKVNW SNTQSRYLPV PYNYSEGIID ADGFKR PEN RGASVTTFSG LKSIAPDGFA NSIANFSVGL KAGIDPNPVM SGKKANYGAV VLTRGGVVRL NFNPGNDSLL STTDNNI AP ISFSFTPFTA AESAVDLTTF KEVTYNQESG LWSYIFDSSL KPSHDGKQTP VTDNMGFSVI TVSRTGIELN QDQATTTL D VAPSALAVQS GIQSTTQTLT GVLPLSEEFS AVIAKDSDQN KIDIYKNNNG LFEIDT UniProtKB: Mgp-operon protein 3 |
-分子 #2: Adhesin P1
| 分子 | 名称: Adhesin P1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycoplasma genitalium (strain ATCC 33530 / G-37 / NCTC 10195) (バクテリア) Mycoplasma genitalium (strain ATCC 33530 / G-37 / NCTC 10195) (バクテリア) |
| 分子量 | 理論値: 128.447648 KDa |
| 組換発現 | 生物種:  Mycoplasma genitalium G37 (バクテリア) Mycoplasma genitalium G37 (バクテリア) |
| 配列 | 文字列: QAVDETLTPW TWNNNNFSSL KITGENPGSF GLVRSQNDNL NISSVTKDNL KYLNAVEKYL DGQQNFAIRR YDNNGRALYD INLAKMENP STVQRGLNGE PIFDPFKGFG LTGNAPTDWN EIKGKVPVEV VQSPHSPNLY FVLLVPKVAL EYHNLNNQVV K ESLEVSSF ...文字列: QAVDETLTPW TWNNNNFSSL KITGENPGSF GLVRSQNDNL NISSVTKDNL KYLNAVEKYL DGQQNFAIRR YDNNGRALYD INLAKMENP STVQRGLNGE PIFDPFKGFG LTGNAPTDWN EIKGKVPVEV VQSPHSPNLY FVLLVPKVAL EYHNLNNQVV K ESLEVSSF NPTQRLQKDS PVKDSSKQGE KLSETTASSM SSGMATSTRA KALKVEVERG SQSDSLLKND FAKKPLKHKN SS GEVKLEA EKEFTEAWKP LLTTDQIARE KGMGATVVSF YDAPYSENHT AFGLVDHIDP KKMVENYPPS WKTPKWNHHG IWD YNARNL LLQTTGFFNP RRHPEWFDEG QAKADNTSPG FKVGDTDHKK DGFKKNSSSP IALPFEAYFA NIGNMVAIGN SVFI FGGNG HATKMFTTNP LSIGVFRIKY TDNFSKSSVT GWPYAVLFGG LINPQTNGLK DLPLGTNRWF EYVPRMAVSG VKWVG NQLV LAGTLTMGDT ATVPRLKYDQ LEKHLNLVAQ GQGLLREDLQ IFTPYGWANR PDIPVGAWLQ DEMGSKFGPH YFLNNP DIQ DNVNNDTVEA LISSYKNTDK LKHVYPYRYS GLYAWQLFNW SNKLTNTPLS ANFVNENSYA PNSLFAAILN EDLLTGL SD KIFYGKENEF AENEADRFNQ LLSLNPNPNT NWARYLNVVQ RFTTGPNLDS STFDQFLDFL PWIGNGKPFS NSPSASTP L PTFSNINVGV KSMITQHLNK ENTRWVFIPN FSPDIWTGAG YRVQSANQKN GIPFEQVKPS NNSTPFDPNS DDNKVTPSG GSSKPTTYPA LPNSISPTSD WINALTFTNK NNPQRNQLLL RSLLGTIPVL INKSGDSNDQ FNKDSEQKWD KTETNEGNLP GFGEVNGLY NAALLHTYGF FGTNTNSTDP KIGFKADSSS SSSSTLVGSG LNWTSQDVGN LVVINDTSFG FQLGGWFITF T DFIRPRTG YLGITLSSLQ DQTIIWADQP WTSFKGSYLD SDGTPKSLWD PTALKSLPNN PTLSPSFQLY QPNKVKAYQT TN TYNKLIE PVDATSAATN MTSLLKLLTT KNIKAKLGKG TASNNGGGVS QTINTITTTG NISEGLKEET SIQAETLKKF FDS KQNNKS EIGIGDSTFT KMDGKLTGVV STPLVNLING QGAT UniProtKB: Adhesin P1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 7.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT | ||||||
| 得られたモデル |  PDB-6yrk: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)