+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10816 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Focused refinement cryo-EM structure of the yeast mitochondrial complex I sub-stoichiometric sulfur transferase subunit | |||||||||
 マップデータ マップデータ | Focused refinement of sub-stoichiometric sulfur transferase | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NADH:Ubiquinone Oxidoreductase / sulfur transferase / sub-stoichiometric / complex I / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報3-mercaptopyruvate sulfurtransferase activity / tRNA wobble position uridine thiolation / thiosulfate-cyanide sulfurtransferase activity / mitochondrion 類似検索 - 分子機能 | |||||||||
| 生物種 |  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) | |||||||||
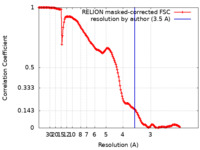
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Hirst J / Grba D | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: Mitochondrial complex I structure reveals ordered water molecules for catalysis and proton translocation. 著者: Daniel N Grba / Judy Hirst /  要旨: Mitochondrial complex I powers ATP synthesis by oxidative phosphorylation, exploiting the energy from ubiquinone reduction by NADH to drive protons across the energy-transducing inner membrane. ...Mitochondrial complex I powers ATP synthesis by oxidative phosphorylation, exploiting the energy from ubiquinone reduction by NADH to drive protons across the energy-transducing inner membrane. Recent cryo-EM analyses of mammalian and yeast complex I have revolutionized structural and mechanistic knowledge and defined structures in different functional states. Here, we describe a 2.7-Å-resolution structure of the 42-subunit complex I from the yeast Yarrowia lipolytica containing 275 structured water molecules. We identify a proton-relay pathway for ubiquinone reduction and water molecules that connect mechanistically crucial elements and constitute proton-translocation pathways through the membrane. By comparison with known structures, we deconvolute structural changes governing the mammalian 'deactive transition' (relevant to ischemia-reperfusion injury) and their effects on the ubiquinone-binding site and a connected cavity in ND1. Our structure thus provides important insights into catalysis by this enigmatic respiratory machine. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10816.map.gz emd_10816.map.gz | 323.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10816-v30.xml emd-10816-v30.xml emd-10816.xml emd-10816.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10816_fsc.xml emd_10816_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10816.png emd_10816.png | 78.6 KB | ||
| マスクデータ |  emd_10816_msk_1.map emd_10816_msk_1.map | 347.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10816.cif.gz emd-10816.cif.gz | 6.4 KB | ||
| その他 |  emd_10816_half_map_1.map.gz emd_10816_half_map_1.map.gz emd_10816_half_map_2.map.gz emd_10816_half_map_2.map.gz | 276.5 MB 276.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10816 http://ftp.pdbj.org/pub/emdb/structures/EMD-10816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10816 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10816_validation.pdf.gz emd_10816_validation.pdf.gz | 942.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10816_full_validation.pdf.gz emd_10816_full_validation.pdf.gz | 941.8 KB | 表示 | |
| XML形式データ |  emd_10816_validation.xml.gz emd_10816_validation.xml.gz | 23.4 KB | 表示 | |
| CIF形式データ |  emd_10816_validation.cif.gz emd_10816_validation.cif.gz | 30.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10816 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10816 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10816 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10816 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6yj5MC  6skkC  6skmC  6sknC  6slqC  6sluC  6smuC  6y9vC  6y9wC  6y9xC  6y9yC  6y9zC  6zdjC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10816.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10816.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused refinement of sub-stoichiometric sulfur transferase | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.055 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10816_msk_1.map emd_10816_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_10816_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_10816_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ST1 (sulfur transferase)
| 全体 | 名称: ST1 (sulfur transferase) |
|---|---|
| 要素 |
|
-超分子 #1: ST1 (sulfur transferase)
| 超分子 | 名称: ST1 (sulfur transferase) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
-分子 #1: Rhodanese-like domain-containing protein
| 分子 | 名称: Rhodanese-like domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
| 分子量 | 理論値: 34.661117 KDa |
| 組換発現 | 生物種:  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) |
| 配列 | 文字列: MSKLISPAEL AKRLSSKETK IFDATWYLPT PANVGKNAYD NYMKKRIPGA LYFDIDAVNT PSKFPHMLPS PQTFENELTK LGVSSDSPI VVYDTQGVFS GPRLVWTFKV FGHDNVQFLN GFEAYTQLPG IPSRPDAYTW GIWDTQVPGK IDPADPPYKV T KARPELVK ...文字列: MSKLISPAEL AKRLSSKETK IFDATWYLPT PANVGKNAYD NYMKKRIPGA LYFDIDAVNT PSKFPHMLPS PQTFENELTK LGVSSDSPI VVYDTQGVFS GPRLVWTFKV FGHDNVQFLN GFEAYTQLPG IPSRPDAYTW GIWDTQVPGK IDPADPPYKV T KARPELVK SFEDVLAIVE KHNGDGAKIR NEVTFIDARP NGRFTGKDAE PRAELSSGHV PGAYSIAFPE VVENGKFKSP EE LKALFAS KGIDGSKPII SMCGSGVTAC VIDLALEIAG IGSRDTNAVY DGSWTEWAQR APTKYIVKEE NLNEANRA UniProtKB: Rhodanese-like domain-containing protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.45 |
|---|---|
| グリッド | モデル: UltrAuFoil / 材質: GOLD / 支持フィルム - 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 詳細: Gold grids saturated with PEG thiol reagent |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2540 / 平均電子線量: 44.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): -2.7 µm / 最小 デフォーカス(補正後): -1.5 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)