+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10357 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bat Influenza A polymerase product dissociation complex using 44-mer vRNA template with mutated oligo(U) sequence | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Influenza / polymerase / viral transcription / RNA / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cap snatching / viral transcription / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / host cell mitochondrion / 7-methylguanosine mRNA capping / virion component / host cell cytoplasm / RNA-directed RNA polymerase / viral translational frameshifting / viral RNA genome replication ...cap snatching / viral transcription / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / host cell mitochondrion / 7-methylguanosine mRNA capping / virion component / host cell cytoplasm / RNA-directed RNA polymerase / viral translational frameshifting / viral RNA genome replication / RNA-dependent RNA polymerase activity / nucleotide binding / DNA-templated transcription / host cell nucleus / RNA binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス) / synthetic construct (人工物) Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス) / synthetic construct (人工物) | |||||||||
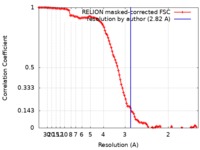
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.82 Å | |||||||||
 データ登録者 データ登録者 | Wandzik JM / Kouba T | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2020 ジャーナル: Cell / 年: 2020タイトル: A Structure-Based Model for the Complete Transcription Cycle of Influenza Polymerase. 著者: Joanna M Wandzik / Tomas Kouba / Manikandan Karuppasamy / Alexander Pflug / Petra Drncova / Jan Provaznik / Nayara Azevedo / Stephen Cusack /   要旨: Influenza polymerase uses unique mechanisms to synthesize capped and polyadenylated mRNAs from the genomic viral RNA (vRNA) template, which is packaged inside ribonucleoprotein particles (vRNPs). ...Influenza polymerase uses unique mechanisms to synthesize capped and polyadenylated mRNAs from the genomic viral RNA (vRNA) template, which is packaged inside ribonucleoprotein particles (vRNPs). Here, we visualize by cryoelectron microscopy the conformational dynamics of the polymerase during the complete transcription cycle from pre-initiation to termination, focusing on the template trajectory. After exiting the active site cavity, the template 3' extremity rebinds into a specific site on the polymerase surface. Here, it remains sequestered during all subsequent transcription steps, forcing the template to loop out as it further translocates. At termination, the strained connection between the bound template 5' end and the active site results in polyadenylation by stuttering at uridine 17. Upon product dissociation, further conformational changes release the trapped template, allowing recycling back into the pre-initiation state. Influenza polymerase thus performs transcription while tightly binding to and protecting both template ends, allowing efficient production of multiple mRNAs from a single vRNP. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10357.map.gz emd_10357.map.gz | 108.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10357-v30.xml emd-10357-v30.xml emd-10357.xml emd-10357.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10357_fsc.xml emd_10357_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10357.png emd_10357.png | 139.9 KB | ||
| Filedesc metadata |  emd-10357.cif.gz emd-10357.cif.gz | 7.1 KB | ||
| その他 |  emd_10357_additional.map.gz emd_10357_additional.map.gz emd_10357_half_map_1.map.gz emd_10357_half_map_1.map.gz emd_10357_half_map_2.map.gz emd_10357_half_map_2.map.gz | 55.7 MB 91.3 MB 91.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10357 http://ftp.pdbj.org/pub/emdb/structures/EMD-10357 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10357 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10357 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10357_validation.pdf.gz emd_10357_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10357_full_validation.pdf.gz emd_10357_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_10357_validation.xml.gz emd_10357_validation.xml.gz | 18.2 KB | 表示 | |
| CIF形式データ |  emd_10357_validation.cif.gz emd_10357_validation.cif.gz | 23.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10357 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10357 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10357 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10357 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6t0rMC  6szuC  6szvC  6t0nC  6t0sC  6t0uC  6t0vC  6t0wC  6t2cC  6tu5C  6tw1C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10357.map.gz / 形式: CCP4 / 大きさ: 115.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10357.map.gz / 形式: CCP4 / 大きさ: 115.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8127 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: LocScale map
| ファイル | emd_10357_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LocScale map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10357_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10357_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bat Influenza A polymerase product dissociation complex using 44-...
| 全体 | 名称: Bat Influenza A polymerase product dissociation complex using 44-mer vRNA template with mutated oligo(U) sequence |
|---|---|
| 要素 |
|
-超分子 #1: Bat Influenza A polymerase product dissociation complex using 44-...
| 超分子 | 名称: Bat Influenza A polymerase product dissociation complex using 44-mer vRNA template with mutated oligo(U) sequence タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: Polymerase
| 超分子 | 名称: Polymerase / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス) Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス) |
-超分子 #3: Nucleic acid
| 超分子 | 名称: Nucleic acid / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Polymerase acidic protein
| 分子 | 名称: Polymerase acidic protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス) Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス)株: A/little yellow-shouldered bat/Guatemala/060/2010(H17N10) |
| 分子量 | 理論値: 85.49093 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GSHHHHHHHH GSGSMENFVR TNFNPMILER AEKTMKEYGE NPQNEGNKFA AISTHMEVCF MYSDFHFIDL EGNTIVKEND DDNAMLKHR FEIIEGQERN IAWTIVNSIC NMTENSKPRF LPDLYDYKTN KFIEIGVTRR KVEDYYYEKA SKLKGENVYI H IFSFDGEE ...文字列: GSHHHHHHHH GSGSMENFVR TNFNPMILER AEKTMKEYGE NPQNEGNKFA AISTHMEVCF MYSDFHFIDL EGNTIVKEND DDNAMLKHR FEIIEGQERN IAWTIVNSIC NMTENSKPRF LPDLYDYKTN KFIEIGVTRR KVEDYYYEKA SKLKGENVYI H IFSFDGEE MATDDEYILD EESRARIKTR LFVLRQELAT AGLWDSFRQS EKGEETLEEE FSYPPTFQRL ANQSLPPSFK DY HQFKAYV SSFKANGNIE AKLGAMSEKV NAQIESFDPR TIRELELPEG KFCTQRSKFL LMDAMKLSVL NPAHEGEGIP MKD AKACLD TFWGWKKATI IKKHEKGVNT NYLMIWEQLL ESIKEMEGKF LNLKKTNHLK WGLGEGQAPE KMDFEDCKEV PDLF QYKSE PPEKRKLASW IQSEFNKASE LTNSNWIEFD ELGNDVAPIE HIASRRRNFF TAEVSQCRAS EYIMKAVYIN TALLN SSCT AMEEYQVIPI ITKCRDTSGQ RRTNLYGFII KGRSHLRNDT DVVNFISLEF SLTDPRNEIH KWEKYCVLEI GDMEIR TSI STIMKPVYLY VRTNGTSKIK MKWGMEMRRC LLQSLQQVES MIEAESAVKE KDMTEPFFRN RENDWPIGES PQGIEKG TI GKVCRVLLAK SVFNSIYASA QLEGFSAESR KLLLLIQAFR DNLDPGTFDL KGLYEAIEEC IINDPWVLLN ASWFNSFL K AVQLSMGSGS GENLYFQ UniProtKB: Polymerase acidic protein |
-分子 #2: RNA-directed RNA polymerase catalytic subunit
| 分子 | 名称: RNA-directed RNA polymerase catalytic subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス) Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス)株: A/little yellow-shouldered bat/Guatemala/060/2010(H17N10) |
| 分子量 | 理論値: 87.936312 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GSGSGSGSGM DVNPMLIFLK VPVQNAISTT FPYTGDPPYS HGTGTGYTMD TVIRTHDYSS RGIWKTNSET GAQQLNPIDG PLPEDNEPS GYAQTDCVLE LIEGLDRSHP GLFETACQET IDAIQQTRVD KLTQGRQTYD WTLNRNQPAA TALANTIEVF R KNGYKLNE ...文字列: GSGSGSGSGM DVNPMLIFLK VPVQNAISTT FPYTGDPPYS HGTGTGYTMD TVIRTHDYSS RGIWKTNSET GAQQLNPIDG PLPEDNEPS GYAQTDCVLE LIEGLDRSHP GLFETACQET IDAIQQTRVD KLTQGRQTYD WTLNRNQPAA TALANTIEVF R KNGYKLNE SGRLIDFLKD VLLSFENDSM EVTTHFQKKK RIRDNHSKKM ITQRTIGKKR VKLTKKNYLI RALTLNTMTK DA ERGKLKR RAIATPGMQI RGFVYFVELL ARNICERLEQ SGLPVGGNEK KAKLANVIKK MMAKSTDEEL SYTITGDNTK WNE NQNPRI FLAMVLRITA GQPEWFRDLL AVAPIMFSNK VARLGRGYMF ESKSMHLRTQ ISAENLSDIN LRYFNEDTKK KIEK IRHLM VEGTASLSPG MMMGMFNMLS TVLGVSVLNL GQREILKRTY WWDGLQSSDD FALIINGHFK EDIQQGVNHF YRTCK LVGI NMSQKKSYIN KTGTFEFTSF FYRYGFVANF SMELPSFGVA GNNESADMSI GTTVIKTNMI NNDLGPATAQ MAIQLF IKD YRYTYRCHRG DTNLETRRTK SIKRLWTETI SKAGLLVADG GPNPYNLRNL HIPEVCLKWS LMDPDYRGRL CNPNNPF VH HMEVESTNLA VVMPAHGPAK SLEYDAVATT HSWTPKRNRS ILNTNQRGIL EDERIYQKCC QVFEKFFPSS TYRRPIGM A SMLDAMLSRA RIDARIDLES GRISSQDFSE ITNTCKAIEA LKRQGSGSGE NLYFQ UniProtKB: RNA-directed RNA polymerase catalytic subunit |
-分子 #3: Polymerase basic protein 2
| 分子 | 名称: Polymerase basic protein 2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス) Influenza A virus (A/little yellow-shouldered bat/Guatemala/060/2010(H17N10)) (A型インフルエンザウイルス)株: A/little yellow-shouldered bat/Guatemala/060/2010(H17N10) |
| 分子量 | 理論値: 91.027141 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GSGSGSGSGM ERIKELMEMV KNSRMREILT TTSVDHMAVI KKYTSGRQEK NPALRMKWMM AMKYPISASS RIREMIPEKD EDGNTLWTN TKDAGSNRVL VSPNAVTWWN RAGPVSDVVH YPRVYKMYFD RLERLTHGTF GPVKFYNQVK VRKRVDINPG H KDLTSREA ...文字列: GSGSGSGSGM ERIKELMEMV KNSRMREILT TTSVDHMAVI KKYTSGRQEK NPALRMKWMM AMKYPISASS RIREMIPEKD EDGNTLWTN TKDAGSNRVL VSPNAVTWWN RAGPVSDVVH YPRVYKMYFD RLERLTHGTF GPVKFYNQVK VRKRVDINPG H KDLTSREA QEVIMEVVFP NEVGARTLSS DAQLTITKEK KEELKNCKIS PIMVAYMLER ELVRRTRFLP IAGATSSTYV EV LHLTQGT CWEQQYTPGG EAENDDLDQT LIIASRNIVR RSIVAIDPLA SLLSMCHTTS ISSEPLVEIL RSNPTDEQAV NIC KAALGI RINNSFSFGG YNFKRVKGSS QRTEKAVLTG NLQTLTMTIF EGYEEFNVSG KRASAVLKKG AQRLIQAIIG GRTL EDILN LMITLMVFSQ EEKMLKAVRG DLNFVNRANQ RLNPMYQLLR HFQKDSSTLL KNWGTEEIDP IMGIAGIMPD GTINK TQTL MGVRLSQGGV DEYSFNERIR VNIDKYLRVR NEKGELLISP EEVSEAQGQE KLPINYNSSL MWEVNGPESI LTNTYH WII KNWELLKTQW MTDPTVLYNR IEFEPFQTLI PKGNRAIYSG FTRTLFQQMR DVEGTFDSIQ IIKLLPFSAH PPSLGRT QF SSFTLNIRGA PLRLLIRGNS QVFNYNQMEN VIIVLGKSVG SPERSILTES SSIESAVLRG FLILGKANSK YGPVLTIG E LDKLGRGEKA NVLIGQGDTV LVMKRKRDSS ILTDSQTALK RIRLEESKGW SHPQFEKGGG SGGGSGGSAW SHPQFEKGR SGGENLYFQ UniProtKB: Polymerase basic protein 2 |
-分子 #4: vRNA
| 分子 | 名称: vRNA / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 14.012289 KDa |
| 配列 | 文字列: AGUAGUAACA AGAGCAAUGU GUCCGUCUCG CCUCUGCUUC UGCU |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 31 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 39.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)