+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0760 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | TRiC at 0.1 mM ADP-AlFx, Conformation 4, 0.1-C4 | |||||||||||||||
 マップデータ マップデータ | TRiC at 0.1 mM ADP-AlFx, Conformation 1, 0.1-C4 | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Chaperonin TRiC/CCT / Allosteric network / ATPase cycle / Conformational landscape / Cryo-EM / CHAPERONE | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Association of TriC/CCT with target proteins during biosynthesis / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / chaperonin-containing T-complex / : / Neutrophil degranulation / ATP-dependent protein folding chaperone / unfolded protein binding / protein folding / ATP hydrolysis activity / ATP binding ...Association of TriC/CCT with target proteins during biosynthesis / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / chaperonin-containing T-complex / : / Neutrophil degranulation / ATP-dependent protein folding chaperone / unfolded protein binding / protein folding / ATP hydrolysis activity / ATP binding / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.69 Å | |||||||||||||||
 データ登録者 データ登録者 | Jin M / Cong Y | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2019 ジャーナル: Proc Natl Acad Sci U S A / 年: 2019タイトル: An ensemble of cryo-EM structures of TRiC reveal its conformational landscape and subunit specificity. 著者: Mingliang Jin / Wenyu Han / Caixuan Liu / Yunxiang Zang / Jiawei Li / Fangfang Wang / Yanxing Wang / Yao Cong /  要旨: TRiC/CCT assists the folding of ∼10% of cytosolic proteins through an ATP-driven conformational cycle and is essential in maintaining protein homeostasis. Here, we determined an ensemble of cryo- ...TRiC/CCT assists the folding of ∼10% of cytosolic proteins through an ATP-driven conformational cycle and is essential in maintaining protein homeostasis. Here, we determined an ensemble of cryo-electron microscopy (cryo-EM) structures of yeast TRiC at various nucleotide concentrations, with 4 open-state maps resolved at near-atomic resolutions, and a closed-state map at atomic resolution, revealing an extra layer of an unforeseen N-terminal allosteric network. We found that, during TRiC ring closure, the CCT7 subunit moves first, responding to nucleotide binding; CCT4 is the last to bind ATP, serving as an ATP sensor; and CCT8 remains ADP-bound and is hardly involved in the ATPase-cycle in our experimental conditions; overall, yeast TRiC consumes nucleotide in a 2-ring positively coordinated manner. Our results depict a thorough picture of the TRiC conformational landscape and its allosteric transitions from the open to closed states in more structural detail and offer insights into TRiC subunit specificity in ATP consumption and ring closure, and potentially in substrate processing. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0760.map.gz emd_0760.map.gz | 6.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0760-v30.xml emd-0760-v30.xml emd-0760.xml emd-0760.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0760.png emd_0760.png | 171.1 KB | ||
| Filedesc metadata |  emd-0760.cif.gz emd-0760.cif.gz | 8.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0760 http://ftp.pdbj.org/pub/emdb/structures/EMD-0760 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0760 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0760 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0760_validation.pdf.gz emd_0760_validation.pdf.gz | 413.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0760_full_validation.pdf.gz emd_0760_full_validation.pdf.gz | 413.5 KB | 表示 | |
| XML形式データ |  emd_0760_validation.xml.gz emd_0760_validation.xml.gz | 6.3 KB | 表示 | |
| CIF形式データ |  emd_0760_validation.cif.gz emd_0760_validation.cif.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0760 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0760 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0760 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0760 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6ks8MC  0756C  0757C  0758C  0759C  0761C  0762C  0763C  0764C  0765C  0766C  0767C  6krdC  6kreC  6ks6C  6ks7C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0760.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0760.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | TRiC at 0.1 mM ADP-AlFx, Conformation 1, 0.1-C4 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.318 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
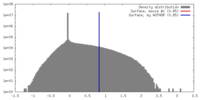
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TRiC complex
| 全体 | 名称: TRiC complex |
|---|---|
| 要素 |
|
-超分子 #1: TRiC complex
| 超分子 | 名称: TRiC complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: TRiC complex at 0.1 mM ADP-AlFx |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 960 KDa |
-分子 #1: T-complex protein 1 subunit alpha
| 分子 | 名称: T-complex protein 1 subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 60.557566 KDa |
| 配列 | 文字列: MSQLFNNSRS DTLFLGGEKI SGDDIRNQNV LATMAVANVV KSSLGPVGLD KMLVDDIGDF TVTNDGATIL SLLDVQHPAG KILVELAQQ QDREIGDGTT SVVIIASELL KRANELVKNK IHPTTIITGF RVALREAIRF INEVLSTSVD TLGKETLINI A KTSMSSKI ...文字列: MSQLFNNSRS DTLFLGGEKI SGDDIRNQNV LATMAVANVV KSSLGPVGLD KMLVDDIGDF TVTNDGATIL SLLDVQHPAG KILVELAQQ QDREIGDGTT SVVIIASELL KRANELVKNK IHPTTIITGF RVALREAIRF INEVLSTSVD TLGKETLINI A KTSMSSKI IGADSDFFSN MVVDALLAVK TQNSKGEIKY PVKAVNVLKA HGKSATESLL VPGYALNCTV ASQAMPKRIA GG NVKIACL DLNLQKARMA MGVQINIDDP EQLEQIRKRE AGIVLERVKK IIDAGAQVVL TTKGIDDLCL KEFVEAKIMG VRR CKKEDL RRIARATGAT LVSSMSNLEG EETFESSYLG LCDEVVQAKF SDDECILIKG TSKHSSSSII LRGANDYSLD EMER SLHDS LSVVKRTLES GNVVPGGGCV EAALNIYLDN FATTVGSREQ LAIAEFAAAL LIIPKTLAVN AAKDSSELVA KLRSY HAAS QMAKPEDVKR RSYRNYGLDL IRGKIVDEIH AGVLEPTISK VKSLKSALEA CVAILRIDTM ITVDPEPPKE DPHDH UniProtKB: T-complex protein 1 subunit alpha |
-分子 #2: T-complex protein 1 subunit beta
| 分子 | 名称: T-complex protein 1 subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.276254 KDa |
| 配列 | 文字列: MSVQIFGDQV TEERAENARL SAFVGAIAVG DLVKSTLGPK GMDKLLQSAS SNTCMVTNDG ATILKSIPLD NPAAKVLVNI SKVQDDEVG DGTTSVTVLS AELLREAEKL IDQSKIHPQT IIEGYRLASA AALDALTKAA VDNSHDKTMF REDLIHIAKT T LSSKILSQ ...文字列: MSVQIFGDQV TEERAENARL SAFVGAIAVG DLVKSTLGPK GMDKLLQSAS SNTCMVTNDG ATILKSIPLD NPAAKVLVNI SKVQDDEVG DGTTSVTVLS AELLREAEKL IDQSKIHPQT IIEGYRLASA AALDALTKAA VDNSHDKTMF REDLIHIAKT T LSSKILSQ DKDHFAELAT NAILRLKGST NLEHIQIIKI LGGKLSDSFL DEGFILAKKF GNNQPKRIEN AKILIANTTL DT DKVKIFG TKFKVDSTAK LAQLEKAERE KMKNKIAKIS KFGINTFINR QLIYDYPEQL FTDLGINSIE HADFEGVERL ALV TGGEVV STFDEPSKCK LGECDVIEEI MLGEQPFLKF SGCKAGEACT IVLRGATDQT LDEAERSLHD ALSVLSQTTK ETRT VLGGG CAEMVMSKAV DTEAQNIDGK KSLAVEAFAR ALRQLPTILA DNAGFDSSEL VSKLRSSIYN GISTSGLDLN NGTIA DMRQ LGIVESYKLK RAVVSSASEA AEVLLRVDNI IRARPRTANR QHM UniProtKB: T-complex protein 1 subunit beta |
-分子 #3: T-complex protein 1 subunit delta
| 分子 | 名称: T-complex protein 1 subunit delta / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.68241 KDa |
| 配列 | 文字列: MSAKVPSNAT FKNKEKPQEV RKANIIAARS VADAIRTSLG PKGMDKMIKT SRGEIIISND GHTILKQMAI LHPVARMLVE VSAAQDSEA GDGTTSVVIL TGALLGAAER LLNKGIHPTI IADSFQSAAK RSVDILLEMC HKVSLSDREQ LVRAASTSLS S KIVSQYSS ...文字列: MSAKVPSNAT FKNKEKPQEV RKANIIAARS VADAIRTSLG PKGMDKMIKT SRGEIIISND GHTILKQMAI LHPVARMLVE VSAAQDSEA GDGTTSVVIL TGALLGAAER LLNKGIHPTI IADSFQSAAK RSVDILLEMC HKVSLSDREQ LVRAASTSLS S KIVSQYSS FLAPLAVDSV LKISDENSKN VDLNDIRLVK KVGGTIDDTE MIDGVVLTQT AIKSAGGPTR KEKAKIGLIQ FQ ISPPKPD TENNIIVNDY RQMDKILKEE RAYLLNICKK IKKAKCNVLL IQKSILRDAV NDLALHFLSK LNIMVVKDIE REE IEFLSK GLGCKPIADI ELFTEDRLGS ADLVEEIDSD GSKIVRVTGI RNNNARPTVS VVIRGANNMI IDETERSLHD ALCV IRCLV KERGLIAGGG APEIEISRRL SKEARSMEGV QAFIWQEFAS ALEVIPTTLA ENAGLNSIKV VTELRSKHEN GELND GISV RRSGTTNTYE EHILQPVLVS TSAITLASEC VKSILRIDDI AFSR UniProtKB: T-complex protein 1 subunit delta |
-分子 #4: T-complex protein 1 subunit epsilon
| 分子 | 名称: T-complex protein 1 subunit epsilon / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 61.995004 KDa |
| 配列 | 文字列: MAARPQQPPM EMPDLSNAIV AQDEMGRPFI IVKDQGNKKR QHGLEAKKSH ILAARSVASI IKTSLGPRGL DKILISPDGE ITITNDGAT ILSQMELDNE IAKLLVQLSK SQDDEIGDGT TGVVVLASAL LDQALELIQK GIHPIKIANG FDEAAKLAIS K LEETCDDI ...文字列: MAARPQQPPM EMPDLSNAIV AQDEMGRPFI IVKDQGNKKR QHGLEAKKSH ILAARSVASI IKTSLGPRGL DKILISPDGE ITITNDGAT ILSQMELDNE IAKLLVQLSK SQDDEIGDGT TGVVVLASAL LDQALELIQK GIHPIKIANG FDEAAKLAIS K LEETCDDI SASNDELFRD FLLRAAKTSL GSKIVSKDHD RFAEMAVEAV INVMDKDRKD VDFDLIKMQG RVGGSISDSK LI NGVILDK DFSHPQMPKC VLPKEGSDGV KLAILTCPFE PPKPKTKHKL DISSVEEYQK LQTYEQDKFK EMIDDVKKAG ADV VICQWG FDDEANHLLL QNDLPAVRWV GGQELEHIAI STNGRIVPRF QDLSKDKLGT CSRIYEQEFG TTKDRMLIIE QSKE TKTVT CFVRGSNKMI VDEAERALHD SLCVVRNLVK DSRVVYGGGA AEVTMSLAVS EEADKQRGID QYAFRGFAQA LDTIP MTLA ENSGLDPIGT LSTLKSKQLK EKISNIGVDC LGYGSNDMKE LFVVDPFIGK KQQILLATQL CRMILKIDNV IISGKD EY UniProtKB: T-complex protein 1 subunit epsilon |
-分子 #5: fusion protein of T-complex protein 1 subunit gamma, rep-His-CBP ...
| 分子 | 名称: fusion protein of T-complex protein 1 subunit gamma, rep-His-CBP and T-complex protein 1 subunit gamma タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 65.423387 KDa |
| 配列 | 文字列: MQAPVVFMNA SQERTTGRQA QISNITAAKA VADVIRTCLG PKAMLKMLLD PMGGLVLTND GHAILREIDV AHPAAKSMLE LSRTQDEEV GDGTTTVIIL AGEILAQCAP YLIEKNIHPV IIIQALKKAL TDALEVIKQV SKPVDVENDA AMKKLIQASI G TKYVIHWS ...文字列: MQAPVVFMNA SQERTTGRQA QISNITAAKA VADVIRTCLG PKAMLKMLLD PMGGLVLTND GHAILREIDV AHPAAKSMLE LSRTQDEEV GDGTTTVIIL AGEILAQCAP YLIEKNIHPV IIIQALKKAL TDALEVIKQV SKPVDVENDA AMKKLIQASI G TKYVIHWS EKMCELALDA VKTVRKDLGQ TVEGEPNFEI DIKRYVRVEK IPGGDVLDSR VLKGVLLNKD VVHPKMSRHI EN PRVVLLD CPLEYKKGES QTNIEIEKEE DWNRILQIEE EQVQLMCEQI LAVRPTLVIT EKGVSDLAQH YLLKGGCSVL RRV KKSDNN RIARVTGATI VNRVEDLKES DVGTNCGLFK VEMIGDEYFS FLDNCKEPLE GSGSGWSHPQ FEKGSGKRRW KKNF IAVSA ANRFKKISSS GALGSGHHHH HHHHGSGLQK ACTIMLRGGS KDILNEIDRN LQDAMAVARN VMLSPSLSPG GGATE MAVS VKLAEKAKQL EGIQQWPYQA VADAMECIPR TLIQNAGGDP IRLLSQLRAK HAQGNFTTGI DGDKGKIVDM VSYGIW EPE VIKQQSVKTA IESACLLLRV DDIVSGVRKQ E UniProtKB: T-complex protein 1 subunit gamma, T-complex protein 1 subunit gamma |
-分子 #6: T-complex protein 1 subunit eta
| 分子 | 名称: T-complex protein 1 subunit eta / タイプ: protein_or_peptide / ID: 6 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 59.802438 KDa |
| 配列 | 文字列: MNFGSQTPTI VVLKEGTDAS QGKGQIISNI NACVAVQEAL KPTLGPLGSD ILIVTSNQKT TISNDGATIL KLLDVVHPAA KTLVDISRA QDAEVGDGTT SVTILAGELM KEAKPFLEEG ISSHLIMKGY RKAVSLAVEK INELAVDITS EKSSGRELLE R CARTAMSS ...文字列: MNFGSQTPTI VVLKEGTDAS QGKGQIISNI NACVAVQEAL KPTLGPLGSD ILIVTSNQKT TISNDGATIL KLLDVVHPAA KTLVDISRA QDAEVGDGTT SVTILAGELM KEAKPFLEEG ISSHLIMKGY RKAVSLAVEK INELAVDITS EKSSGRELLE R CARTAMSS KLIHNNADFF VKMCVDAVLS LDRNDLDDKL IGIKKIPGGA MEESLFINGV AFKKTFSYAG FEQQPKKFNN PK ILSLNVE LELKAEKDNA EVRVEHVEDY QAIVDAEWQL IFEKLRQVEE TGANIVLSKL PIGDLATQFF ADRNIFCAGR VSA DDMNRV IQAVGGSIQS TTSDIKPEHL GTCALFEEMQ IGSERYNLFQ GCPQAKTCTL LLRGGAEQVI AEVERSLHDA IMIV KRALQ NKLIVAGGGA TEMEVSKCLR DYSKTIAGKQ QMIINAFAKA LEVIPRQLCE NAGFDAIEIL NKLRLAHSKG EKWYG VVFE TENIGDNFAK FVWEPALVKI NALNSATEAT NLILSVDETI TNKGSESANA GMMPPQGAGR GRGMPM UniProtKB: T-complex protein 1 subunit eta |
-分子 #7: T-complex protein 1 subunit theta
| 分子 | 名称: T-complex protein 1 subunit theta / タイプ: protein_or_peptide / ID: 7 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 61.735102 KDa |
| 配列 | 文字列: MSLRLPQNPN AGLFKQGYNS YSNADGQIIK SIAAIRELHQ MCLTSMGPCG RNKIIVNHLG KIIITNDAAT MLRELDIVHP AVKVLVMAT EQQKIDMGDG TNLVMILAGE LLNVSEKLIS MGLSAVEIIQ GYNMARKFTL KELDEMVVGE ITDKNDKNEL L KMIKPVIS ...文字列: MSLRLPQNPN AGLFKQGYNS YSNADGQIIK SIAAIRELHQ MCLTSMGPCG RNKIIVNHLG KIIITNDAAT MLRELDIVHP AVKVLVMAT EQQKIDMGDG TNLVMILAGE LLNVSEKLIS MGLSAVEIIQ GYNMARKFTL KELDEMVVGE ITDKNDKNEL L KMIKPVIS SKKYGSEDIL SELVSEAVSH VLPVAQQAGE IPYFNVDSIR VVKIMGGSLS NSTVIKGMVF NREPEGHVKS LS EDKKHKV AVFTCPLDIA NTETKGTVLL HNAQEMLDFS KGEEKQIDAM MKEIADMGVE CIVAGAGVGE LALHYLNRYG ILV LKVPSK FELRRLCRVC GATPLPRLGA PTPEELGLVE TVKTMEIGGD RVTVFKQEQG EISRTSTIIL RGATQNNLDD IERA IDDGV AAVKGLMKPS GGKLLPGAGA TEIELISRIT KYGERTPGLL QLAIKQFAVA FEVVPRTLAE TAGLDVNEVL PNLYA AHNV TEPGAVKTDH LYKGVDIDGE SDEGVKDIRE ENIYDMLATK KFAINVATEA ATTVLSIDQI IMAKKAGGPR APQGPR PGN WDQED UniProtKB: T-complex protein 1 subunit theta |
-分子 #8: T-complex protein 1 subunit zeta
| 分子 | 名称: T-complex protein 1 subunit zeta / タイプ: protein_or_peptide / ID: 8 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 59.997559 KDa |
| 配列 | 文字列: MSLQLLNPKA ESLRRDAALK VNVTSAEGLQ SVLETNLGPK GTLKMLVDGA GNIKLTKDGK VLLTEMQIQS PTAVLIARAA AAQDEITGD GTTTVVCLVG ELLRQAHRFI QEGVHPRIIT DGFEIARKES MKFLDEFKIS KTNLSNDREF LLQVARSSLL T KVDADLTE ...文字列: MSLQLLNPKA ESLRRDAALK VNVTSAEGLQ SVLETNLGPK GTLKMLVDGA GNIKLTKDGK VLLTEMQIQS PTAVLIARAA AAQDEITGD GTTTVVCLVG ELLRQAHRFI QEGVHPRIIT DGFEIARKES MKFLDEFKIS KTNLSNDREF LLQVARSSLL T KVDADLTE VLTPIVTDAV LSVYDAQADN LDLHMVEIMQ MQHLSPKDTT FIKGLVLDHG GRHPDMPTRV KNAYVLILNV SL EYEKTEV NSGFFYSSAD QRDKLAASER KFVDAKLKKI IDLKNEVCGM DPDKGFVIIN QKGIDPMSLD VFAKHNILAL RRA KRRNME RLQLVTGGEA QNSVEDLSPQ ILGFSGLVYQ ETIGEEKFTY VTENTDPKSC TILIKGSTHY ALAQTKDAVR DGLR AVANV LKDKNIIPGA GAFYIALSRY LRSANMNKLG AKGKTKTGIE AFAEALLVIP KTLVKNSGFD PLDVLAMVED ELDDA QDSD ETRYVGVDLN IGDSCDPTIE GIWDSYRVLR NAITGATGIA SNLLLCDELL RAGRSTLKET PQ UniProtKB: T-complex protein 1 subunit zeta |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 38.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)